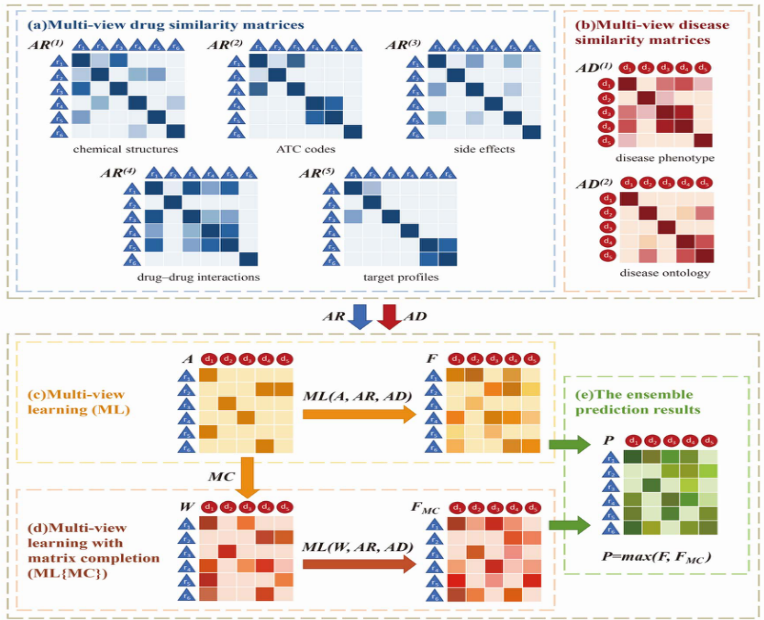
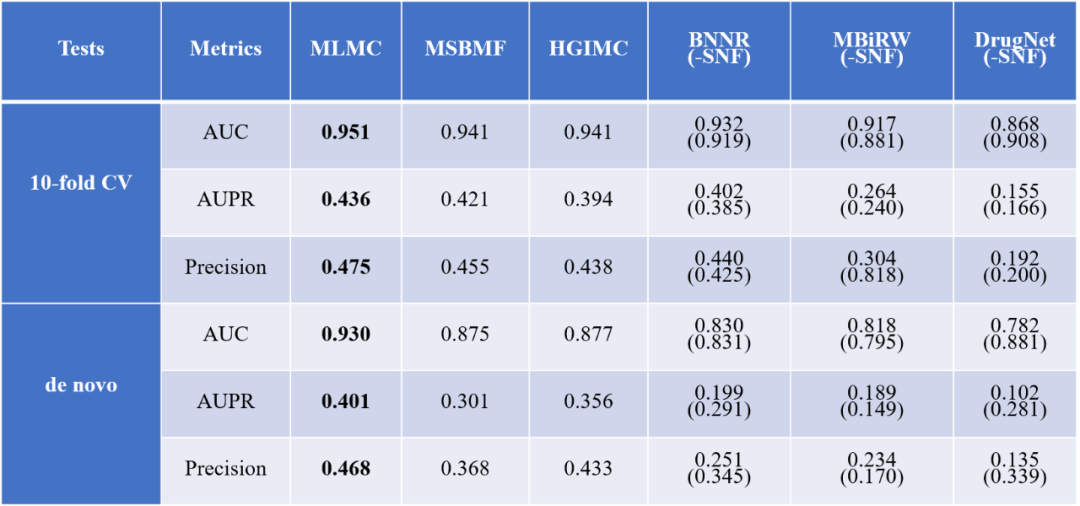
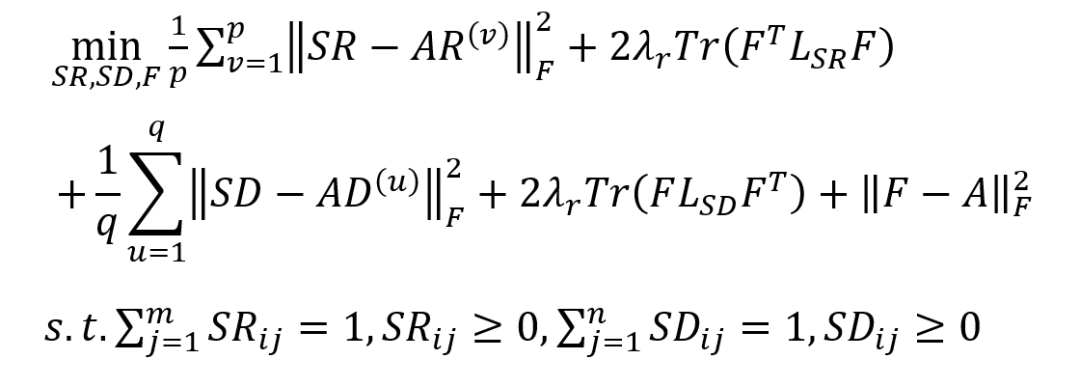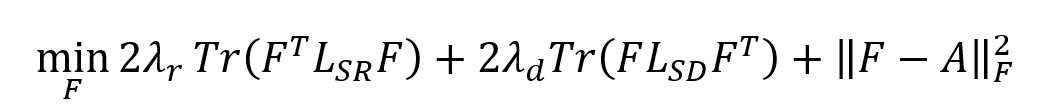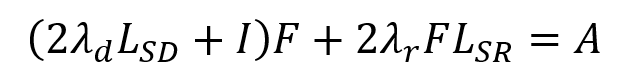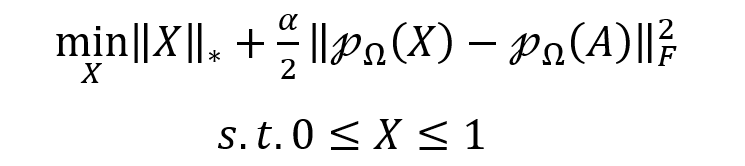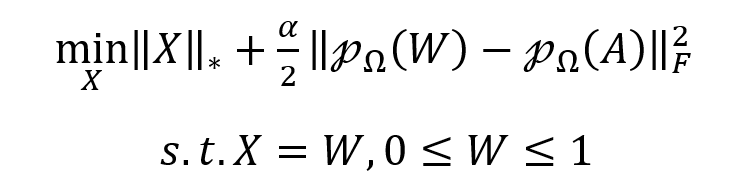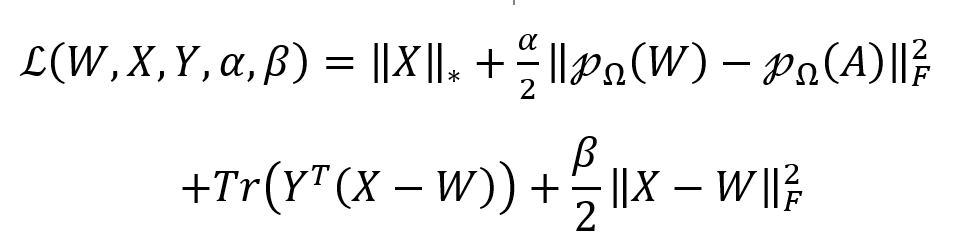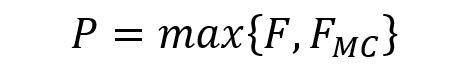药物适应症的确定是药物开发过程的关键部分。药物重新定位旨在为现有药物寻找潜在的适应症,以替代昂贵和耗时的传统药物发现过程。因此作者提出了一种基于矩阵补全的多视图学习(MLMC)方法来预测药物和疾病之间的潜在关联。MLMC的整体工作流程如图1所示。计算药物定位的机器学习方法通常将关联预测视为使用药物和疾病先验信息的二元分类问题,MLMC可以在很大程度上避免机器学习对特征提取和负样本选择的依赖问题。通过10倍交叉验证和de novo测试评估,MLMC比当前最先进的方法更高的预测精度。案例研究表明MLMC可以有效的帮助发现新的药物-疾病关联。
图1 MLMC的整体工作流程********[1]************数据处理
从DrugBank数据库和Online Mendelian Inheritance in Man (OMIM)数据库上获取已确认的1933种已知药物-疾病关联,包含593种药物和313种疾病。根据药物的化学结构、解剖学治疗学及化学分类系统(ATC)代码、副作用、药物-药物相互作用和靶点谱计算了五种药物相似性,即化学结构相似性AR(1)、ATC代码相似性AR(2)、副作用相似性AR(3)、药物-药物相互作用相似性AR(4)和靶标相似度AR(5)。根据疾病表型和本体论的计算了疾病表型相似性AD(1)和Disease Ontology(DO)相似度AD(2)两种疾病相似性。方法从不同来源收集五种药物相似性和两种疾病相似性数据后,首先采用拉普拉斯图正则化的多视图学习,获得两个综合相似性矩阵并同时推断潜在的药物适应症。随后引入矩阵补全方法,在原药物-疾病关联矩阵中填补一些正项,再次执行多视图学习算法。最终采用集成策略来整合上述两个操作。1.多视图学习(ML)
现有数据集多含有固有噪声,单个相似度矩阵则多不完整且稀疏,以上因素都会影响预测方法的准确性。利用多视图学习不仅能基于不同角度充分反映实体特征多种相似性信息以进行关联预测,还可以有效地整合来自多个生物数据源的相似性信息。 多视图方法将药物和疾病的多种信息视为不同的视图,通过整合这些多数据源来提高预测性能。假设在药物空间中有 p=5个视图,对应于相似矩阵AR(1)、AR(2)、AR(3)、AR(4)、AR(5),并且疾病空间中有q=2个视图,表示为AD(1)、AD(2)。研究人员提出一个多视图学习的目标函数,它通过最小化AR中不同相似视图的不一致性来学习一个综合药物相似矩阵SR,根据多视图相似AD以同样的方式得到一个综合疾病相似矩阵SD 。此外,在目标函数中引入拉普拉斯图正则化项,防止学习到的综合相似度矩阵过拟合,最终用于预测药物-疾病关联矩阵F。下面的目标函数展示了多视图学习的全过程:
AR(ν)表示每个视图对应的药物相似度矩阵,AD(ν)表示疾病相似度矩阵,Tr(·)和‖·‖F分别表示矩阵的迹和Frobenius范数。LSR是SR的拉普拉斯矩阵,定义为LSR = DSR - (SRT + SR)/2。DSR是对角矩阵,其第i个对角项为∑j (SRij + SRji) / 2。LSD表示为 SD的拉普拉斯矩阵。A代表原始药物疾病关联矩阵,F是预测的关联矩阵。并且引入了两个参数λr和λd来平衡学习相似矩阵和预测关联矩阵。 固定SD和SR,上述目标函数转化为如下子问题:
上述方程对F取的导数并将其设置为零,得到:
最终方程直接求解为Sylvester方程。********2.矩阵补全的多视图学习(ML{MC})
通过临床试验验证的药物-疾病关联只有少数,而未知或潜在关联的数量要多得多,从而导致关联矩阵稀疏且不完整。因此引入有界矩阵补全(BMC)作为多视图学习方法的预处理步骤。填充药物-疾病关联矩阵中缺失的条目使多视图学习的信息更加丰富,有助于提高预测性能。公式如下:
‖X‖*表示X的核范数,定义为X的所有奇异值和。α是平衡核范数和误差项的参数。A是原始药物-疾病关联矩阵。Ω是包含A中所有已知条目的集合,pΩ 是到Ω上的投影算子。 上述模型可以通过交替方向乘子算法(ADMM)求解,设W是一个新的辅助矩阵:
增广拉格朗日函数变为:
Y是拉格朗日乘数,β > 0是惩罚参数。α和β设置均为10。然后使用ADMM交替更新Wk+1、Xk+1和Yk+1:
当BMC的最后一次迭代收敛时,得到一个更新的药物-疾病关联矩阵W,其中填充了稀疏的条目。并删除矩阵W中分数小于阈值(根据经验设置为 0.8)的条目。 得到更新后的关联矩阵W后,矩阵补全的多视图学习模型(ML{MC})可以表示为: 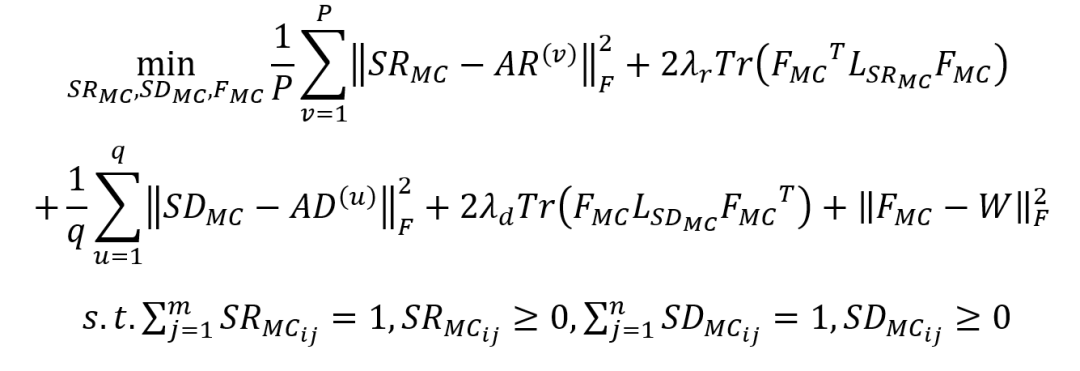
在ML{MC}中,MC作为一种预处理方法,在关联矩阵中引入更可靠的元素,丰富了药物网络和疾病网络之间的边缘,解决了稀疏关联矩阵的问题。但在MC有效提高数据质量的同时,也不可避免地会引入一些新的噪音,例如错误地预填充了某些交互信息。因此采用集成策略来整合它们的预测结果,以提高预测性能并避免噪声干扰。计算最终预测分数的过程描述如下:
其中F是ML方法的预测药物-疾病关联概率,FMC是由ML{MC}引入的预测关联概率。最大操作是取两个矩阵中每个元素的最大值,P代表最终的预测分数。结果与讨论使用10倍交叉验证和de novo测试来评估MLMC的性能。1.与其他药物重新定位方法比较****
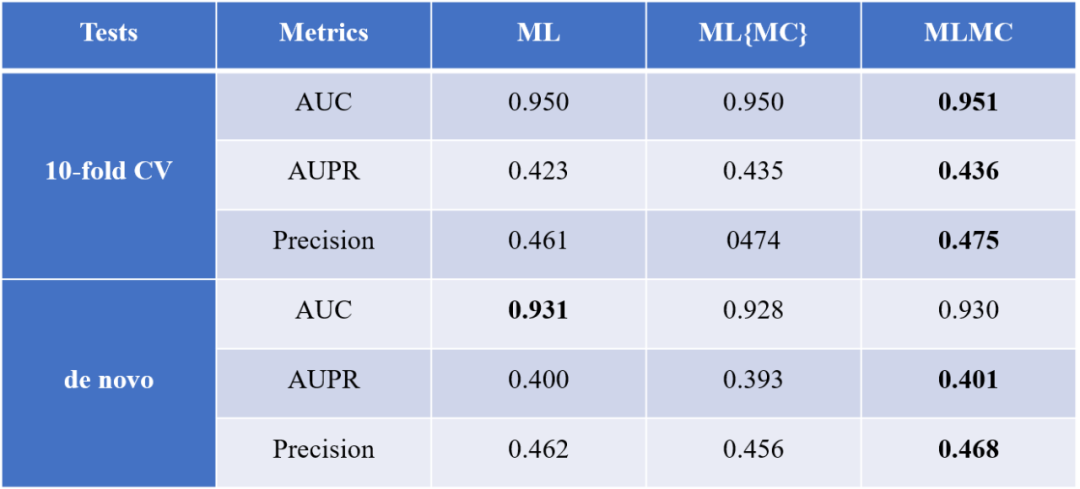
MLMC与MSBMF、HGIMC、BNNR、MBiRW和DrugNet这5种最先进的方法进行比较。表1表明,MLMC在经典数据集上的10倍交叉验证和de novo测试中均优于其他比较方法。 表1 MLMC与其他药物重新定位方法的 AUC、AUPR和精度值******[1]**********
为了消除不同方法的相似性差异,对MLMC和多相似性融合方法之间进行了综合比较。利用SNF算法更新五个药物相似性和两个疾病相似性以获得融合的药物和疾病相似性矩阵,将这些融合矩阵依次输入上述5种方法模型中,对应的方法分别重命名为BNNR-SNF、MBiRW-SNF和DrugNet-SNF。MSBMF和 HGIMC都是基于多源相似性的方法,因此无需处理。使用SNF算法的结果如表1所示。MLMC在10倍交叉验证和de novo测试中仍然达到最佳预测性能。10倍交叉验证和de novo测试的ROC和PR曲线分别如图2所示。 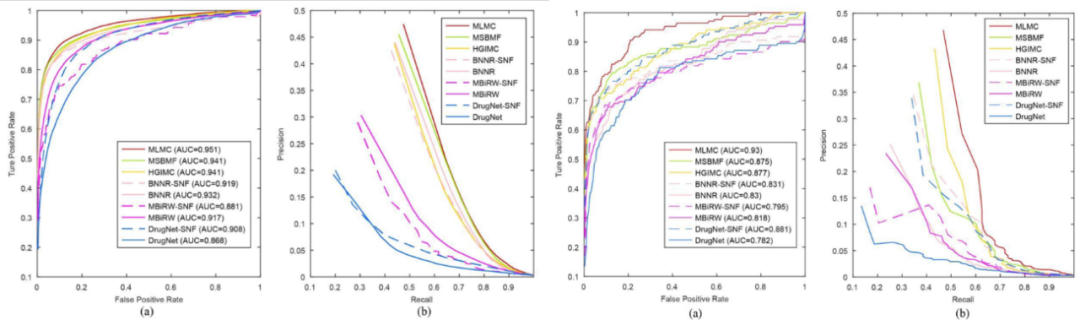
与其他最新的数据融合方法SNF、clusDCA、RWRF和RWRFN进行比较,将五个药物相似矩阵和两个疾病相似矩阵计算综合相似矩阵的过程替换为其他数据融合比较方法,实验结果如表2所示。MLMC在10倍交叉验证和de novo测试中表现最好,显示了多视图融合学习的优势。表2 MLMC与其他数据融合方法的AUC、AUPR和精度值******[1]**********
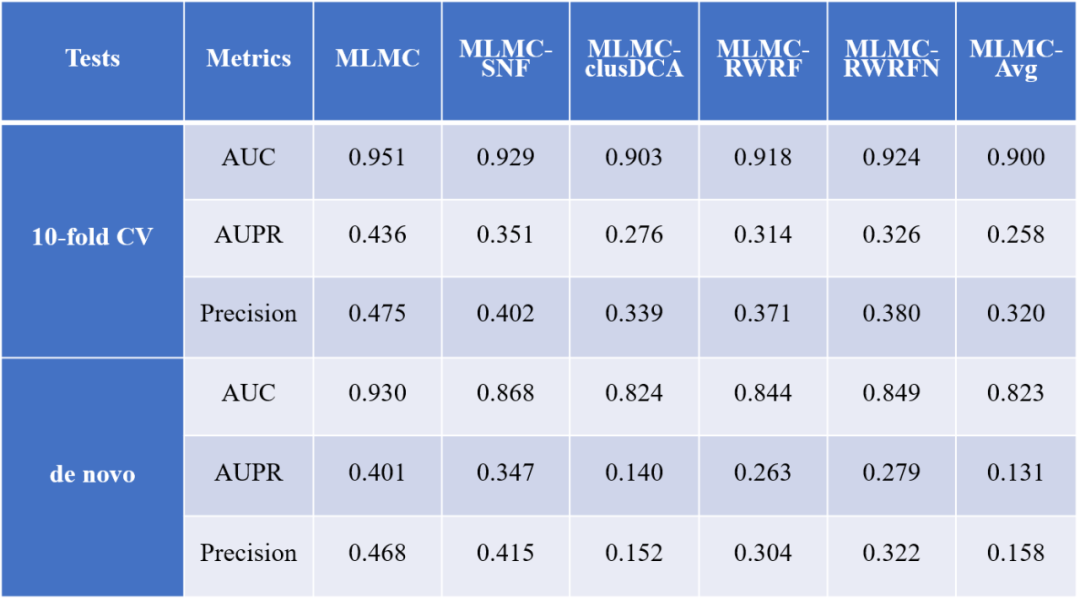
ML和ML{MC}作为MLMC模型的组件,分别评估其性能。表3显示了所有模型在10倍交叉验证和de novo测试的评估的结果。10倍交叉验证的结果表明MLMC效果最好,说明ML和ML{MC}的集成提升了预测性能。进一步发现ML{MC}的性能接近MLMC,并且两者都明显优于ML,这证明了MC在ML中的有效性。 表 3 ML、ML{MC}和MLMC的AUC、AUPR和精度值******[1]**********
为了确认其在实际应用中的性能,研究人员进行了案例研究。根据 MLMC的预测分数对所有可能的配对进行排名,文献检索验证的结果表明,10个预测结果中有6个被证实。选择四种具有代表性的药物作为例子进行案例研究,包含gemcitabine, methotrexate, leucovorin和doxorubicin。这四种药物相应的前五个候选适应症中有两个被CTD数据库所报道。**6.**其他数据集的实验
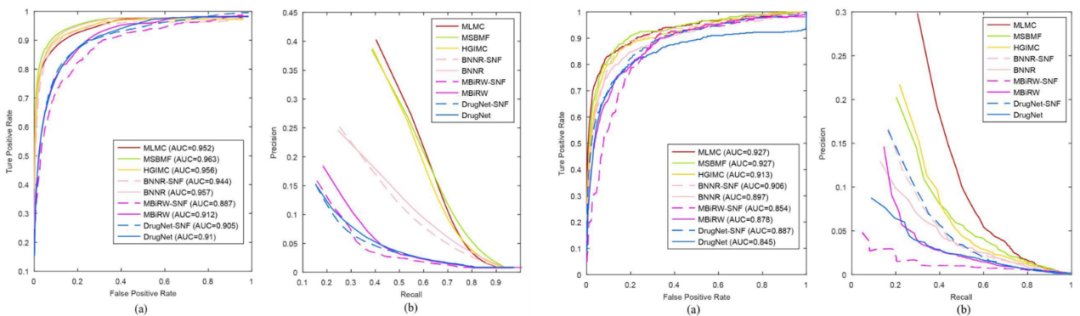
为了研究MLMC算法对不同数据集的适应性和可靠性,在Cdataset和Ydataset数据集上进行实验。实验结果表明MLMC在10倍交叉验证和中de novo测试中都获得最佳性能。图3显示了Cdataset数据集上10倍交叉验证和de novo测试的ROC和PR曲线。在Ydataset数据集上的ROC和PR曲线如图4所示。 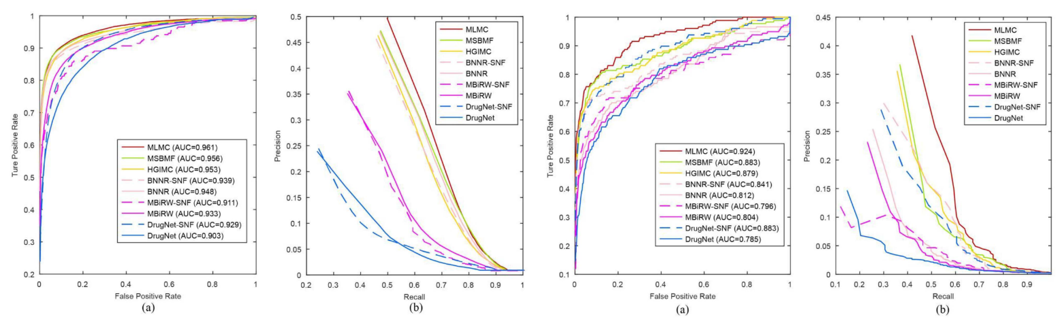
研究人员利用矩阵补全方法的多视图学习建立了一种新的药物重定位模型MLMC。该方法首先从五个药物相似性矩阵和两个疾病相似性矩阵中获得最佳相似性矩阵,同时更新药物-疾病关联矩阵。再引入矩阵补全为多视图学习模型添加一些正条目,以进一步提高多视图学习方法的预测性能。分析与验证表明MLMC优于其他现有的药物重新定位方法,为未来利用计算方法加速开发药物重定位提供了新的思路。
**参考文献 **
[1] Yan Y, Yang M, Zhao H, et al. Drug repositioning based on multi-view learning with matrix completion. Brief Bioinform. 2022. 23(3): bbac054.
供稿:邹萍萍
校稿:李诗良/沈子豪编辑:毛丽韫华东理工大学/上海市新药设计重点实验室/李洪林教授课题组▼招聘博后▼华东理工大学李洪林教授团队诚聘博士后