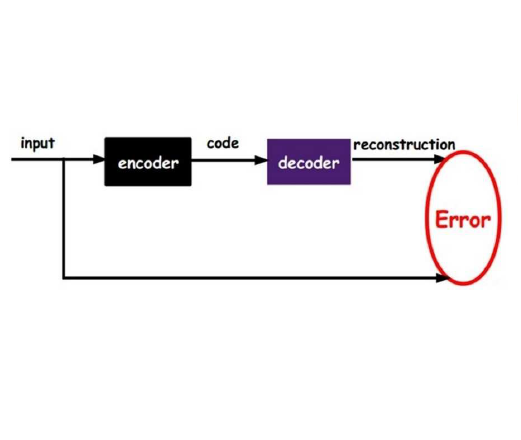
Purpose: This work addresses the detection of Helicobacter pylori (H. pylori) in histological images with immunohistochemical staining. This analysis is a time demanding task, currently done by an expert pathologist that visually inspects the samples. Given the effort required to localise the pathogen in images, a limited number of annotations might be available in an initial setting. Our goal is to design an approach that, using a limited set of annotations, is capable of obtaining results good enough to be used as a support tool. Methods: We propose to use autoencoders to learn the latent patterns of healthy patches and formulate a specific measure of the reconstruction error of the image in HSV space. ROC analysis is used to set the optimal threshold of this measure and the percentage of positive patches in a sample that determines the presence of H. pylori. Results: Our method has been tested on an own database of 245 Whole Slide Images (WSI) having 117 cases without H. pylori and different density of the bacteria in the remaining ones. The database has 1211 annotated patches, with only 163 positive patches. This dataset of positive annotations was used to train a baseline thresholding and an SVM using the features of a pre-trained RedNet18 and ViT models. A 10-fold cross-validation shows that our method has better performance with 91% accuracy, 86% sensitivity, 96% specificity and 0.97 AUC in the diagnosis of H. pylori. Conclusion: Unlike classification approaches, our shallow autoencoder with threshold adaptation for the detection of anomalous staining is able to achieve competitive results with a limited set of annotated data. This initial approach is good enough to be used as a guide for fast annotation of infected patches.
翻译:暂无翻译