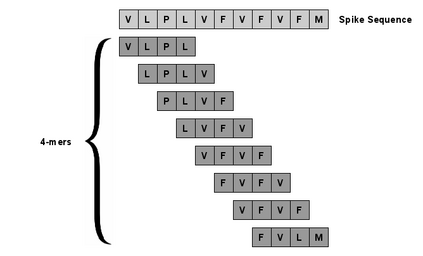
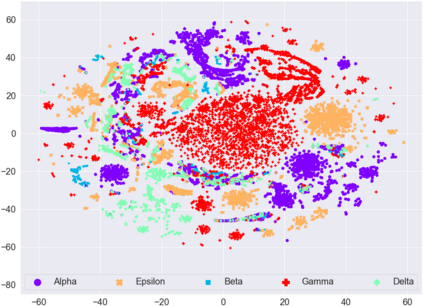
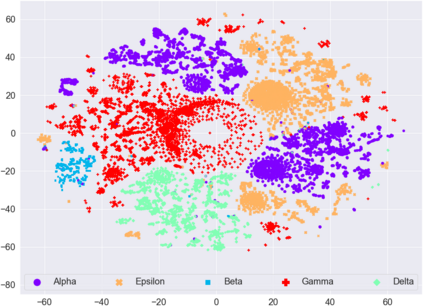
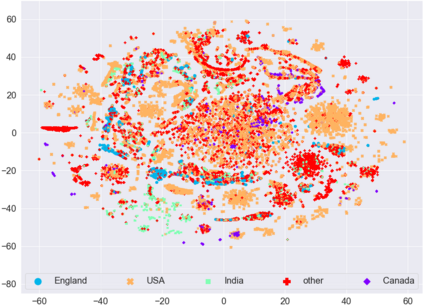
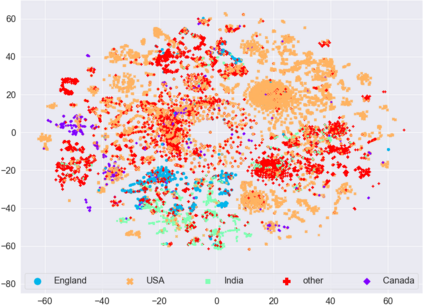
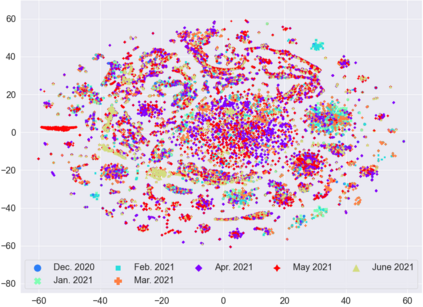
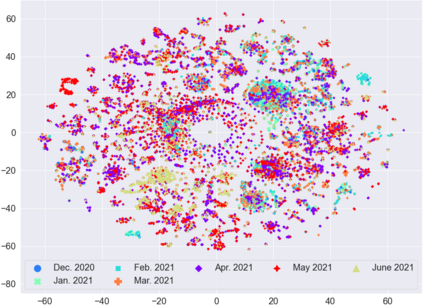
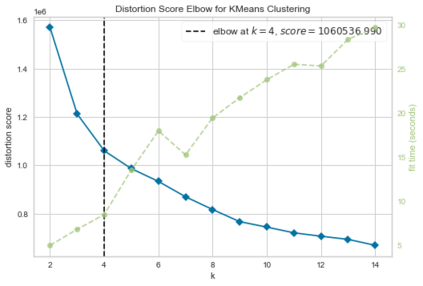
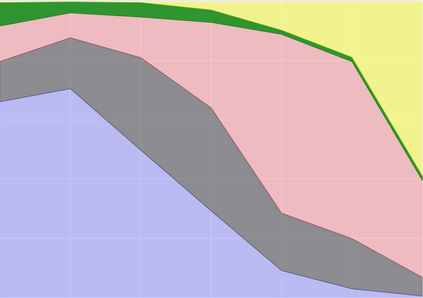
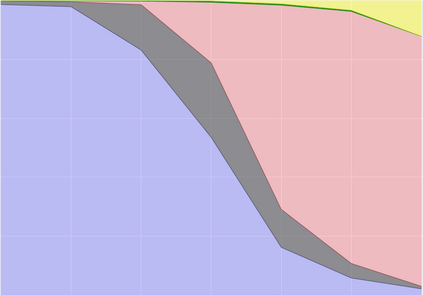
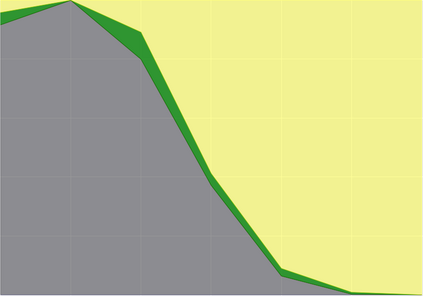
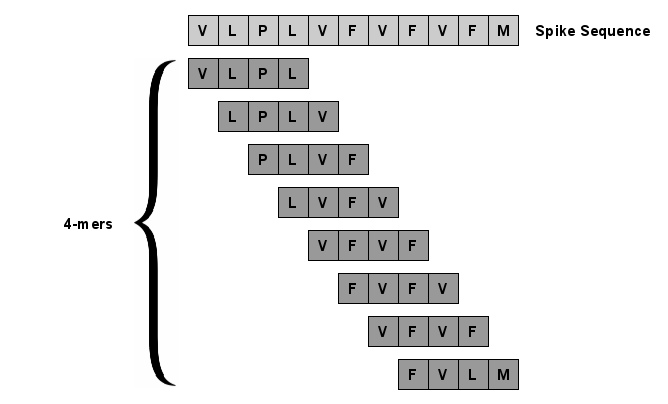
SARS-CoV-2, like any other virus, continues to mutate as it spreads, according to an evolutionary process. Unlike any other virus, the number of currently available sequences of SARS-CoV-2 in public databases such as GISAID is already several million. This amount of data has the potential to uncover the evolutionary dynamics of a virus like never before. However, a million is already several orders of magnitude beyond what can be processed by the traditional methods designed to reconstruct a virus's evolutionary history, such as those that build a phylogenetic tree. Hence, new and scalable methods will need to be devised in order to make use of the ever increasing number of viral sequences being collected. Since identifying variants is an important part of understanding the evolution of a virus, in this paper, we propose an approach based on clustering sequences to identify the current major SARS-CoV-2 variants. Using a $k$-mer based feature vector generation and efficient feature selection methods, our approach is effective in identifying variants, as well as being efficient and scalable to millions of sequences. Such a clustering method allows us to show the relative proportion of each variant over time, giving the rate of spread of each variant in different locations -- something which is important for vaccine development and distribution. We also compute the importance of each amino acid position of the spike protein in identifying a given variant in terms of information gain. Positions of high variant-specific importance tend to agree with those reported by the USA's Centers for Disease Control and Prevention (CDC), further demonstrating our approach.
翻译:与任何其他病毒一样,SARS-COV-2像任何其他病毒一样,在病毒传播过程中继续变异。与任何其他病毒不同的是,在GISAID等公共数据库中,SARS-COV-2的现有序列数量已经达到几百万。这一数量的数据有可能发现病毒的进化动态,但100万个数量级已经超出传统方法所能处理的范围,传统方法是为了重建病毒的进化历史,例如建立植物树。因此,需要设计新的和可扩展的方法,以便利用不断增长的病毒序列数量。由于确定变异体是了解病毒演变的一个重要部分,因此在本文件中,我们提出一种基于组合序列的方法,以确定目前主要的SARS-COV-2变异体。我们使用以美元为单位的特性矢量生成和高效的特性选择方法,我们的方法能够有效地确定变异体,并且能和可伸缩到数百万个序列。这种组合方法使我们得以在每种变体的变体位置上发现每一种变体的变体,我们每个变体在每种变体的变体的变体的变体上,每个变体的变体的变体在每种变体的变体的变体的变体的变体的变体上,我们在每一变体的变体上都的变体的变体的变体的变体的变体的变体的变体的变体的变体的变体的变种的变种的变种性的变种的变种性的变体的变种的变种的变体的变体的变种性的变种性的变种性的变种性的变种的变种性的变种性的变种性的变种的变种性的变种性的变种性的变种性的变种性的变种性的变种的变种性的变种性的变种性的变种性的变种性的变种性的变种性的变种性的变种性的变式的变种性上,,我们的变式的变种的变种性的变种的变种性的变种的变种性的变种的变种的变种的变种的变种的变种的变种的变种的变种的变种的变种的变种的变种