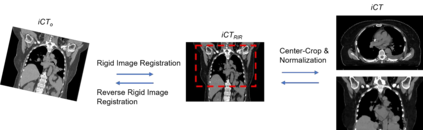
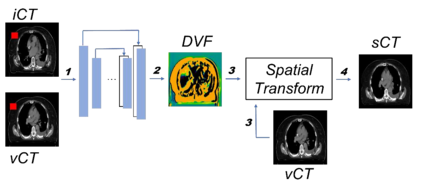
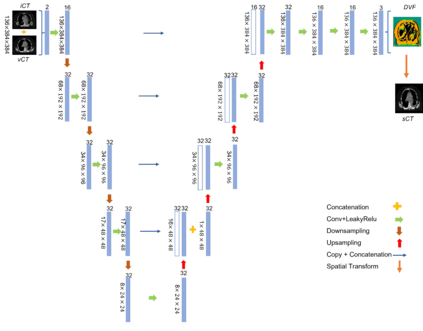
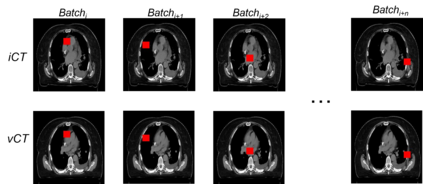
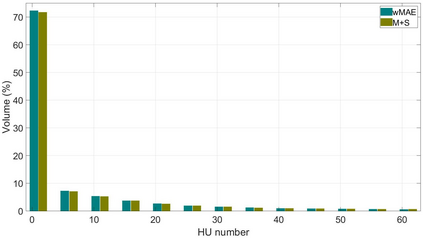
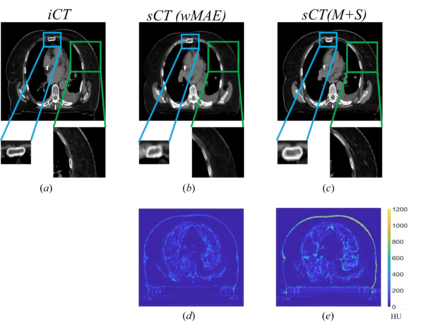
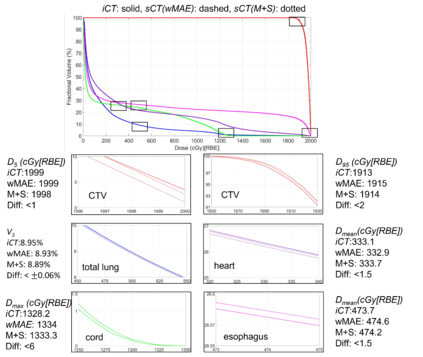
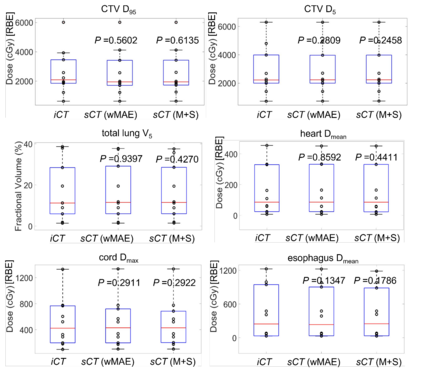
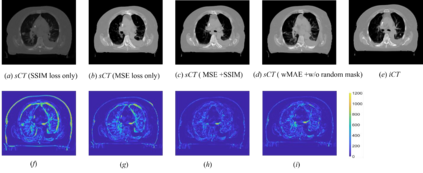
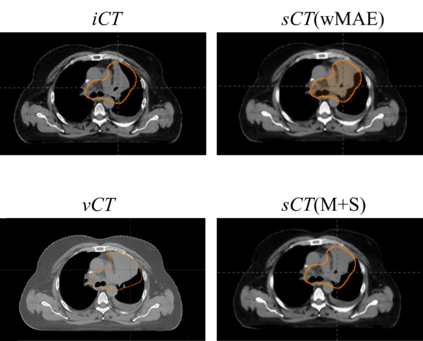
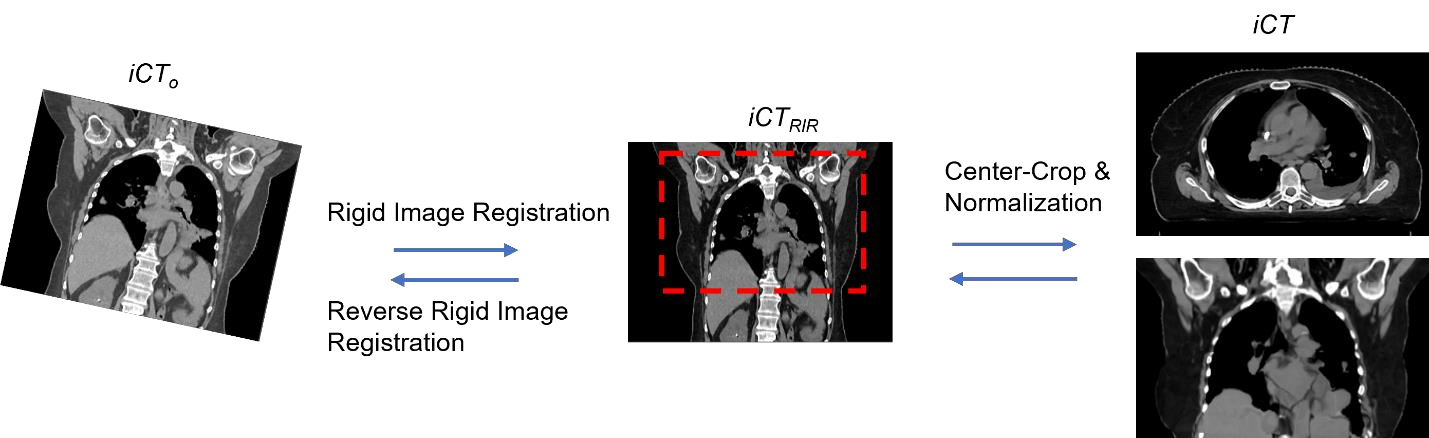
Purpose: In some proton therapy facilities, patient alignment relies on two 2D orthogonal kV images, taken at fixed, oblique angles, as no 3D on-the-bed imaging is available. The visibility of the tumor in kV images is limited since the patient's 3D anatomy is projected onto a 2D plane, especially when the tumor is behind high-density structures such as bones. This can lead to large patient setup errors. A solution is to reconstruct the 3D CT image from the kV images obtained at the treatment isocenter in the treatment position. Methods: An asymmetric autoencoder-like network built with vision-transformer blocks was developed. The data was collected from 1 head and neck patient: 2 orthogonal kV images (1024x1024 voxels), 1 3D CT with padding (512x512x512) acquired from the in-room CT-on-rails before kVs were taken and 2 digitally-reconstructed-radiograph (DRR) images (512x512) based on the CT. We resampled kV images every 8 voxels and DRR and CT every 4 voxels, thus formed a dataset consisting of 262,144 samples, in which the images have a dimension of 128 for each direction. In training, both kV and DRR images were utilized, and the encoder was encouraged to learn the jointed feature map from both kV and DRR images. In testing, only independent kV images were used. The full-size synthetic CT (sCT) was achieved by concatenating the sCTs generated by the model according to their spatial information. The image quality of the synthetic CT (sCT) was evaluated using mean absolute error (MAE) and per-voxel-absolute-CT-number-difference volume histogram (CDVH). Results: The model achieved a speed of 2.1s and a MAE of <40HU. The CDVH showed that <5% of the voxels had a per-voxel-absolute-CT-number-difference larger than 185 HU. Conclusion: A patient-specific vision-transformer-based network was developed and shown to be accurate and efficient to reconstruct 3D CT images from kV images.
翻译:目的:在一些质子治疗设施中,病人定位依靠两个固定的互相垂直的、倾斜角度固定的二维kV图像,因为当前没有三维的在床上成像。在kV图像中,由于病人的三维解剖被投影到二维平面上,特别是当肿瘤位于高密度结构(如骨头)后面时,肿瘤的可见性非常有限。这可能导致较大的病人定位误差。解决方案是从在治疗位置取得的治疗等中心的kV图像重建三维CT图像。
方法:开发了一个基于视觉变换块的非对称自动编码器网络。数据来自1个头颈部患者:2个正交的kV图像(1024x1024体素)、1个带填充的3D CT(512x512x512),在取得kV图像之前从射线CT-On-Rails中获取,以及基于CT的2个数字重建放射学图像(DRR)(512x512)。我们每隔8个体素对kV图像进行采样,每隔4个体素对DRR和CT进行采样,从而形成了一个由262144个样本组成的数据集,其中每个方向的图像的尺寸为128。在训练中,同时利用了kV和DRR图像,并鼓励编码器从两种图像中学习联合特征图。在测试中,仅使用独立的kV图像。通过按照它们的空间信息连接模型生成的sCT的sCT连接,实现了全尺寸的合成CT(sCT)。使用平均绝对误差(MAE)和每体素绝对CT数字差异体积直方图(CDVH)评估了合成CT(sCT)的图像质量。
结果:该模型达到了2.1秒的速度和小于40HU的MAE。CDVH显示<5%的体素有大于185 HU的每体素绝对CT数字差异。
结论:此前所述基于视觉变换器的患者特异性网络被开发出来,并且已经被证明可以准确、高效地从kV图像中重建出3D CT图像。