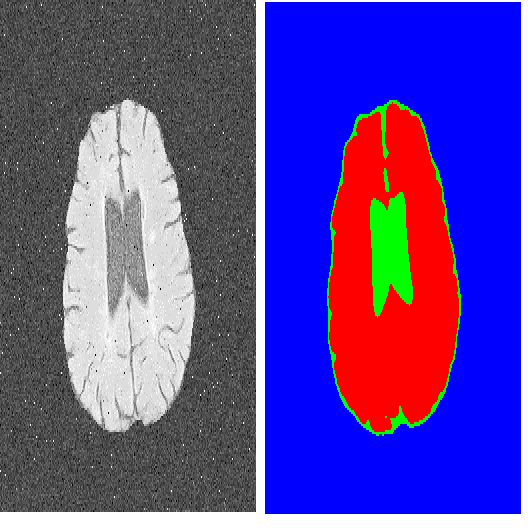
Recent studies on contrastive learning have achieved remarkable performance solely by leveraging few labels in the context of medical image segmentation. Existing methods mainly focus on instance discrimination and invariant mapping. However, they face three common pitfalls: (1) tailness: medical image data usually follows an implicit long-tail class distribution. Blindly leveraging all pixels in training hence can lead to the data imbalance issues, and cause deteriorated performance; (2) consistency: it remains unclear whether a segmentation model has learned meaningful and yet consistent anatomical features due to the intra-class variations between different anatomical features; and (3) diversity: the intra-slice correlations within the entire dataset have received significantly less attention. This motivates us to seek a principled approach for strategically making use of the dataset itself to discover similar yet distinct samples from different anatomical views. In this paper, we introduce a novel semi-supervised 2D medical image segmentation framework termed Mine yOur owN Anatomy (MONA), and make three contributions. First, prior work argues that every pixel equally matters to the model training; we observe empirically that this alone is unlikely to define meaningful anatomical features, mainly due to lacking the supervision signal. We show two simple solutions towards learning invariances - through the use of stronger data augmentations and nearest neighbors. Second, we construct a set of objectives that encourage the model to be capable of decomposing medical images into a collection of anatomical features in an unsupervised manner. Lastly, we both empirically and theoretically, demonstrate the efficacy of our MONA on three benchmark datasets with different labeled settings, achieving new state-of-the-art under different labeled semi-supervised settings
翻译:最近的对比学习研究仅利用少量标签在医学图像分割的上下文中实现了显著的性能。现有的方法主要侧重于实例区分和不变映射。然而,它们面临三个常见问题:(1)尾部:医学图像数据通常遵循隐式的长尾类分布。盲目利用训练中的所有像素会导致数据不平衡问题,并造成性能恶化;(2)一致性:由于不同解剖特征之间的类内变化,仍不清楚分割模型是否学习了有意义且一致的解剖特征;和(3)多样性:整个数据集内部切片之间的相关性受到显著较少的关注。这促使我们寻找一种基于数据集本身来发现不同解剖视图的类似但不同样本的原则性方法。在本文中,我们介绍了一种新颖的半监督2D医学图像分割框架,称为Mine yOur owN Anatomy(MONA),并做出了三个贡献。首先,先前的工作认为每个像素对模型训练同样重要。但是,由于缺乏监督信号,光这个不太可能定义有意义的解剖特征。我们展示了两种简单的解决方案,以通过使用更强的数据增强和最近邻学习不变性。其次,我们构建了一组目标,可以在无监督的情况下鼓励模型能够将医学图像分解为解剖特征的集合。最后,我们实践和理论上证明了我们的MONA在三个基准数据集上的有效性,实现了不同标记半监督设置下的新的最先进水平。