作者 | 王永康审核 | 熊展坤
今天给大家分享美国密西根州立大学Jiliang Tang教授团队表达在KDD 2022上的一篇论文“Graph Neural Networks for Multimodal Single-Cell Data Integration”。研究针对单细胞的三个关键任务:模态预测、模态匹配和联合嵌入,提出了一种通用图神经网络scMoGNN。该方法对单细胞的不同模态分别进行建模,根据单细胞测序数据构建GNN网络,将不同组学的生物知识添加到图网络中作为额外的结构性信息,从而捕捉细胞和模态之间的高阶结构关系。同时,该方法表现出高度灵活性,可在不同模式的单细胞任务进行扩展使用,有效解决传统的单细胞数据整合技术的局限性。实验结果表明,该方法表现出优异性能,在三个任务上均领先当前基准模型。
Part1概要
单细胞技术的快速发展使得同时测量细胞中多种模式的分子特征成为可能,从而为单细胞提供了前所未有的多模态数据。这不仅为不同模态之间的关系带来了新的见解,也提供了对细胞系统的整体理解和认识。如何有效地利用多模态数据的互补信息来研究单细胞状态,并在利用多模态数据的同时整合大量的单模态数据,成为单细胞基因组学的巨大的挑战。对此,近期有相关研究整理出单细胞的三大主要任务:
- 模态预测:旨在根据一种模态的特征信息,去预测另一种模态的特征信息;
- 模态匹配:侧重于识别在不同模态之间具有对应关系的细胞;
- 联合嵌入:将不同模态的特征信息嵌入到统一的低维空间中,便于下游任务的分析;
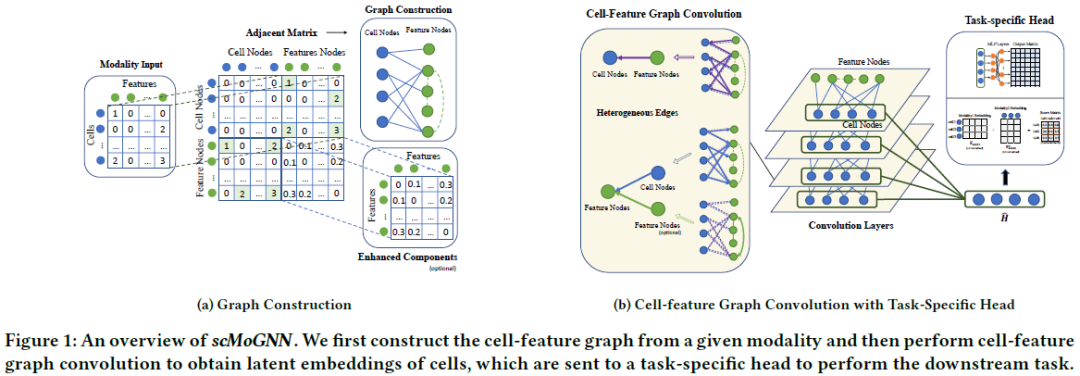
作者认为当前的大部分研究方法将每个细胞视为单独的输入,不考虑细胞之间或不同模态之间可能的交互关系,然而这种交互信息在单细胞数据的稀疏特征学习过程中是必不可少的。因此,作者提出一个通用图神经网络scMoGNN,用于解决不同模态的单细胞数据整合问题。该方法通过将单细胞与模态特征之间的交互关系建模为图结构,进而采用GNN网络通过结构信息解决先前单细胞数据整合技术的局限性。基于构建的图结构,可以很容易地将外部的生物知识(如基因之间的相互作用)整合到图网络中。
Part2方法
1概念定义
本文主要使用三种模态的单细胞数据,包括GEX(转录组数据),ATAC(DNA数据),ADT(蛋白质数据)。每种类型的数据可表示为,即N个细胞数目,每个细胞的特征维度为K。同时对于每种模态数据,可以针对细胞和特征信息构建二部图,其中为细胞节点,表示特征节点。以下定义单细胞的三个关键任务:
- 模态预测:给定一种模态数据,目标是为每个细胞的特征值预测另一种模态信息。也即需要学习一种模态转化方法,使得,同时使用均方根误差(RMSE)来量化初始和预测的特征值之间的差异。
- 模态匹配:给定两种模态数据和,目的是学习两种模态转换方法和,将两种类型数据转化至统一特征空间中,并使得两者之间的差异最小。即,是自定义的打分函数,用于评估模态特征在转化空间的相似性。
- 联合嵌入:给定两种模态数据和,目的是学习三种模态转化方法,对两种模态数据进行有效地特征提取,便于下游任务分析。即,将两种模态分别转换后进行特征拼接,再次转化后获得模态嵌入特征。
2图网络结构
在二部图中,表示细胞节点和特征节点之间的关联状态,整张图的形式可以描述为下式,对角线分别表示细胞节点之间以及特征节点之间的关系。由于细胞之间不存在先验生物知识,因此;特征节点之间的联系需要根据实际的生物任务来具体设置。 鉴于图上存在不同的关系边类型(如细胞-细胞,细胞-特征,特征-特征),需要分别进行处理。具体来说,使用不同的网络参数来对节点-边信息进行聚合,进而使得不同类型的节点产生具有差异性的分布信息。例如,对于细胞与其邻居特征之间的关系可描述为如下式。表示边的权重, 和表示待学习的模型参数,用于权重的标准化过程。
同理,对于特征与其邻居细胞之间的关系可描述为如下式:
因此在图网络消息传播过程中,存在如下两种传播方式:
3任务调整
模态预测
针对不同的模态任务,作者补充相应的生物学知识用于增强特征信息。例如在GEX-ADT和GEX-ATAC中,作者使用MSigDB数据库中的hallmark基因集,用于标识基因特征之间的生物关联性。因此细胞和基因特征的图结构可描述为下式。 标识基因特征之间的关系。 由于细胞节点和特征节点标识不同的生物含义,因此在消息传播时无法直接将两者进行整合。作者分别处理不同类型邻域内的节点消息,具体如下式: ; 是可学习超参数,用于确定特征内部和细胞-特征之间的消息比例。模态信息在经过多层的图卷积操作后,从最终的卷积结果中提取细胞节点的嵌入并进行聚合,而后通过全连接层转换到目标模态空间内。表示不同细胞节点嵌入的权重。
模态匹配
任务目标要求预测一对不同模态的单细胞数据,两数据集中相应数据具有成对性(源于同一细胞)的评分。首先根据模态预测的过程,分别对两种模态的数据各自进行cell-feature图的构建,以及细胞嵌入的获取,即。而后通过计算细胞嵌入之间的余弦相似度来获得两模态的打分矩阵,,并通过softmax方法将评分转化为不同细胞之间的相似性概率: 除了相似性评分外,作者构建了预测损失和重构损失用于增强模态匹配的效果,具体如下: 表示两模态的原始数据,表示相应的模态转化器。最终作者将和结合,作为模态匹配任务的目标函数。
联合嵌入
任务目标是学习不同模态细胞的嵌入,用于更好的描述细胞内部的异质性,便于下游的任务分析(如批次效应移除)。作者将不同模态的信息依据模态预测方式进行特征提取,并将得到细胞特征进行拼接,形成一张更大的cell-feature图。鉴于细胞类型信息的重要性,作者根据新的图结构,获得细胞节点的嵌入信息,即给每个细胞添加T维度的信息,同时评估细胞所属类型的概率;该任务的目标函数如下: 表示对两种模态数据通过LSI算法进行预处理,而后将特征拼接,以此构建重构损失项。式子第二项表示分类损失,第三项为正则化。
Part3实验结果
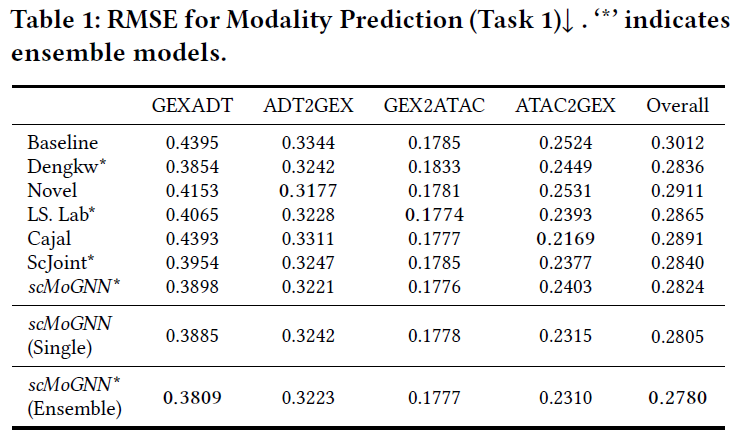
研究评估了scMoGNN框架针对上述三个任务的有效性,并在本实验中遵循多模态单细胞数据竞赛中的官方设置和数据集,将所提出框架与竞赛中的优胜者进行比较,具体结果如下: 在模态预测任务中,总体结果表明了scMoGNN框架的有效性,并且在某些特定情况下该方法具有巨大的性能优势。
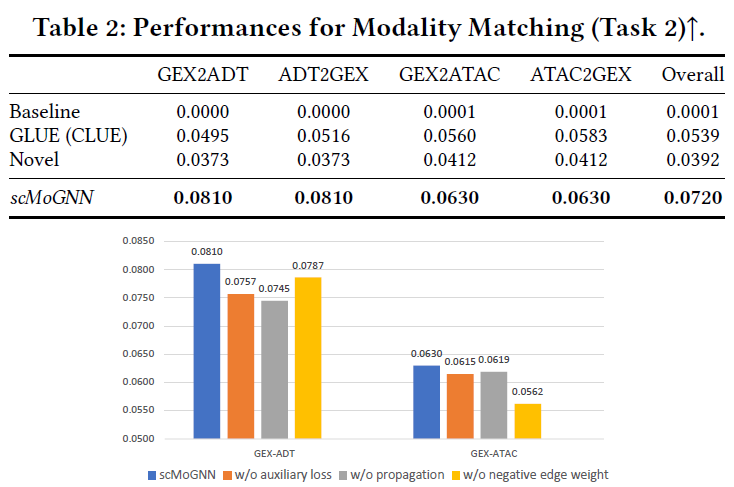
在模态匹配任务中,结果表明scMoGNN框架在不同模态的匹配转换过程中,均达到最优的效果。
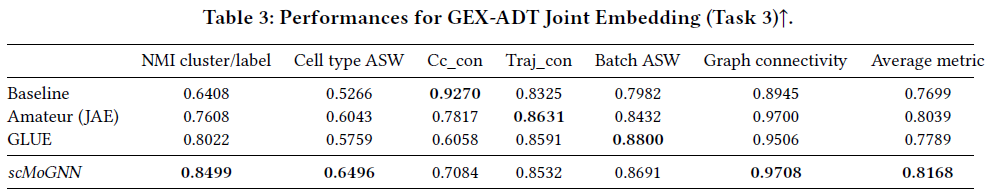
在联合嵌入任务中,scMoGNN在GEX-ADT任务中的表现明显优于其他两个模型。
更多详细的实验结果请查看原文。
Part4总结
本研究提出了一个基于scMoGNN的通用框架,通过捕获细胞和特征之间高阶结构信息,以此用于多模态单细胞数据的整合。实验结果表明该方法能有效地应用于单细胞的三个关键任务,情态预测,情态匹配和联合嵌入,相对于任务中的其他模型,表现出具有显著的优势。
Part5参考文献
论文地址:https://dl.acm.org/doi/abs/10.1145/3534678.3539213 代码地址:https://github.com/OmicsML/dance