生成式人工智能(Generative Artificial Intelligence,GenAI)已成为推动生物信息学变革的重要方法,它常常促进基因组学、蛋白质组学、转录组学、结构生物学以及药物发现等领域的进展。为系统识别和评估这些不断增长的研究成果,本综述依据系统性综述与荟萃分析的首选报告项目(PRISMA)方法,提出了六个研究问题(Research Questions, RQs)。其目标在于评估GenAI在方法学创新、预测性能和专业化方面的关键策略,并识别在高级建模、数据密集型发现与综合生物分析中的潜在前沿方向。 RQ1 强调了GenAI在多个生物信息学子领域(如序列分析、分子设计与综合数据建模)中的多样化应用,这些应用通过模式识别与输出生成展现出优于传统方法的性能。 RQ2 指出,经过适配的专用模型架构优于通用模型,其优势归因于有针对性的预训练与上下文感知策略。 RQ3 识别出GenAI在分子分析与数据整合等生物信息学领域中的显著益处,这些方法提升了复杂分析的准确性并降低了错误率。 RQ4 表明在结构建模、功能预测与合成数据生成等方面取得了改进,并通过标准化基准进行了验证。 RQ5 指出当前的主要限制包括可扩展性不足与数据偏差,这些问题影响了模型的泛化能力,并提出未来研究应聚焦于稳健评估与具备生物学基础的建模方法。 RQ6 说明分子数据集(如 UniProtKB 与 ProteinNet12)、细胞数据集(如 CELLxGENE 与 GTEx)以及文本资源(如 PubMedQA 与 OMIM)为GenAI模型的训练与泛化提供了广泛支持。
关键词: 生成式人工智能,Transformer模型,蛋白质语言模型,多组学整合,精准医学
1 引言(Introduction)
近年来,生物信息学经历了前所未有的转型,而这一变革在很大程度上得益于生成式人工智能(Generative Artificial Intelligence,GenAI)的发展 [1]。传统的生物数据分析主要依赖基于规则的方法、统计模型以及针对特定任务设计的专用算法 [2, 3]。然而,从基因组序列到蛋白质结构,再到单细胞转录组的现代组学数据,其庞大的数据量与复杂性对传统方法提出了严峻挑战 [4]。随着生成模型(尤其是基于Transformer的模型和基础模型,foundation models)的快速发展,计算生物学正经历着范式重构 [5, 6]。 基于这些进展,本文系统综述了生成式人工智能在生物信息学中的最新研究进展,归纳了其在生成建模方面的发展脉络,梳理了其在基因组学、蛋白质组学与多组学中的主要应用,并指出了阻碍其可靠落地的关键挑战,同时展望了推动稳健且可解释生物信息建模的未来方向。这一研究至关重要,因为GenAI正逐步渗透到生物研究的各个层面,涵盖数据解读、分子设计以及系统级分析等创新方向。
1.1 生成式人工智能在生物数据中的优势
GenAI模型在从大型、未标注数据集中捕捉上下文关系方面表现卓越,尤其适用于数据噪声大或缺乏标注的生物学任务 [7, 8]。通过在大规模序列、分子图或基因表达谱等数据上进行训练,这些模型能够学习高维模式,并以极少监督完成下游推理任务 [8]。与传统模型相比,生成式方法具有更强的灵活性,支持零样本(zero-shot)、少样本(few-shot)以及迁移学习(transfer learning)[9]。 此外,GenAI的研究已扩展到对生物系统的结构、空间与功能特征进行编码,从而支持多样化的生物建模任务 [10, 11]。这些模型促进了诸如基因调控元件预测、蛋白质功能注释、药物靶点相互作用建模以及基于单细胞数据的细胞特征生成等应用。其能够生成符合生物规律的输出、整合异质模态数据并支持自然语言交互的能力,大大拓展了生物信息学研究的边界 [12]。
1.2 当前研究的不足
现有综述文献 [13, 6, 14, 15, 16] 探讨了大型语言模型(LLMs)和基础模型在生物信息学中的作用,重点覆盖蛋白质结构预测、药物发现、基因表达分析和调控序列注释等领域。然而,只有少数综述对生成式人工智能在生物信息学中的作用进行了系统评估。 其中四项研究 [13, 6, 14, 15] 讨论了多模态整合、蛋白质建模与药物发现等应用,仅有两项 [13, 6] 详细探讨了指令微调(instruction tuning)。尽管多智能体学习系统(multi-agent learning systems)、自动化流程与会话式接口在构建智能生物信息系统中具有重要意义,但这些方向在现有研究中仍缺乏深入探讨与融合,限制了GenAI驱动的发现与分析潜力。 此外,当前研究普遍忽视了这些系统层面的关键因素,导致对如何实现GenAI在生物信息学工作流中的无缝集成、实时交互与可扩展部署的理解存在明显缺口。 为填补这些空白,本文聚焦以下 11 个主题维度: * 多领域覆盖(MDC, Multi-Domain Coverage) * 生成模型聚焦(GMF, Generative Model Focus) * 模型架构多样性(MAD, Model Architecture Diversity) * 指令微调与提示学习(ITP, Instruction Tuning and Prompting) * 分词与嵌入设计(TED, Tokenization and Embedding Design) * 多智能体学习系统(MALS, Multi-Agent Learning Systems) * 计算型GenAI工作流自动化(CGWA, Computational GenAI Workflow Automation) * 会话式接口(CI, Conversational Interfaces) * 多模态整合(MMI, Multi-Modal Integration) * 蛋白质设计与结构生物学(PDSB, Protein Design and Structural Biology) * 药物发现应用(DDA, Drug Discovery Application)
通过这些维度,本研究旨在提供一个全面而综合的视角,展示GenAI如何重塑生物信息学研究与实践。
1.3 研究目标与研究问题(Purpose and Research Questions, RQs)
本综述的核心目标是系统评估GenAI在生物信息学中的演变角色,包括其应用、模型架构及在特定生物领域中的影响。研究由以下 六个核心研究问题(RQs) 引导,以考察GenAI方法学、领域性能与新兴挑战: * RQ1:GenAI模型在生物信息学中如何应用?
具体而言,它们实现了哪些新任务,与传统方法相比有何改进? 目的: 探讨GenAI在生物信息学中的实际应用及其相对于传统方法的优势。 * RQ2:哪些模型与架构最为有效?
例如,通用大型语言模型与领域专用模型在生物序列数据上的表现有何差异? 目的: 识别在生物数据建模中表现最佳的模型类型与架构特征。 * RQ3:哪些生物信息学领域最能从GenAI中受益?
目的: 确定GenAI在不同生物领域中的主要应用与优势体现。 * RQ4:GenAI如何改进蛋白质结构/功能预测、多组学数据整合及代码生成等任务?有哪些基准或案例验证其效果?
目的: 评估GenAI在核心生物任务中的提升表现及其实证依据。 * RQ5:当前生成模型在数据需求、可解释性与生物知识整合方面存在哪些局限?未来方向是什么?
目的: 识别可扩展性、透明性与生物融合性方面的瓶颈,并展望未来研究方向。 * RQ6:分子、细胞与文本/知识型数据集如何分别或协同促进GenAI模型的训练与泛化?
目的: 理解不同生物数据模态如何塑造GenAI的学习、整合与泛化能力。
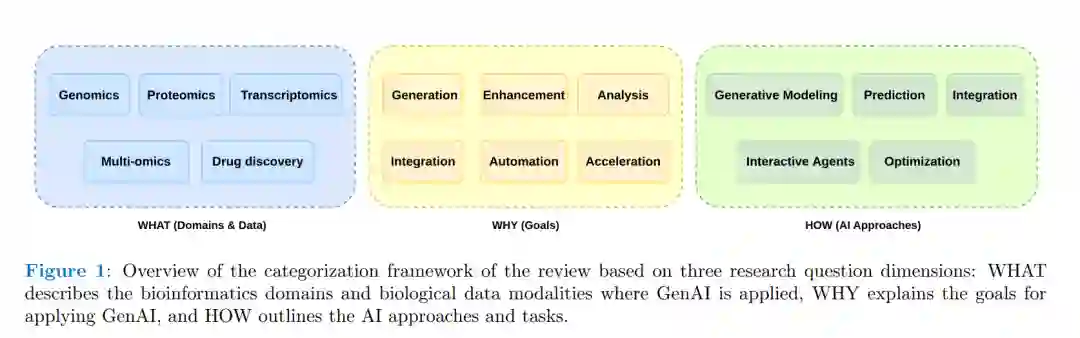
本文的框架(见图1)以“WHAT、WHY、HOW”三个问题为主线,明确了GenAI驱动生物信息学的应用领域、研究动机与计算策略。
1.4 本文贡献(Contributions)
本文在生成式人工智能与生物信息学的交叉研究中做出了以下 五项主要贡献: 1. 系统性文献综述: 全面分析了GenAI在基因组序列建模、蛋白质表示与设计、药物发现以及单细胞分析中的应用,揭示了其带来的新能力。 1. 现有综述比较: 对比了既有文献的关注领域,揭示了当前研究的不足与空白。 1. 六个研究问题的提出: 系统探讨了GenAI的应用、模型架构、领域表现与面临的挑战,展示其在多种生物任务中的日益增长的影响力。 1. 任务改进与基准分析: 详细讨论了近期研究中的任务改进与性能评估,验证了生成模型在生物信息学中的有效性。 1. 未来研究方向: 提出了基于生物学原理的GenAI框架构建路径,强调将LLM作为推理模块、在可验证工具中实现输出落地,以提升模型的可靠性;同时建议发展多模态生物整合的新方法,以推动科学发现导向的GenAI模型。 1. 关键数据集分析: 将核心数据资源划分为分子、细胞与文本模态三类,系统总结了它们在GenAI与LLM生物信息学应用中的作用与来源。
1.5 论文结构(Paper Organization)
本文共分为六个部分: * 第2节 讨论相关工作; * 第3节 描述系统综述所采用的方法; * 第4节 回答并分析六个核心研究问题(RQs); * 第5节 综合讨论研究结果,提出观察与见解; * 第6节 总结全文,归纳主要发现并展望GenAI在生物信息学中的未来作用。
论文整体组织框架如 图2 所示。