导语
2024年8月,国际顶尖著名杂志 Nature Methods 发表特刊(Special issue),探讨人工智能如何在生物学中广泛使用并为生物学家带来巨大帮助。特刊通过评论文章(Comment)、观点综述文章(Perspective)、原创研究文章(Research Article)等,讨论了计算生物学领域的各方面话题。**
研究领域:AI for Biology,人工智能,基因组学,蛋白质组学,复杂生命系统******
董弘禹 | 编译
文章标题:Focus on advanced AI in biology 期刊名称:Nature Methods 特刊链接:https://www.nature.com/collections/ahhdhbhgha
本期 Nature Methods 特刊主要包括以下主题:
- 大语言模型在生物医学领域(如电子病历等临床数据)的应用;
- Transformer架构和大模型帮助解析单细胞数据和高维空间组学数据;
- 开发从序列映射到表型的模型进而揭示调控机制;
- 采用蛋白质模型预测生物分子(蛋白-蛋白、蛋白小分子)间相互作用;
- 基于AI的蛋白质工程改进Crisper碱基编辑系统并塑造蛋白质组学;
- 通过多模态深度学习模型促进生物图像的准确解析和大脑神经系统重建;
- 利用人工智能方法解读免疫系统异质性以及肿瘤发生发展;
探讨AI发展值得注意的问题与挑战:包括数据泄露问题、伦理隐私问题、可解释性问题。
特刊内容丰富,能够帮助领域内的研究者速览生物信息学各方向的前沿进展,并促进学科交叉与融合。鉴于此,我们计划对特刊内的重点文章进行详细解读与编译,以飨读者。以下对第一篇社论(Editorial)文章的编译。
文章标题:Embedding AI in biology 文章来源:Nature Methods 文章链接:https://www.nature.com/articles/s41592-024-02391-7
先进的人工智能方法正在迅速改变生物数据的获取和分析方式。
作为致力于传播生命科学方法发展的期刊编辑,我们和我们服务的社区群体有共同的目标——一直在人工智能浪潮做准备,这个浪潮将会影响生命科学的几乎所有领域。AI并不是一个新趋势,我们在过去几年中发表的几篇社论中已经有所涉及,在2021年的年度方法中(Method of the Year 2021),我们强调并赞赏高精度蛋白质结构预测的开创性成就。然而,计算方法(如生成式模型)的迅猛发展正方方面面影响我们的生活,进而启发我们组织这个专刊,我们通过收集行业各位领军人物的意见想法,突出点明了领域前沿方向与进展。
AI for Biology 这个主题内容非常丰富和复杂。尽管我们在这个话题上涵盖了广泛的方向,但我们承认这份专刊仍然不能涵盖到方方面面。在我们深入了解AI在不同领域潜在影响的一系列评论文章之前,我们邀请您阅读James Zou和他的同事们的引导性文章,该文章介绍了大语言模型——机器学习中最具影响力的最新发展之一——以及它们在生物研究中的广泛应用[1]。
AI应用在基因组学(如单细胞、空间转录组学)的经典例子
高级机器学习方法对数据的需求很大。随着高通量组学技术(特别是在单细胞水平)的快速发展,具有多模态的超大数据集(一般涵盖数百万细胞)为模型训练提供了理想的数据来源。在一篇观点文章中,Fabian Theis和他的同事们提供了一个全面的概述,介绍了Transformer(一种强大的深度学习架构)及其在单细胞分析中的应用。通过制定预训练策略并利用Transformer架构,擅长多种下游任务的大模型在许多领域越来越受欢迎[2]。 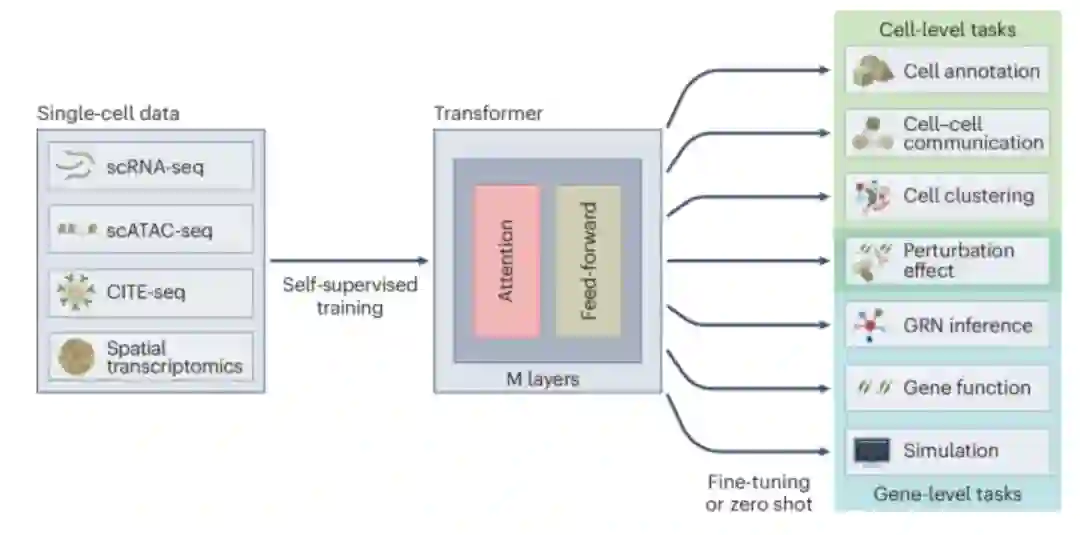
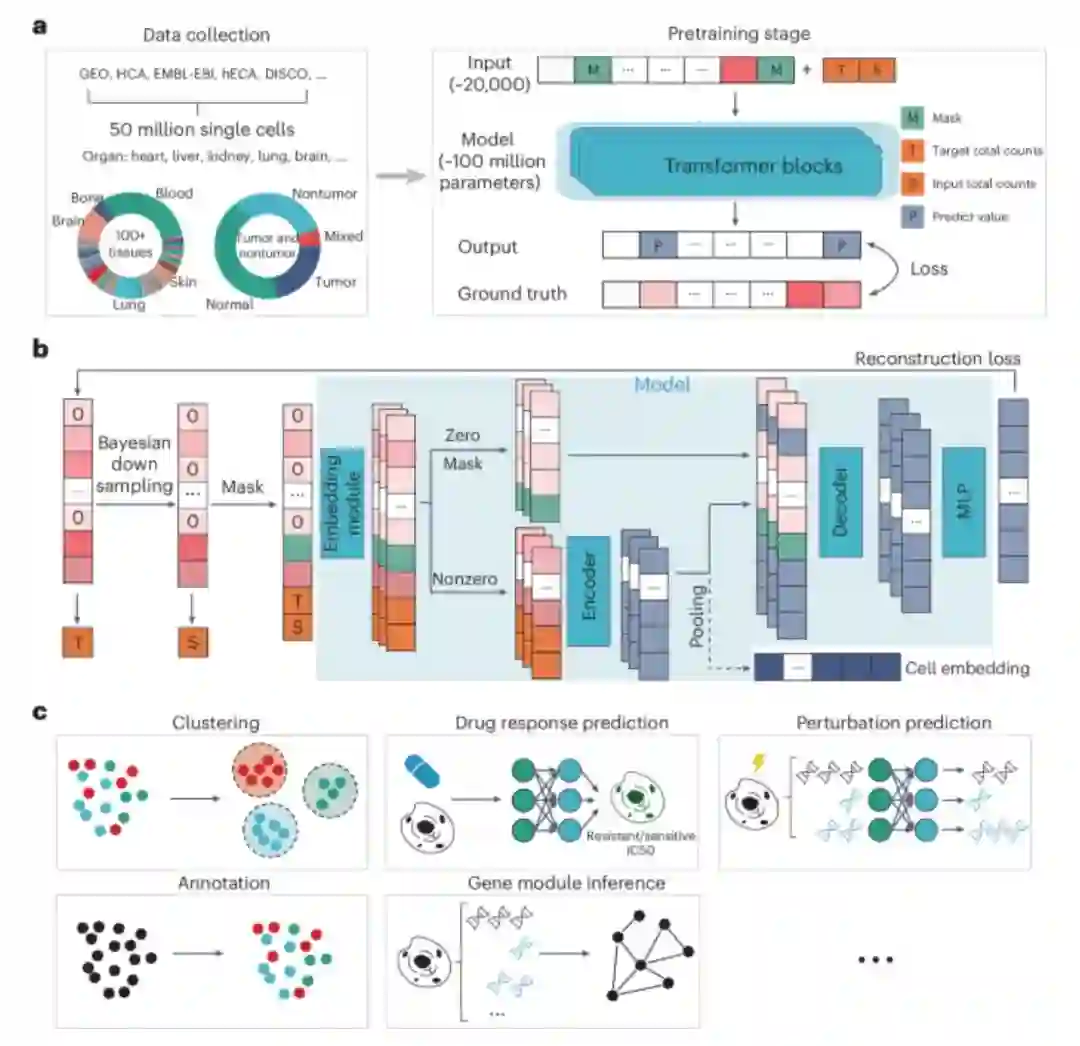
遵循类似的方式,来自多伦多大学的Bo Wang团队,以及Jianzhu Ma、Xuegong Zhang和Le Song团队,分别介绍了两个单细胞基础模型(scGPT[3]和scFoundation[4]),并展示了它们在细胞类型注释、扰动预测和其他任务中的能力。在另一篇研究论文中,Wenpin Hou和Zhicheng Ji指出,GPT-4在使用单细胞RNA测序数据注释细胞类型方面可以实现最先进的性能[5]。Mohammad Lotfollahi的新闻稿[6]系统总结了并比较了这些工作,并讨论了这个领域的未来方向。
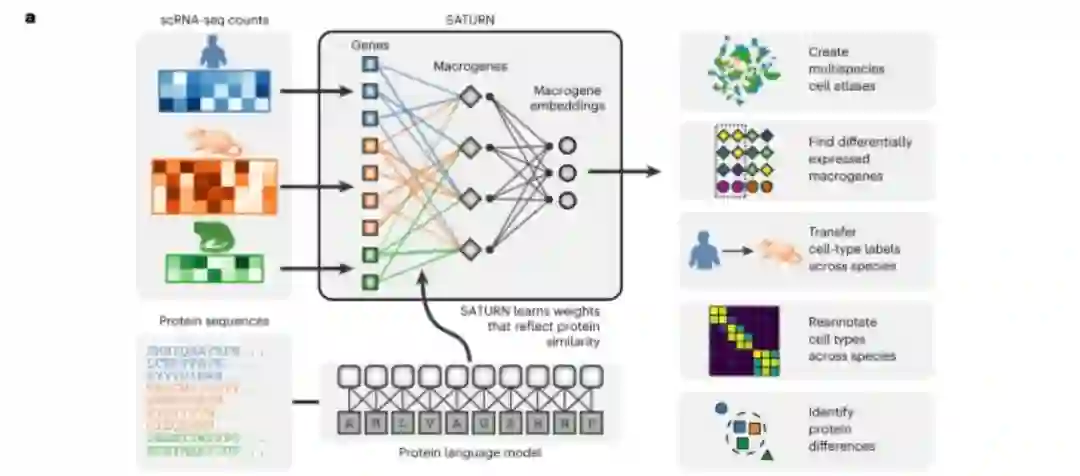
同样在这期专刊中,Jure Leskovec和他的团队整合了来自不同物种的单细胞RNA测序数据集,通过使用大型蛋白质语言模型,从而学习到蛋白质低维表示,这朝着构建通用细胞低维表示迈出了一步[7]。此外,Lior Pachter和他的同事们提出了一个基于变分自编码器的框架,用于模拟转录和剪接动力学过程[8]。
机器学习方法在基因组学中产生影响的另一个领域是将序列映射到表型,Alexander Sasse、Maria Chikina和Sara Mostafavi在评论文章中讨论了这一点[9]。通过利用从许多不同的细胞类型和条件下收集的多模态数据,这些从序列映射到功能的模型旨在揭示不同遗传和环境因素影响下各种分子层面相互作用和调控的机制。
空间转录组学是一个蓬勃发展的领域(我们在2020年年度方法中强调了这一点)。这些技术使得收集大量高维数据成为可能;因此,深度学习中的方法对于挖掘这些复杂且信息丰富的数据集至关重要,并且将继续如此。在一篇研究论文中,Zhi Wei和他的团队为空间组学分析开发了空间感知的深度生成模型[10]。在评论文章中,Mingyao Li和团队讨论了AI在空间组学的现状和未来[11]。
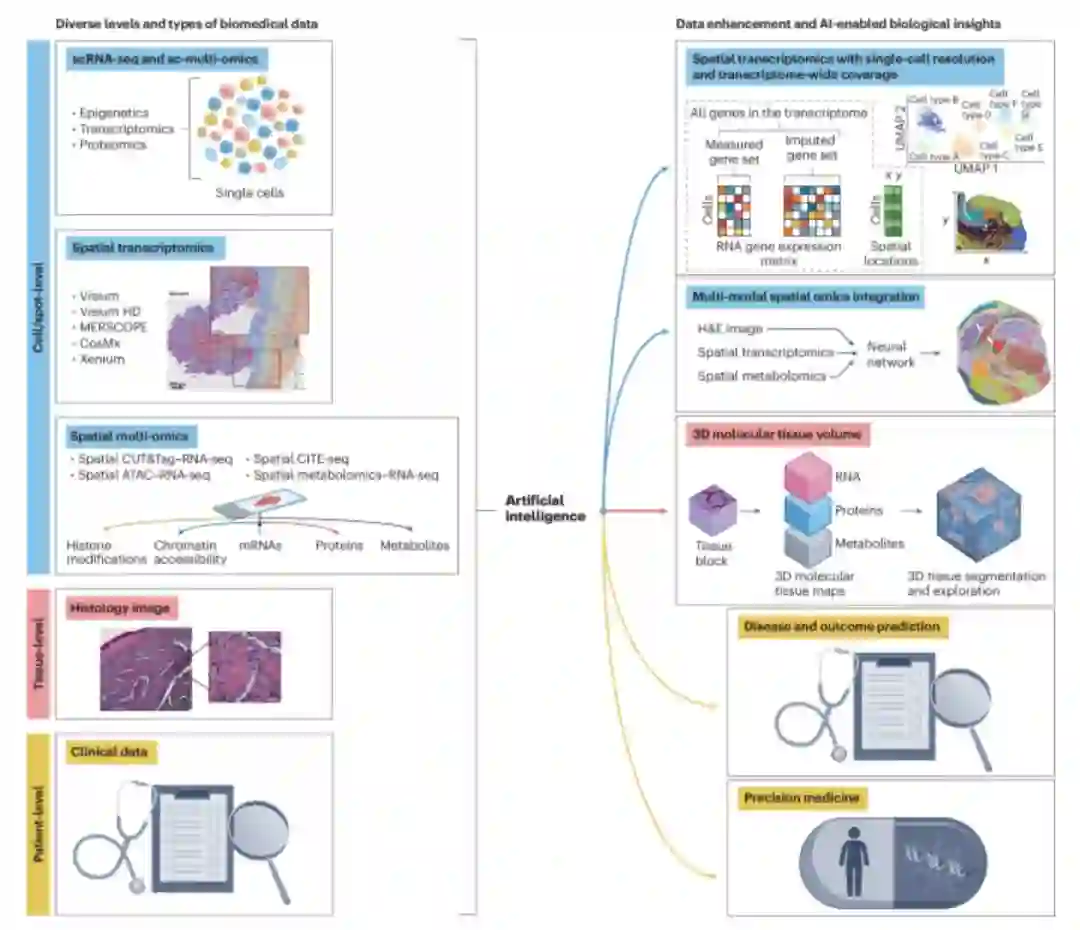
AI应用在蛋白领域(如蛋白结构预测、蛋白质工程、蛋白组学)的经典例子
运用计算方法研究蛋白质已经有很长一段时间历史了。预测生物分子相互作用的方法,如AlphaFold3和RoseTTAFold-AllAtom,已经取得了巨大的进步。然而,仍然存在些许挑战。Minkyung Baek的评论强调了为了捕捉这些相互作用的生物物理复杂性仍然需要做出的努力[12]。
在这个专刊中,我们还介绍了由Mohammed AlQuraishi、Nazim Bouatta等团队开发的OpenFold[13],这个工作展示了完全开源的AlphaFold2实现方式。基于深度学习的方法,如Barrett Powell和Joseph Davis开发的的TomoDRGN[14],以及Ellen Zhong、Abhay Kotecha团队开发的CryoDRGN-ET[15],也被用来模拟来自冷冻电子扫描显微镜数据集的连续构象和组成异质性。
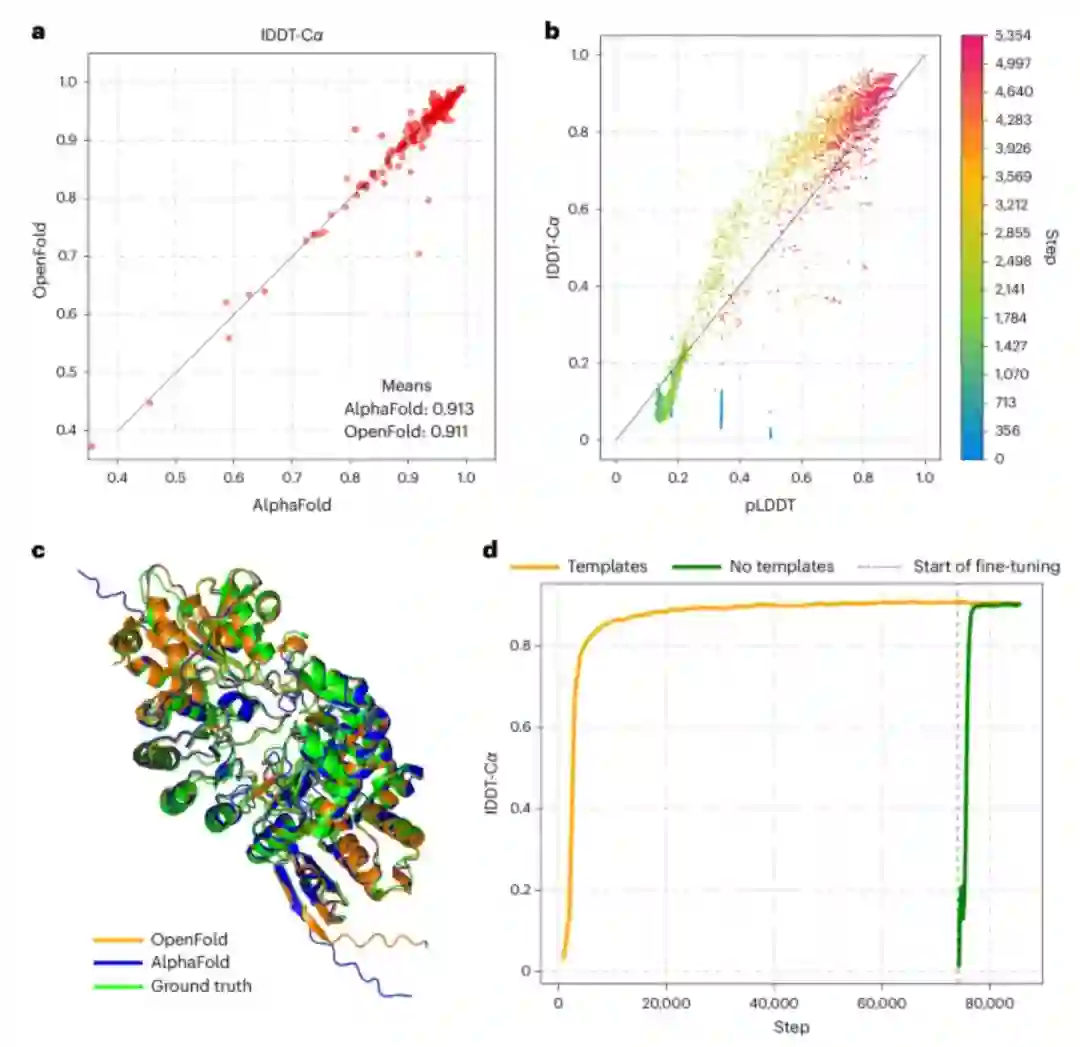
Omar Abudayyeh和Jonathan Gootenberg的评论文章强调了AI如何改变分子和细胞工程[16]。AI有助于理解不同的蛋白质系统,并发现如CRISPR等蛋白质工具。基于AI的蛋白质工程也在提高CRISPR编辑效率,并增强我们扰动细胞的能力,甚至可以构建“虚拟细胞”。
此外,高级AI方法也将塑造蛋白质组学领域:在他们的评论中,Benjamin Gyori和Olga Vitek描述了AI方法将如何帮助研究人员利用来自零散来源的知识,推进基于质谱的蛋白质组学数据分析,进而完成机制的发现和功能的解释[17]。
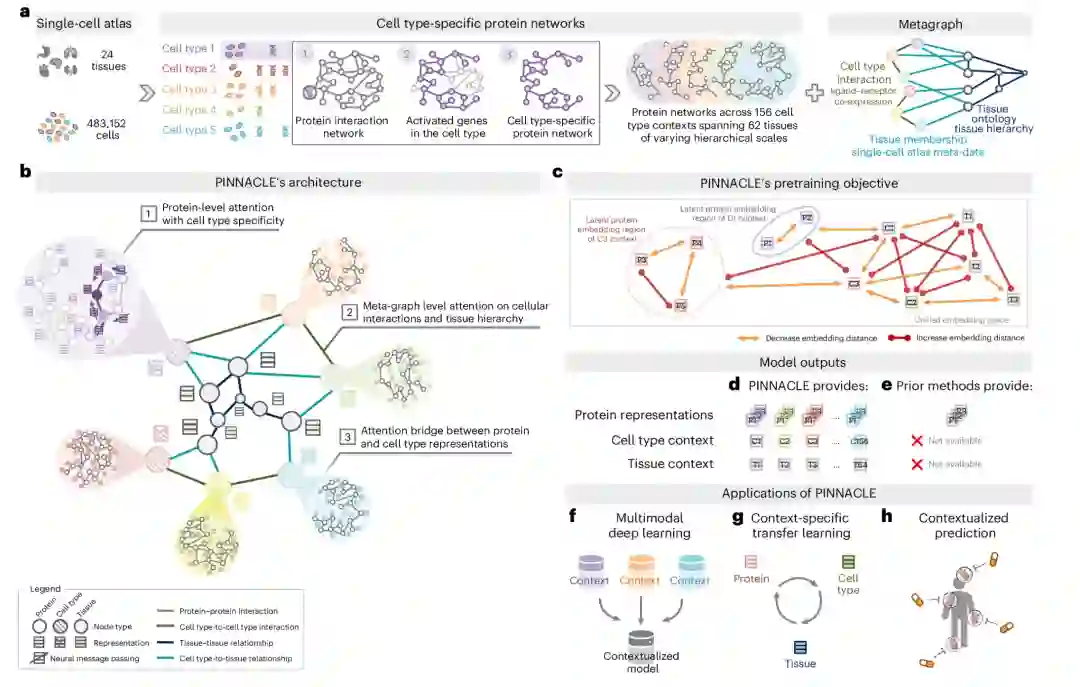
专刊中也介绍了由Marinka Zitnik和其团队开发的PINNACLE框架[18],这是一个AI模型,它在考虑生物学背景信息的情况下学习蛋白质表示。在生成细胞类型特异的蛋白质表示时,模型考虑了多种生物背景信息,如单细胞转录组数据、蛋白质-蛋白质相互作用网络、细胞类型到细胞类型的相互作用和组织层次等。相信这个模型将会为整个领域提供一定的方向指引。
PINNACLE在考虑生物学背景信息的情况下学习蛋白质表示https://www.nature.com/articles/s41592-024-02341-3
AI应用在成像领域的经典例子
随着计算机视觉的发展,显微成像领域也已经取得了长足的进步。但在许多方面,这种跨学科工作仍处于起步阶段。Shanghang Zhang、Jianxu Chen团队的评论文章讨论了这个方向的未来趋势[19]。在文中,他们指出,数据量更大、参数更多的深度学习模型往往在效果上更好,以及大型多模态模型可能在生物成像方面提供巨大突破,甚至有潜力超越人类的识图能力。Bo Yan和他的同事们的研究论文表明,一个通用的基础模型可以被训练来执行荧光显微镜图像重建任务,这突出了一点:更大模型能够处理更多训练数据,并能够完成多样化的下游任务,如去噪、各向同性重建和跨模态图像生成[20]。

本期两篇通讯(Correspondence)文章介绍了利用大型语言模型进行生物图像分析的工具。Wei Ouyang和他的团队通过他们的BioImage.IO聊天机器人,将大型语言模型的便捷性直接呈现在分析图像的生物学家眼前[21],该聊天机器人使用户能够广泛汲取社区范围的知识,并通过撰写提示词轻松地完成复杂的生物图像分析任务。Loïc Royer介绍了Omega,这是一个基于大型语言模型的对话智能体[22],可以辅助从设计实验到实施生物图像分析整个过程。这些工具无疑将帮助具有不同专业知识的图像分析师方便快捷地完成任务。
AI应用在建模复杂生物系统的经典例子
AI可以在研究复杂生物系统领域大展身手,一个最显著的例子是大脑的连接组重建。无论是来自果蝇、小鼠还是人类的数据,重建大脑都需要在大规模电子显微镜数据集中详细准确地分割神经元及其突起。在他们的评论中,Michał Januszewski和Viren Jain讨论了基础模型在解决与连接组学相关的计算挑战方面的潜力[23]。
另一个值得注意的例子是,如何解析免疫系统的复杂性和异质性。AI的最新进展可能使研究人员能够理解人类免疫系统的局限性。在他们的评论中,Eloise Berson、Thomas Montine、Nima Aghaeepour和团队成员讨论了AI方法在推进免疫学研究以及探索这个领域尚未解决的挑战方面起到的作用[24]。在癌症研究中,Elham Azizi和他的同事们讨论了AI在促进新发现方面的作用。新的机器学习模型有望解决关键问题,如整合具有异质性的数据、量化和建模细胞,以及在肿瘤发生、转移和失调的背景下识别因果调控网络[25]。
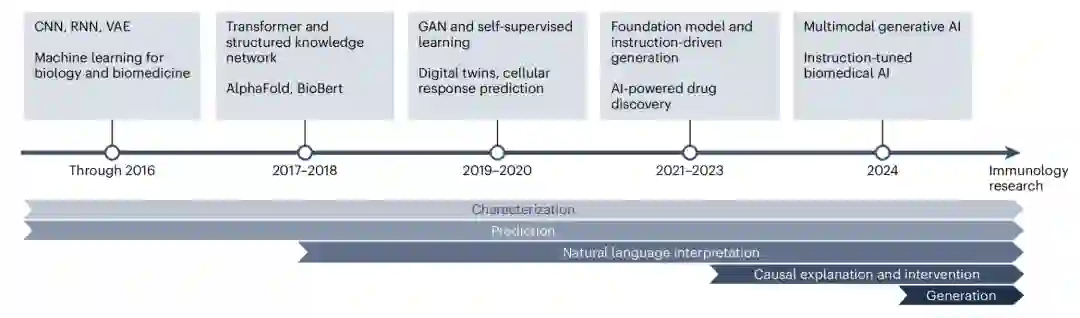
探讨人工智能的经典问题(如数据泄露问题、伦理问题、可解释性问题等)
尽管基于AI的方法在生物学中取得了值得称赞的成就,但挑战依然存在,其中一些是特定领域的,另一些是普遍的。
生物数据通常嘈杂且有偏见,并且在质量和数量上具有高水平的异质性。在许多情况下,很难知道真相,即使是手动注释也不是无误的。正如David Blumenthal、Dominik Grimm、Olga Kalinina、Markus List和同事们的观点文章指出,这些挑战可能会限制AI模型的准确性和泛化能力。此外,生物数据集之间的复杂依赖关系也可能导致数据泄露,该文章还讨论了机器学习模型中这种泄露的来源。研究者们提出了一些注意点,可以帮助识别模型是否出现了数据泄露,并避免由数据泄露导致的问题[26]。
AI for Biology不仅是使用生物学数据完成分类或预测任务,更重要的,生物学家渴望使用AI从他们的数据中学习生物学知识,并指导他们设计新的实验和转化策略。因此,许多机器学习方法的黑箱属性经常成为一个主要障碍,这使得可解释的机器学习成为一个有吸引力的替代方案。在他们的观点中,Ameet Talwalkar、Jian Ma和同事们回顾了使用可解释机器学习的方法和建议,以及在大型语言模型时代新发展的机遇和陷阱[27]。在另一篇评论中,Oded Rotem和Assaf Zaritsky讨论了在生物成像中可解释和可解释AI的重要性,以及如何通过理解黑箱来引导图像分析中的新生物学发现[28]。
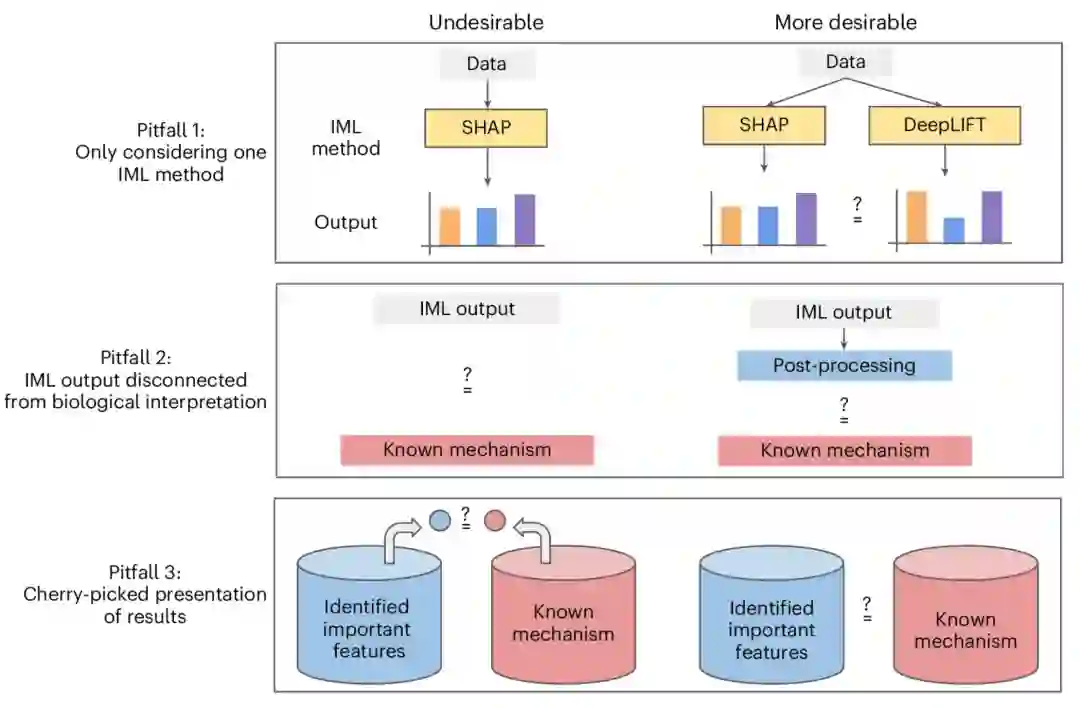
值得庆幸的是,**与AI潜在危害相关的伦理方面问题正在得到越来越多关注和重视。**在评论中,伦理学家Carina Prunkl讨论了使用AI进行科学研究的伦理含义,并强调关键的风险缓解策略将取决于是否能够完成有效的教育和高效的管制[29]。利用AI进行自我教育这一需求确实变得日益清晰。美国国家科学基金会主任Sethuraman Panchanathan将利用AI进行教育视为素养,正如他在本月的技术特辑(Technology Feature)“追求AI素养”中与Vivien Marx分享的一样,**科学家们正在寻求更好的培训和教育机会,以便在使用和构建AI工具的过程中提升素养。**我们欢迎整个研究社区就这些重要话题进行更多的讨论和采取行动[30]。
作为编辑,我们正在积极探索如何持续改进,以发表更多高质量基于AI的方法论文。我们已经发布了由社区开发的机器学习报告指南,并制定了详细的政策,通过要求共享数据模型和代码,从而实现全过程透明和结果可复现。随着AI与生物学之间更紧密的交叉合作,我们相信不久将再次回到这个激动人心的话题。
参考文献
Simon, E. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02354-y (2024). 1. Szalata, A. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02353-z (2024). 1. Cui, H. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02201-0 (2024). 1. Hao, M. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02305-7 (2024). 1. Hou, W. & Ji, Z. Nat. Methods https://doi.org/10.1038/s41592-024-02235-4 (2024). 1. Lotfollahi, M. Nat. Methods https://doi.org/10.1038/s41592-024-02367-7 (2024). 1. Rosen, Y. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02191-z (2024). 1. Carilli, M., Gorin, G., Choi, Y., Chari, T. & Pachter, L. Nat.Methods https://doi.org/10.1038/s41592-024-02365-9(2024). 1. Sasse, A. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02331-5 (2024). 1. Tian, T., Zhang, J., Lin, X., Wei, Z. & Hakonarson, H. Nat.Methods https://doi.org/10.1038/s41592-024-02257-y(2024). 1. Coleman, K. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02363-x (2024). 1. Baek, M. Nat. Methods https://doi.org/10.1038/s41592-024-02350-2 (2024). 1. Ahdritz, G. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02272-z (2024). 1. Powell, B. M. & Davis, J. H. Nat. Methods https://doi.org/10.1038/s41592-024-02210-z (2024). 1. Rangan, R. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02340-4 (2024). 1. Abudayyeh, O. O. & Gootenberg, J. S. Nat. Methodshttps://doi.org/10.1038/s41592-024-02338-y (2024). 1. Gyori, B. M. & Vitek, O. Nat. Methods https://doi.org/10.1038/s41592-024-02324-4 (2024). 1. Li, M. M. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02341-3 (2024). 1. Zhang, S. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02334-2 (2024). 1. Ma, C., Tan, W., He, R. & Yan, B. Nat. Methods https://doi.org/10.1038/s41592-024-02244-3 (2024). 1. Lei, W. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02370-y (2024). 1. Royer, L. A. Nat. Methods https://doi.org/10.1038/s41592-024-02310-w (2024). 1. Januszewski, M. & Jain, V. Nat. Methods https://doi.org/10.1038/s41592-024-02336-0 (2024). 1. Berson, E. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02351-1 (2024). 1. Fan, J. L. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02364-w (2024). 1. Bernett, J. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02362-y (2024). 1. Chen, V. et al. Nat. Methods https://doi.org/10.1038/s41592-024-02359-7 (2024). 1. Rotem, O. & Zaritsky, A. Nat. Methods https://doi.org/10.1038/s41592-024-02322-6 (2024). 1. Prunkl, C. Nat. Methods https://doi.org/10.1038/s41592-024-02332-4 (2024). 1. Marx, V. Nat. Methods https://doi.org/10.1038/s41592-024-02369-5 (2024).
参考文献可上下滑动查看