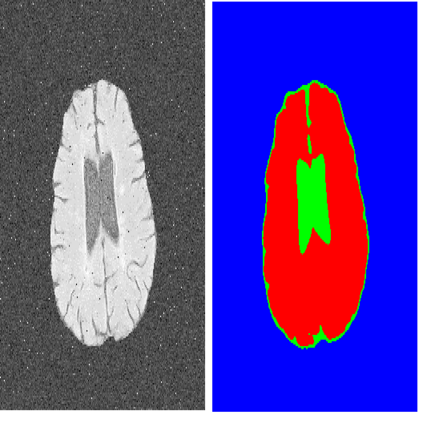
The U-Net architecture, built upon the fully convolutional network, has proven to be effective in biomedical image segmentation. However, U-Net applies skip connections to merge semantically different low- and high-level convolutional features, resulting in not only blurred feature maps, but also over- and under-segmented target regions. To address these limitations, we propose a simple, yet effective end-to-end depthwise encoder-decoder fully convolutional network architecture, called Sharp U-Net, for binary and multi-class biomedical image segmentation. The key rationale of Sharp U-Net is that instead of applying a plain skip connection, a depthwise convolution of the encoder feature map with a sharpening kernel filter is employed prior to merging the encoder and decoder features, thereby producing a sharpened intermediate feature map of the same size as the encoder map. Using this sharpening filter layer, we are able to not only fuse semantically less dissimilar features, but also to smooth out artifacts throughout the network layers during the early stages of training. Our extensive experiments on six datasets show that the proposed Sharp U-Net model consistently outperforms or matches the recent state-of-the-art baselines in both binary and multi-class segmentation tasks, while adding no extra learnable parameters. Furthermore, Sharp U-Net outperforms baselines that have more than three times the number of learnable parameters.
翻译:U-Net 架构建在完全连通的网络上, 已证明在生物医学图像分割方面是有效的。 然而, U-Net 将连接跳过连接, 以合并精密的低层和高层共生特征, 不仅导致地貌图模糊, 而且还造成分解过度和分解不足的目标区域。 为了解决这些局限性, 我们提出了一个简单而有效的端到端的深度深层次编码解码器- 完全连动网络架构, 叫做 Sharp U- Net, 用于二进制和多级生物医学图像分割。 Sharp U- Net 的关键原理是, 而不是应用平坦跳连接连接, 而是使用精锐的内核过滤器对编码图进行深度连接, 在合并编码器和分解码特性之前, 不仅使用了精细的内核过滤器, 而且还使用了精细的中间特征图, 与编码器图的大小相同。 使用这个精锐的过滤层, 我们不仅能够将精密的内脏的内分解性地拼, 而且还可以在整个网络结构中平整层进行训练阶段, 。 我们的精细的基化了六级的实验显示的底线系,,, 在最新的基底线条列中,, 在不断的基系中, 的细线条列中, 显示,,, 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的 的