时间序列深度学习:状态 LSTM 模型预测太阳黑子(中)
作者:徐瑞龙 整理分享量化投资与固定收益相关的文章
博客专栏:
https://www.cnblogs.com/xuruilong100
5 用 Keras 构建状态 LSTM 模型
首先,我们将在回测策略的某个样本上用 Keras 开发一个状态 LSTM 模型。然后,我们将模型套用到所有样本,以测试和验证模型性能。
5.1 单个 LSTM 模型
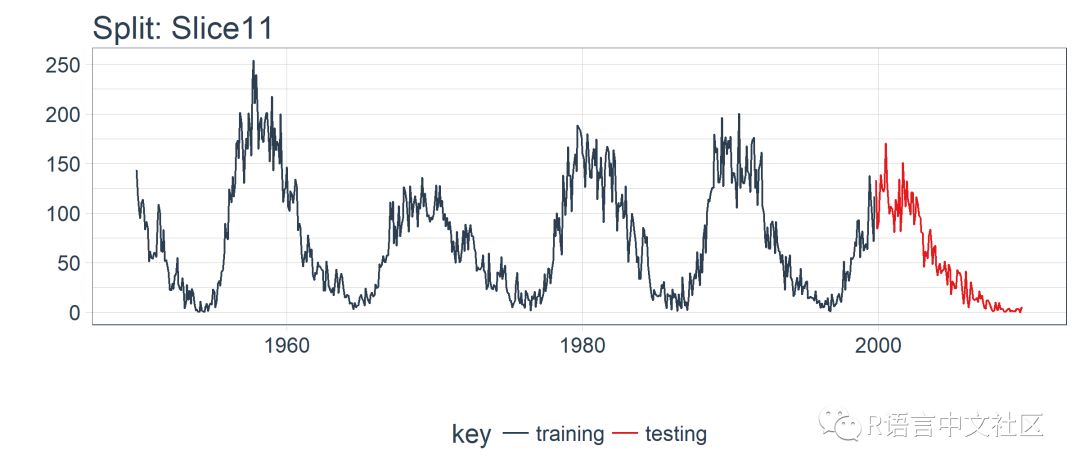
对单个 LSTM 模型,我们选择并可视化最近一期的分割样本(Slice11),这一样本包含了最新的数据。
split <- rolling_origin_resamples$splits[[11]] split_id <- rolling_origin_resamples$id[[11]]
5.1.1 可视化该分割样本
我么可以用 plot_split() 函数可视化该分割,设定 expand_y_axis = FALSE 以便将横坐标缩放到样本本身的范围。
plot_split( split,
expand_y_axis = FALSE,
size = 0.5) +
theme(legend.position = "bottom") +
ggtitle(glue("Split: {split_id}"))
5.1.2 数据准备
首先,我们将训练和测试数据集合成一个数据集,并使用列 key 来标记它们来自哪个集合(training 或 testing)。请注意,tbl_time 对象需要在调用 bind_rows() 时重新指定索引,但是这个问题应该很快在 dplyr 包中得到纠正。
df_trn <- training(split) df_tst <- testing(split) df <- bind_rows( df_trn %>% add_column(key = "training"), df_tst %>% add_column(key = "testing")) %>%
as_tbl_time(index = index) df
## # A time tibble: 720 x 3
## # Index: index
## index value key
## <date> <dbl> <chr>
## 1 1949-11-01 144. training
## 2 1949-12-01 118. training
## 3 1950-01-01 102. training
## 4 1950-02-01 94.8 training
## 5 1950-03-01 110. training
## 6 1950-04-01 113. training
## 7 1950-05-01 106. training
## 8 1950-06-01 83.6 training
## 9 1950-07-01 91.0 training
## 10 1950-08-01 85.2 training
## # ... with 710 more rows
5.1.3 用 recipe 做数据预处理
LSTM 算法要求输入数据经过中心化并标度化。我们可以使用 recipe 包预处理数据。我们用 step_sqrt 来转换数据以减少异常值的影响,再结合 step_center 和 step_scale 对数据进行中心化和标度化。最后,数据使用 bake() 函数实现处理转换。
rec_obj <- recipe(value ~ ., df) %>%
step_sqrt(value) %>%
step_center(value) %>%
step_scale(value) %>%
prep() df_processed_tbl <- bake(rec_obj, df) df_processed_tbl
## # A tibble: 720 x 3
## index value key
## <date> <dbl> <fct>
## 1 1949-11-01 1.25 training
## 2 1949-12-01 0.929 training
## 3 1950-01-01 0.714 training
## 4 1950-02-01 0.617 training
## 5 1950-03-01 0.825 training
## 6 1950-04-01 0.874 training
## 7 1950-05-01 0.777 training
## 8 1950-06-01 0.450 training
## 9 1950-07-01 0.561 training
## 10 1950-08-01 0.474 training
## # ... with 710 more rows
接着,记录中心化和标度化的信息,以便在建模完成之后可以将数据逆向转换回去。平方根转换可以通过乘方运算逆转回去,但要在逆转中心化和标度化之后。
center_history <- rec_obj$steps[[2]]$means["value"] scale_history <- rec_obj$steps[[3]]$sds["value"]
c("center" = center_history, "scale" = scale_history)
## center.value scale.value
## 7.549526 3.545561
5.1.4 规划 LSTM 模型
我们需要规划下如何构建 LSTM 模型。首先,了解几个 LSTM 模型的专业术语:
张量格式(Tensor Format):
预测变量(X)必须是一个 3 维数组,维度分别是:
samples、timesteps和features。第一维代表变量的长度;第二维是时间步(滞后阶数);第三维是预测变量的个数(1 表示单变量,n 表示多变量)输出或目标变量(y)必须是一个 2 维数组,维度分别是:
samples和timesteps。第一维代表变量的长度;第二维是时间步(之后阶数)
训练与测试:
训练与测试的长度必须是可分的(训练集长度除以测试集长度必须是一个整数)
批量大小(Batch Size):
批量大小是在 RNN 权重更新之前一次前向 / 后向传播过程中训练样本的数量
批量大小关于训练集和测试集长度必须是可分的(训练集长度除以批量大小,以及测试集长度除以批量大小必须是一个整数)
时间步(Time Steps):
时间步是训练集与测试集中的滞后阶数
我们的例子中滞后 1 阶
周期(Epochs):
周期是前向 / 后向传播迭代的总次数
通常情况下周期越多,模型表现越好,直到验证集上的精确度或损失不再增加,这时便出现过度拟合
考虑到这一点,我们可以提出一个计划。我们将预测窗口或测试集的长度定在 120 个月(10年)。最优相关性发生在 125 阶,但这并不能被预测范围整除。我们可以增加预测范围,但是这仅提供了自相关性的最小幅度增加。我们选择批量大小为 40,它可以整除测试集和训练集的观察个数。我们选择时间步等于 1,这是因为我们只使用 1 阶滞后(只向前预测一步)。最后,我们设置 epochs = 300,但这需要调整以平衡偏差与方差。
# Model inputs
lag_setting <- 120 # = nrow(df_tst)
batch_size <- 40
train_length <- 440
tsteps <- 1
epochs <- 300
5.1.5 2 维与 3 维的训练、测试数组
下面将训练集和测试集数据转换成合适的形式(数组)。记住,LSTM 模型要求预测变量(X)是 3 维的,输出或目标变量(y)是 2 维的。
# Training Set
lag_train_tbl <- df_processed_tbl %>%
mutate(value_lag = lag(value, n = lag_setting)) %>%
filter(!is.na(value_lag)) %>%
filter(key =="training") %>%
tail(train_length) x_train_vec <- lag_train_tbl$value_lag x_train_arr <- array(
data = x_train_vec, dim = c(length(x_train_vec), 1, 1)) y_train_vec <- lag_train_tbl$value y_train_arr <- array(
data = y_train_vec, dim = c(length(y_train_vec), 1))
# Testing Set
lag_test_tbl <- df_processed_tbl %>%
mutate(
value_lag = lag( value, n = lag_setting)) %>%
filter(!is.na(value_lag)) %>%
filter(key =="testing") x_test_vec <- lag_test_tbl$value_lag x_test_arr <- array(
data = x_test_vec,
dim = c(length(x_test_vec), 1, 1)) y_test_vec <- lag_test_tbl$value y_test_arr <- array(
data = y_test_vec,
dim = c(length(y_test_vec), 1))
5.1.6 构建 LSTM 模型
我们可以使用 keras_model_sequential() 构建 LSTM 模型,并像堆砖块一样堆叠神经网络层。我们将使用两个 LSTM 层,每层都设定 units = 50。第一个 LSTM 层接收所需的输入形状,即[时间步,特征数量]。批量大小就是我们的批量大小。我们将第一层设置为 return_sequences = TRUE 和 stateful = TRUE。第二层和前面相同,除了 batch_size(batch_size只需要在第一层中指定),另外 return_sequences = FALSE 不返回时间戳维度(从第一个 LSTM 层返回 2 维数组,而不是 3 维)。我们使用 layer_dense(units = 1),这是 Keras 序列模型的标准结尾。最后,我们在 compile() 中使用 loss ="mae" 以及流行的 optimizer = "adam"。
model <- keras_model_sequential()
model %>%
layer_lstm(
units = 50,
input_shape = c(tsteps, 1),
batch_size = batch_size,
return_sequences = TRUE,
stateful = TRUE) %>%
layer_lstm(
units = 50,
return_sequences = FALSE,
stateful = TRUE) %>%
layer_dense(units = 1) model %>%
compile(loss = 'mae', optimizer = 'adam') model
## Model
## ______________________________________________________________________
## Layer (type) Output Shape Param #
## ======================================================================
## lstm_1 (LSTM) (40, 1, 50) 10400
## ______________________________________________________________________
## lstm_2 (LSTM) (40, 50) 20200
## ______________________________________________________________________
## dense_1 (Dense) (40, 1) 51
## ======================================================================
## Total params: 30,651
## Trainable params: 30,651
## Non-trainable params: 0
## ______________________________________________________________________
5.1.7 拟合 LSTM 模型
下一步,我们使用一个 for 循环拟合状态 LSTM 模型(需要手动重置状态)。有 300 个周期要循环,运行需要一点时间。我们设置 shuffle = FALSE 来保存序列,并且我们使用 reset_states() 在每个循环后手动重置状态。
for (i in 1:epochs) { model %>%
fit(x = x_train_arr, y = y_train_arr, batch_size = batch_size,
epochs = 1, verbose = 1, shuffle = FALSE) model %>% reset_states()
cat("Epoch: ", i) }
5.1.8 使用 LSTM 模型预测
然后,我们可以使用 predict() 函数对测试集 x_test_arr 进行预测。我们可以使用之前保存的 scale_history 和 center_history 转换得到的预测,然后对结果进行平方。最后,我们使用 reduce() 和自定义的 time_bind_rows() 函数将预测与一列原始数据结合起来。
# Make Predictions
pred_out <- model %>%
predict(x_test_arr, batch_size = batch_size) %>% .[,1] # Retransform values
pred_tbl <- tibble(
index = lag_test_tbl$index,
value = (pred_out * scale_history + center_history)^2) # Combine actual data with predictions
tbl_1 <- df_trn %>%
add_column(key = "actual") tbl_2 <- df_tst %>%
add_column(key = "actual") tbl_3 <- pred_tbl %>%
add_column(key = "predict")
# Create time_bind_rows() to solve dplyr issue
time_bind_rows <- function(data_1, data_2, index) { index_expr <- enquo(index)
bind_rows(data_1, data_2) %>%
as_tbl_time(index = !! index_expr) } ret <- list(tbl_1, tbl_2, tbl_3) %>%
reduce(time_bind_rows, index = index) %>%
arrange(key, index) %>%
mutate(key = as_factor(key)) ret
## # A time tibble: 840 x 3
## # Index: index
## index value key
## <date> <dbl> <fct>
## 1 1949-11-01 144. actual
## 2 1949-12-01 118. actual
## 3 1950-01-01 102. actual
## 4 1950-02-01 94.8 actual
## 5 1950-03-01 110. actual
## 6 1950-04-01 113. actual
## 7 1950-05-01 106. actual
## 8 1950-06-01 83.6 actual
## 9 1950-07-01 91.0 actual
## 10 1950-08-01 85.2 actual
## # ... with 830 more rows
5.1.9 评估单个分割样本上 LSTM 模型的表现
我们使用 yardstick 包里的 rmse() 函数评估表现,rmse() 返回均方误差平方根(RMSE)。我们的数据以“长”格式的形式存在(使用 ggplot2 可视化的最佳格式),所以需要创建一个包装器函数 calc_rmse() 对数据做预处理,以适应 yardstick::rmse() 的要求。
calc_rmse <- function(prediction_tbl) { rmse_calculation <- function(data) { data %>%
spread(key = key, value = value) %>%
select(-index) %>%
filter(!is.na(predict)) %>%
rename(
truth = actual,
estimate = predict) %>%
rmse(truth, estimate) } safe_rmse <- possibly( rmse_calculation, otherwise = NA)
safe_rmse(prediction_tbl) }
我们计算模型的 RMSE。
calc_rmse(ret)
## [1] 31.81798
RMSE 提供的信息有限,我们需要可视化。注意:当我们扩展到回测策略中的所有样本时,RMSE 将在确定预期误差时派上用场。
5.1.10 可视化一步预测
下一步,我们创建一个绘图函数——plot_prediction(),借助 ggplot2 可视化单一样本上的结果。
# Setup single plot function
plot_prediction <- function(data,id,
alpha = 1,
size = 2,
base_size = 14) {rmse_val <- calc_rmse(data)
g <- data %>%
ggplot(aes(index, value, color = key)) +
geom_point(alpha = alpha, size = size) +
theme_tq(base_size = base_size) +
scale_color_tq() +
theme(legend.position = "none") +
labs(
title = glue(
"{id}, RMSE: {round(rmse_val, digits = 1)}"),
x = "", y = "")return(g) }
我们设置 id = split_id,在 Slice11 上测试函数。
ret %>%
plot_prediction(id = split_id, alpha = 0.65) +
theme(legend.position = "bottom")
LSTM 模型表现相对较好! 我们选择的设置似乎产生了一个不错的模型,可以捕捉到数据中的趋势。预测在下一个上升趋势前抢跑了,但总体上好过了我的预期。现在,我们需要通过回测来查看随着时间推移的真实表现!
5.2 在 11 个样本上回测 LSTM 模型
一旦我们有了能在一个样本上工作的 LSTM 模型,扩展到全部 11 个样本上就相对简单。我们只需创建一个预测函数,再套用到 rolling_origin_resamples 中抽样计划包含的数据上。
5.2.1 构建一个 LSTM 预测函数
这一步看起来很吓人,但实际上很简单。我们将 5.1 节的代码复制到一个函数中。我们将它作为一个安全函数,对于任何长时间运行的函数来说,这是一个很好的做法,可以防止单个故障停止整个过程。
predict_keras_lstm <- function(split,
epochs = 300,
...) { lstm_prediction <- function(split, epochs,
...) { # 5.1.2 Data Setup df_trn <- training(split) df_tst <- testing(split) df <- bind_rows( df_trn %>% add_column(key = "training"), df_tst %>% add_column(key = "testing")) %>%
as_tbl_time(index = index)
# 5.1.3 Preprocessing rec_obj <- recipe(value ~ ., df) %>%
step_sqrt(value) %>%
step_center(value) %>%
step_scale(value) %>%
prep() df_processed_tbl <- bake(rec_obj, df) center_history <- rec_obj$steps[[2]]$means["value"] scale_history <- rec_obj$steps[[3]]$sds["value"]
# 5.1.4 LSTM Plan lag_setting <- 120 # = nrow(df_tst) batch_size <- 40 train_length <- 440 tsteps <- 1 epochs <- epochs
# 5.1.5 Train/Test Setup lag_train_tbl <- df_processed_tbl %>% mutate( value_lag = lag(value, n = lag_setting)) %>% filter(!is.na(value_lag)) %>% filter(key =="training") %>% tail(train_length) x_train_vec <- lag_train_tbl$value_lag x_train_arr <- array( data = x_train_vec, dim = c(length(x_train_vec), 1, 1)) y_train_vec <- lag_train_tbl$value y_train_arr <- array( data = y_train_vec, dim = c(length(y_train_vec), 1)) lag_test_tbl <- df_processed_tbl %>% mutate( value_lag = lag(value, n = lag_setting)) %>% filter(!is.na(value_lag)) %>% filter(key =="testing") x_test_vec <- lag_test_tbl$value_lag x_test_arr <- array( data = x_test_vec, dim = c(length(x_test_vec), 1, 1)) y_test_vec <- lag_test_tbl$value y_test_arr <- array( data = y_test_vec, dim = c(length(y_test_vec), 1))
# 5.1.6 LSTM Model model <- keras_model_sequential() model %>% layer_lstm( units = 50, input_shape = c(tsteps, 1), batch_size = batch_size, return_sequences = TRUE, stateful = TRUE) %>% layer_lstm( units = 50, return_sequences = FALSE, stateful = TRUE) %>% layer_dense(units = 1) model %>% compile(loss = 'mae', optimizer = 'adam')
# 5.1.7 Fitting LSTM for (i in 1:epochs) { model %>% fit(x = x_train_arr, y = y_train_arr, batch_size = batch_size, epochs = 1, verbose = 1, shuffle = FALSE) model %>% reset_states() cat("Epoch: ", i) }
# 5.1.8 Predict and Return Tidy Data # Make Predictions pred_out <- model %>% predict(x_test_arr, batch_size = batch_size) %>% .[,1] # Retransform values pred_tbl <- tibble( index = lag_test_tbl$index, value = (pred_out * scale_history + center_history)^2) # Combine actual data with predictions tbl_1 <- df_trn %>% add_column(key = "actual") tbl_2 <- df_tst %>% add_column(key = "actual") tbl_3 <- pred_tbl %>% add_column(key = "predict")
# Create time_bind_rows() to solve dplyr issue time_bind_rows <- function(data_1, data_2, index) { index_expr <- enquo(index) bind_rows(data_1, data_2) %>% as_tbl_time(index = !! index_expr) } ret <- list(tbl_1, tbl_2, tbl_3) %>% reduce(time_bind_rows, index = index) %>% arrange(key, index) %>% mutate(key = as_factor(key))
return(ret) } safe_lstm <- possibly(lstm_prediction, otherwise = NA) safe_lstm(split, epochs, ...) }
我们测试下 predict_keras_lstm() 函数,设置 epochs = 10。返回的数据为长格式,在 key 列中标记有 actual 和 predict。
predict_keras_lstm(split, epochs = 10)
## # A time tibble: 840 x 3
## # Index: index
## index value key
## <date> <dbl> <fct>
## 1 1949-11-01 144. actual
## 2 1949-12-01 118. actual
## 3 1950-01-01 102. actual
## 4 1950-02-01 94.8 actual
## 5 1950-03-01 110. actual
## 6 1950-04-01 113. actual
## 7 1950-05-01 106. actual
## 8 1950-06-01 83.6 actual
## 9 1950-07-01 91.0 actual
## 10 1950-08-01 85.2 actual
## # ... with 830 more rows
5.2.2 将 LSTM 预测函数应用到 11 个样本上
既然 predict_keras_lstm() 函数可以在一个样本上运行,我们现在可以借助使用 mutate() 和 map() 将函数应用到所有样本上。预测将存储在名为 predict 的列中。注意,这可能需要 5-10 分钟左右才能完成。
sample_predictions_lstm_tbl <- rolling_origin_resamples %>%
mutate(predict = map(splits, predict_keras_lstm, epochs = 300))
现在,我们得到了 11 个样本的预测,数据存储在列 predict 中。
sample_predictions_lstm_tbl
## # Rolling origin forecast resampling
## # A tibble: 11 x 3
## splits id predict
## * <list> <chr> <list>
## 1 <S3: rsplit> Slice01 <tibble [840 x 3]>
## 2 <S3: rsplit> Slice02 <tibble [840 x 3]>
## 3 <S3: rsplit> Slice03 <tibble [840 x 3]>
## 4 <S3: rsplit> Slice04 <tibble [840 x 3]>
## 5 <S3: rsplit> Slice05 <tibble [840 x 3]>
## 6 <S3: rsplit> Slice06 <tibble [840 x 3]>
## 7 <S3: rsplit> Slice07 <tibble [840 x 3]>
## 8 <S3: rsplit> Slice08 <tibble [840 x 3]>
## 9 <S3: rsplit> Slice09 <tibble [840 x 3]>
## 10 <S3: rsplit> Slice10 <tibble [840 x 3]>
## 11 <S3: rsplit> Slice11 <tibble [840 x 3]>
5.2.3 评估回测表现
通过将 calc_rmse() 函数应用到 predict 列上,我们可以得到所有样本的 RMSE。
sample_rmse_tbl <- sample_predictions_lstm_tbl %>%
mutate(rmse = map_dbl(predict, calc_rmse)) %>%
select(id, rmse) sample_rmse_tbl
## # Rolling origin forecast resampling
## # A tibble: 11 x 2
## id rmse
## * <chr> <dbl>
## 1 Slice01 48.2
## 2 Slice02 17.4
## 3 Slice03 41.0
## 4 Slice04 26.6
## 5 Slice05 22.2
## 6 Slice06 49.0
## 7 Slice07 18.1
## 8 Slice08 54.9
## 9 Slice09 28.0
## 10 Slice10 38.4
## 11 Slice11 34.2
sample_rmse_tbl %>%
ggplot(aes(rmse)) +
geom_histogram(
aes(y = ..density..), fill = palette_light()[[1]], bins = 16) +
geom_density(
fill = palette_light()[[1]], alpha = 0.5) +
theme_tq() +
ggtitle("Histogram of RMSE")
而且,我们可以总结 11 个样本的 RMSE。专业提示:使用 RMSE(或其他类似指标)的平均值和标准差是比较各种模型表现的好方法。
sample_rmse_tbl %>%
summarize(
mean_rmse = mean(rmse),
sd_rmse = sd(rmse))
## # Rolling origin forecast resampling
## # A tibble: 1 x 2
## mean_rmse sd_rmse
## <dbl> <dbl>
## 1 34.4 13.0
5.2.4 可视化回测的结果
我们可以创建一个 plot_predictions() 函数,把 11 个回测样本的预测结果绘制在一副图上!!!
plot_predictions <- function(sampling_tbl, predictions_col, ncol = 3,
alpha = 1,
size = 2,
base_size = 14,
title = "Backtested Predictions") { predictions_col_expr <- enquo(predictions_col)
# Map plot_split() to sampling_tbl sampling_tbl_with_plots <- sampling_tbl %>% mutate( gg_plots = map2( !! predictions_col_expr, id, .f = plot_prediction, alpha = alpha, size = size, base_size = base_size)) # Make plots with cowplot plot_list <- sampling_tbl_with_plots$gg_plots p_temp <- plot_list[[1]] + theme(legend.position = "bottom") legend <- get_legend(p_temp) p_body <- plot_grid(plotlist = plot_list, ncol = ncol) p_title <- ggdraw() + draw_label( title, size = 18, fontface = "bold", colour = palette_light()[[1]]) g <- plot_grid( p_title, p_body, legend, ncol = 1, rel_heights = c(0.05, 1, 0.05)) return(g) }
结果在这里。在一个不容易预测的数据集上,这是相当令人印象深刻的!
sample_predictions_lstm_tbl %>%
plot_predictions(
predictions_col = predict, alpha = 0.5,
size = 1,
base_size = 10,
title = "Keras Stateful LSTM: Backtested Predictions")
公众号后台回复关键字即可学习
回复 R R语言快速入门及数据挖掘
回复 Kaggle案例 Kaggle十大案例精讲(连载中)
回复 文本挖掘 手把手教你做文本挖掘
回复 可视化 R语言可视化在商务场景中的应用
回复 大数据 大数据系列免费视频教程
回复 量化投资 张丹教你如何用R语言量化投资
回复 用户画像 京东大数据,揭秘用户画像
回复 数据挖掘 常用数据挖掘算法原理解释与应用
回复 机器学习 人工智能系列之机器学习与实践
回复 爬虫 R语言爬虫实战案例分享