本周我们给大家带来PMO(Practical Molecular Optimization),至今为止最大型的一个分子优化benchmark。文章题为Sample Efficiency Matters: A Benchmark for Practical Molecular Optimization[1],由MIT的Connor Coley组和UIUC的Jimeng Sun组共同完成。在本文中,作者实现/收集/扩展了25种分子优化的代表性方法,并且测试了每一种方法在23个优化性质上的性能(5次独立测试),提出了AUC Top-X的metric,着重对比了算法的优化效率(sample efficiency),在实现分子优化算法的真正应用的道路上迈出了关键的一步。本工作的所有代码开源在了 https://github.com/wenhao-gao/mol_opt ,所有的方法都可以使用一行代码跑通,为将来的算法发展奠定了基础,提供了baseline。点击原文链接可以访问论文。 内容: * 背景与动机 * 方法简介 * 实验结论 * 开源使用 * 总结
背景与动机
药物、材料的发现,本身是一个约束下的多目标优化任务,我们称之为分子优化。分子优化的目标是在一定的化学空间内(例如可合成的化学空间),找到具有理想性质(诸如良好的生物活性与代谢性质,良好的光电效率与稳定性等)的化学分子。由于分子结构本身的复杂性,长久以来最为广泛应用的方法是筛选(Screening),即枚举可能的分子并测试每一个分子的性质。而另一类被称为从头设计的算法则不同,试图通过组合优化等快速得到最优的分子结构,有加速药物设计的潜力。尤其是在过去的4-5年,随着深度学习的突破性进展,新的分子优化算法层出不穷,诸如LSTM (long short term memory network),变分自编码器(variational autoencoder VAE),生成对抗网络(generative adversarial network GAN),深度增强学习(deep reinforcement learning),图神经网络(graph neural network, GNN)等均被应用,可谓进入了百家争鸣的时代。然而在表面的繁华,即许多模型方法文章的提出之下,如何公正地恒量模型的表现,有意义地比较模型却仍存在很大的问题:1)其中最大的问题在于大多数算法文章都不会提及算法的样本效率,即不限定算法调用Oracle来标注数据的次数。目前几乎所有的算法都是随机算法,而无限的oracle budget只会让方法收敛于被探索的化学空间中的最优,因而允许的oracle budget越大,对传统的top-X的指标越有利;2)许多近年的文章仍然仅使用一些简单且没有意义的如QED、LogP等的oracle,甚至自己提出一个新的oracle跑些许实验也是常见的,使得读者不可能直接进行跨文章的比较;3)大多数文章并没有进行深入的调参和重复实验来验证算法的鲁棒性。在分子优化的领域,算法本身多样性强,且代码质量也良莠不齐,很难进行大规模的比较,因此之前的几个Benchmark如Guacamol, TDC等均只有个位数个方法的比较。为了解决这些问题,本文提出了一种名为practical molecular optimization(PMO)的benchmark,试图为分子优化统一度量衡,促进其进入大一统时代。
方法简介
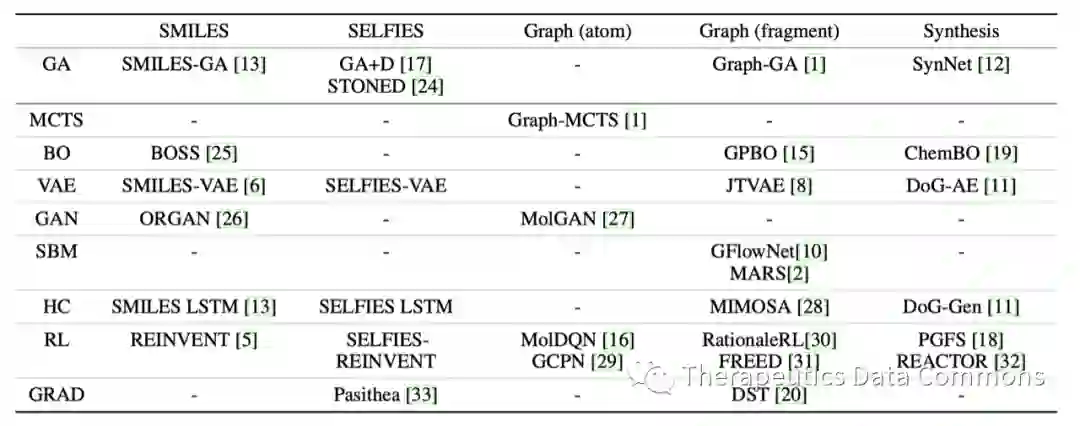
一个分子优化方法有两个基础的组成部分:分子组合方式和优化算法。分子组合方式隐式地定义了算法所探索的化学空间,而优化算法则决定在该空间中如何探索。分子的组合方式类似于机器学习中分子表达方式,主要有以下几种:(1)SMILES字符串;(2)SELFIES字符串;(3)基于原子(atom)的分子图(molecular graph);(4)基于基团(fragment)的分子图(可包括原子);(5)基于化学反应的合成图。目前被应用的优化算法主要包含以下9类, genetic algorithm(GA,遗传算法), Monte Carlo Tree Search(MCTS,蒙特卡洛树搜索), Bayesian optimization (BO,贝叶斯优化), variational autoencoder(VAE,变分自编码器), generative adversarial network(GAN,生成对抗网络),score-based model (SBM,基于分数的概率模型), hill climbing (HC,爬坡算法), reinforcement learning(RL, 增强学习), gradient ascent(GRAD,梯度上升)。每种优化算法理论上都可以和一种分子组合方式一起构成一个完整的分子优化方法,例如Graph GA就是通过遗传算法和基于基团的分子图组合方式构成的。作者实现/收集/扩展得到了目前最有代表性的25种分子优化方法进行比较。
作者认为一个理想的分子优化方法本身应该具有强大的优化能力、高效的样本效率、对不同优化目标的适应能力,以及对超参和随机种子的一定的鲁棒性。为了能够更公正、有效地比较不同算法在这些目标上的表现,作者选择了QED,DRD2,JNK3,GSK3,以及来自Guacamol的19个oracle,共23个oracle进行测试,所有模型限制在10,000次oracle调用以内。此外,在模型评价上,在被广泛使用的Top-X,也即所有测试过的分子中最好的X个的平均分数以外,作者提出了AUC Top-X这一metric来更好地区别样本效率的强弱。AUC Top-X即Top-X对调用oracle次数的曲线的线下面积,在最终分数相近时,更早达到该分数的方法,也即样本效率更高的方法会有优势。最终作者使用了6个相关的指标来衡量优化的性能(AUC-TOP1/10/100,和TOP1/10/100的平均数),其中以AUC-Top-10为主要metric。作者对每个方法都进行了调参,且每个任务上均跑了五次独立测试,汇报了结果的均值和方差。
实验结论
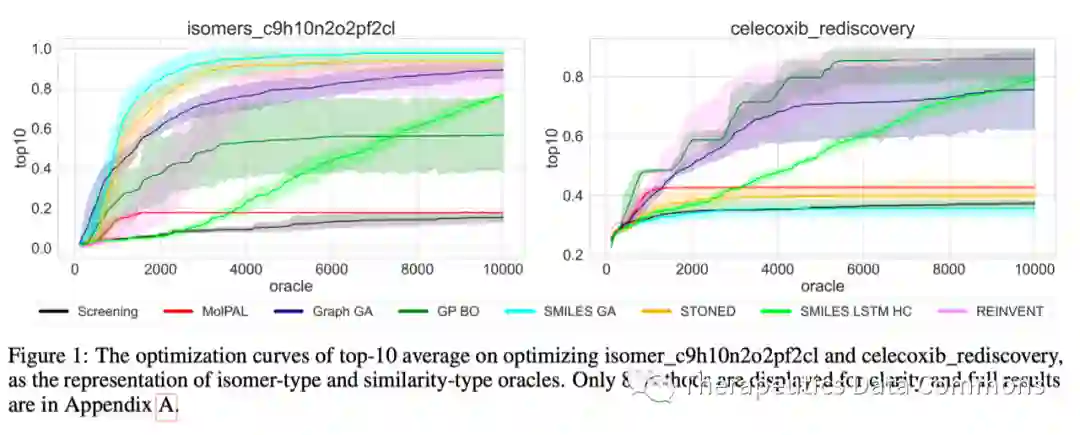
样本效率是优化方法的重要指标,目前所有方法,除了在一些极简单的oracle以外,均不能在调用几百次oracle的情况下完成优化。这说明目前所有的分子优化算法在小分子上均难以直接以实验测量为oracle优化分子。通过比较top-X和AUC Top-X的排名,我们也可以看出一些在Top-X是哪个表现尚可的方法在AUC Top-X上表现不佳,这说明了这些我们以前认为很强的算法,如Hill Climbing,在使用样本使用效率方面并不不强,证明了AUC这一指标的意义。 传统方法仍然强大,在本文的测试中,综合排名前两位的方法分别是REINVENT(基于SMILES的增强学习方法),Graph GA(基于分子图的遗传算法)。其中,REINVENT是由AstraZeneca公司在2017年开发的,使用了基于policy gradient的增强学习来训练GRU生成SMILES字符串,而遗传算法拥有几十年的历史,并不需要深度学习,实现简单。这两个方法均没有发表在AI顶会,而反观许多近些年AI顶会的论文变现不佳,令人深思。这也侧面反映了标准benchmark在分子优化中的迫切性。
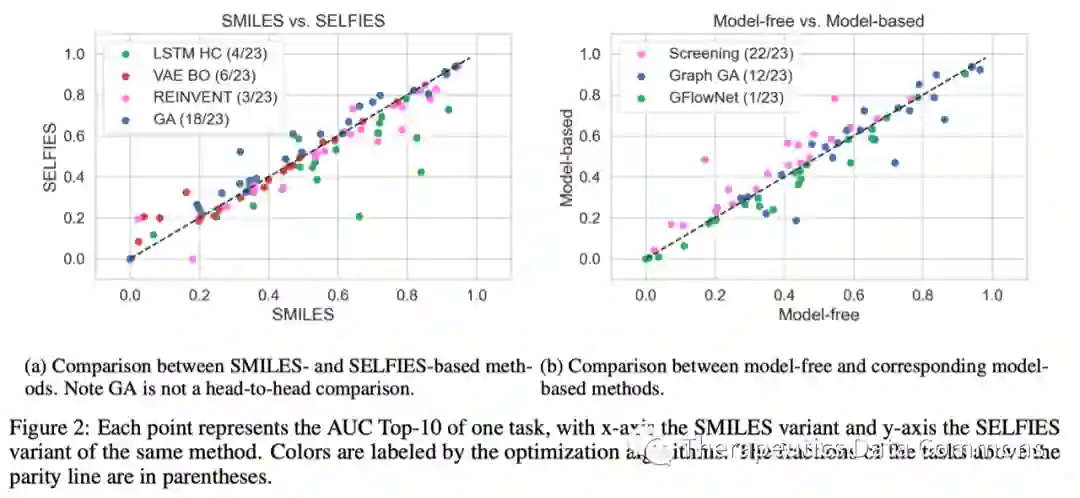
SMILES没有明显的短板,曾经SMILES因其无法保证生成的分子的有效性而饱受诟病,而SELFIES因为能够保证任意字符组合的有效性而大受追捧。然而,本文通过一系列SMILES和SELFIES的对比试验中发现,SMILES在大部分的情况下都要强于SELFIES。作者认为这主要是因为目前的语言模型已经能够学习SMILES的语法,保证生成的分子有足够多的有效分子,因而磨平了SELFIES的优势。另一方面,通过案例研究发现,实际上在SELFIES中也存在语法错误的问题,只是在SMILES中出现语法错误会报错,而在SELFIES中则把错误掩盖起来,虽然得到的分子在键价上是有效的,但并没有带来有效的化学空间探索,详情可见附录D。 Model-based方法理论上可以比model-free方法更高效,但需要精心的设计。model-based的优化方法是通过训练一个predictive model作为真实oracle的替代品,以优化之,来达到更高效的优化的目的。这一原则在Screening和MolPAL的比较之间得到了验证,但在其他模型里效果却参差不齐。作者将其归因于predictive model训练的质量,在predictive model训练不充分时,model-based的优化方法反而会被误导进歧途。另一方面,表现相对较好的MolPAL和GP BO均评估了不确定性用以指导分子的选取。这说明了model-based的优化方法需要更精心的设计,简单加一个GNN训练一下并不一定有理想中的提升。
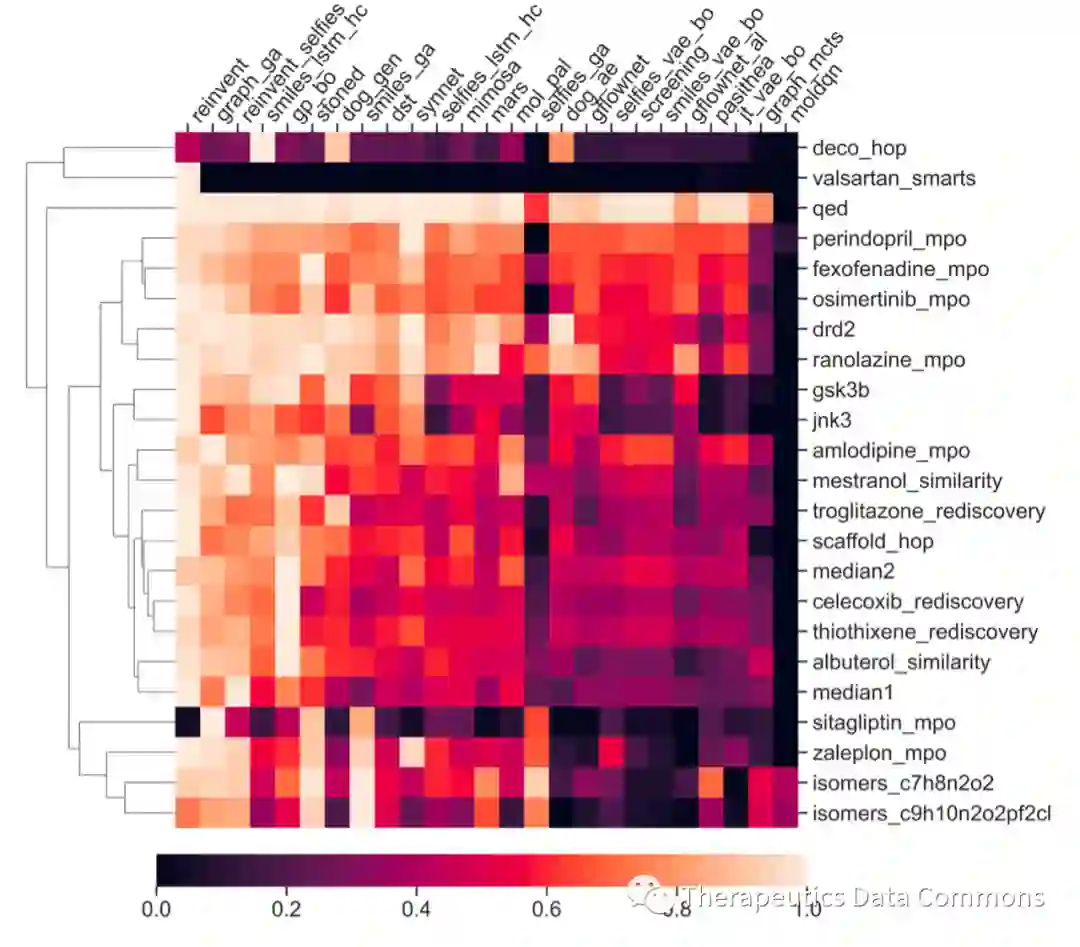
不同方法适用于不同的任务(优化目标),作者发现比如在一些基于异构体的任务中(isomer_c7h8n2o2, isomer_c9h10n2o2pf2cl, sitagliptin_mpo, 和zaleplon_mpo),基于字符串的遗传算法方法(比如SMILES-GA,STONED基于SELFIES)效果更佳。而在基于相似度的任务中,这些方法效果并不理想。这说明了不同的优化方法在不同类型的任务上有不同的适应性,取决于oracle的landscape。作者通过所有方法的相对表现进行聚类,分析得到所测试的oracle有基于异构体的、基于相似度的、基于片段是否存在的几种大类,其中直接使用机器学习方法拟合实验数据的JNK3和GSK3则与基于相似度的oracle更为接近。在真正使用时,我们仍需要根据任务的特点,选择恰当的分子优化算法。 超参数的选择和多次测试也很关键,比如通过超参数调整我们得到了相比于REINVENT的原始paper中更好的结果,说明了在比较不同模型时,进行调参的重要性。
开源使用
本工作的另一亮点就是可重复性。所有的代码,数据和预训练模型公开在了 https://github.com/wenhao-gao/mol_opt 。所有的数据准备和模型预训练(pretraining)均被提供,使得所有的方法都可以使用一行代码跑通,比如 python run.py graph_ga --task production --n_runs 5 --oracles qed 即使用基于分子图的遗传算法(Graph GA)优化QED(类药性),进行五次独立重复实验。通过将task更换为tune既可实现调参。除了少数方法外,大部分方法都可以在一个统一的环境下运行。对于想优化其他性质的用户也可以构建一个类TDC的oracle function来进行优化。其他详情请见github。
总结
本篇文章进行了目前为止最大规模的分子优化算法基准测试,着眼于样本效率这一以往被忽视的重要的性质,分析了目前算法的表现并给出了今后算法开发的建议。值得注意的是本文重点在样本效率,因此没有广泛讨论其他诸如可合成性、多样性等重要性质。作者声明会继续维护并完善这一benchmark,以使得分子优化算法尽快达到实用水平。
Therapeutics Data Commons(TDC)致力于推动机器学习和生命医学领域的融合,让更多的机器学习/生物医药研究者可以无门槛参与到实用且有价值的数据驱动模型开发中来。我们核心团队决定不定期suí biàn gē在本公众号分享领域相关的文章的解读,希望能对读者有所帮助,有任何反馈,或者有希望解读的文章与专题,欢迎在公众号后台留言!我们也欢迎相关专题的投稿,有兴趣的请关注!
作者:高文昊 符天凡 审稿:黄柯鑫 编辑:高文昊
参考资料
[1] Gao, W., Fu, T., Sun, J., & Coley, C. W. (2022). Sample Efficiency Matters: A Benchmark for Practical Molecular Optimization. arXiv preprint arXiv:2206.12411.