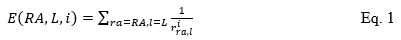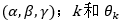计算机辅助药物设计中的一项重要任务是发现与致病蛋白具有高结合亲和力的先导化合物。由于蛋白质-配体结合亲和力预测在很大程度上依赖于配体在结合口袋中的位置,因此从配体众多对接姿势中挑选出最优结合姿势至关重要,这将影响到虚拟筛选结果的可信度。本文介绍的是2022年12月发表在国际知名生物信息学期刊《Briefings in Bioinformatics》(IF=13.994)上发表的一篇题为《A fully differentiable ligand pose optimization framework guided by deep learning and a traditional scoring function》的研究论文。该论文重点阐述了如何基于深度学习算法进行小分子构象的选择和局部优化。论文的共同第一作者是王泽琛(山东大学物理学院)和郑良振博士(上海智峪生科和中科院深圳先进院),共同通讯作者是郑博士和李伟峰教授(山东大学物理学院)。
图1. 小分子构象优化论文截图
在基于结构的虚拟筛选中,分子对接通常被用于探索配体在结合到蛋白质上时的潜在构象。当前的分子对接程序主要受制于打分函数的精度,致使较高的假阳性率。随着蛋白质-配体结构与结合亲和力数据的持续扩增,基于机器学习和深度学习的打分函数有望实现更高的精度。研究表明,打分函数的打分能力 (scoring power) 和对接能力 (docking power) 之间并没有很强的关联性。尽管一些基于机器学习和深度学习的打分函数已经被证明具有较强的打分能力,但是在对接任务中却表现很差,甚至不如传统的打分函数。因此,设计一个基于机器学习或深度学习的打分函数来选择配体的近天然构象,并明确地指导分子对接场景中的采样过程是非常有必要的。
虽然近些年基于机器学习和深度学习的打分函数层出不穷,但是它们大多聚焦于亲和力预测,当迁移到分子对接场景上时表现非常有限。此外,当前应用于分子对接场景的打分函数通常是对计算机产生的配体结合姿势进行重新打分,如GNINA。然而,基于深度学习算法来指导配体在蛋白质结合口袋中的结合行为仍然处于起步期。在大规模分子对接任务中,快速且有效地提升配体结合姿势的质量具有重要意义。
方法介绍
在评估对接能力时,一般认为RMSD(小分子的方均根偏差)越小的结合姿势具有更大的结合亲和力。因此,对于同一个蛋白质-配体复合物的不同结合状态,RMSD值可以近似成表征亲和力大小的另一种形式。论文作者提出的DeepRMSD是一个预测配体结合姿势RMSD的打分函数,将其与AutoDock Vina打分函数相结合 (DeepRMSD+Vina) 可以实现更高的对接成功率。鉴于DeepRMSD+Vina是对配体坐标完全可导的,因此基于该打分函数应用梯度回传构建了一个配体构象优化框架。
作者使用了PDBBind v2019中的蛋白质-配体复合物结构以及AutoDock Vina生成的对接姿势作为DeepRMSD的训练、验证集,同时使用了主流打分函数评价标准CASF-2016以及交叉对接数据集DISCO作为测试集。分子对接产生的结合姿势与天然构象之间的RMSD作为模型训练的标签。
在特征提取方面,作者首先计算了蛋白质与配体原子两两之间的距离,并对这些距离值分别作-1和-6次幂处理,然后根据特定的蛋白质-配体原子组合方式和距离处理方式分别进行求和,进而得到该原子组合的特征值 (Eq.1):
式中RA和L分别是蛋白质和配体的原子类型,i为-1或-6。将所有的特征值对接在一起形成该蛋白质-配体最终的特征向量,应用多层感知机 (MLP) 来拟合特征向量与RMSD之间的关系。这种特征描述方式使得DeepRMSD分数对分子坐标可导,从而为接下来搭建配体构象优化框架奠定了基础。
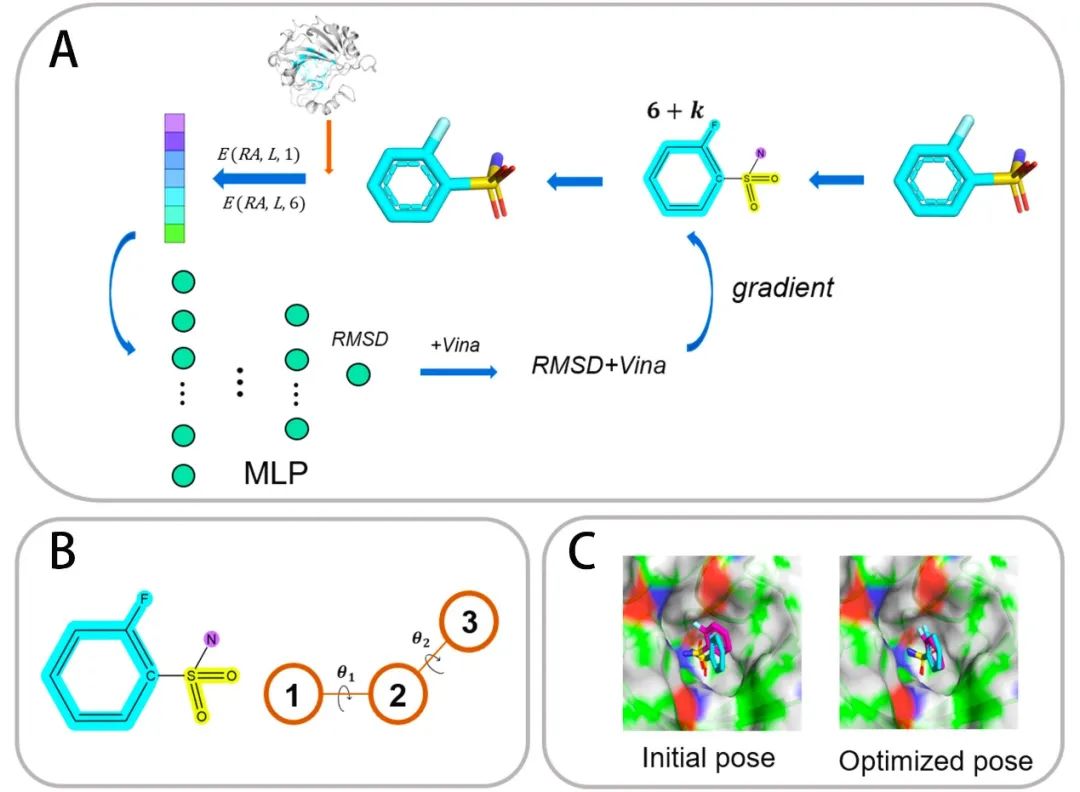
作者定义了一个长度为6+k的向量
来表征配体的构象。其中该向量的前六个值分别是第一个原子的3D坐标和在空间中绕x, y, z轴旋转的角度
指的是配体可旋转键的数目和第k个可旋转键的扭转角度。在构建配体构象优化框架时,并没有直接对配体每个原子的坐标进行改变,而是通过对分子进行平移、旋转以及扭转分子内部的可旋转键来实现,进而保证了配体分子在优化过程中结构的合理性。配体构象优化流程如图2所示:首先,配体分子被编码成长度为6+k的向量,这是该优化框架中对配体结合姿势最原始的表示。然后,由该向量还原出配体的3D坐标,用于提取蛋白质-配体相互作用特征以及计算Vina score。特征向量被输入到神经网络中来产生结合姿势的RMSD,将RMSD值与Vina score结合在一起作为最后的综合得分。至此,一个以配体向量为输入、以DeepRMSD+Vina的综合打分为输出的计算图构建完成。为了实现配体结合姿势的迭代优化,每当DeepRMSD+Vina打分完成,便计算综合分数对配体向量的导数,并将其作用于当前的配体向量来生成新的配体向量。当DeepRMSD+Vina分数不再显著下降时,优化停止。
图2. 基于DeepRMSD+Vina构建的配体构象优化框架
结果描述
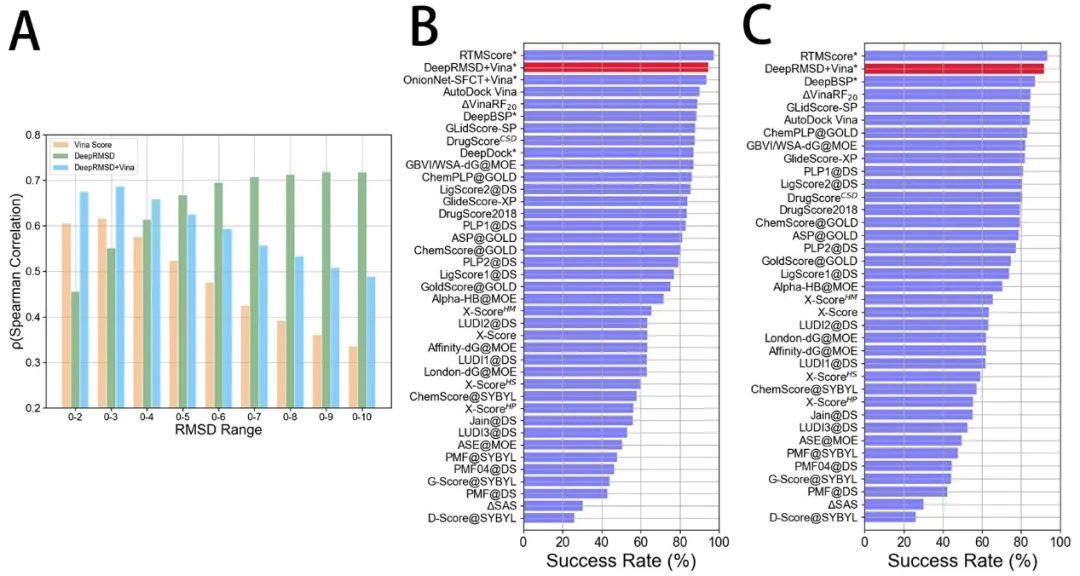
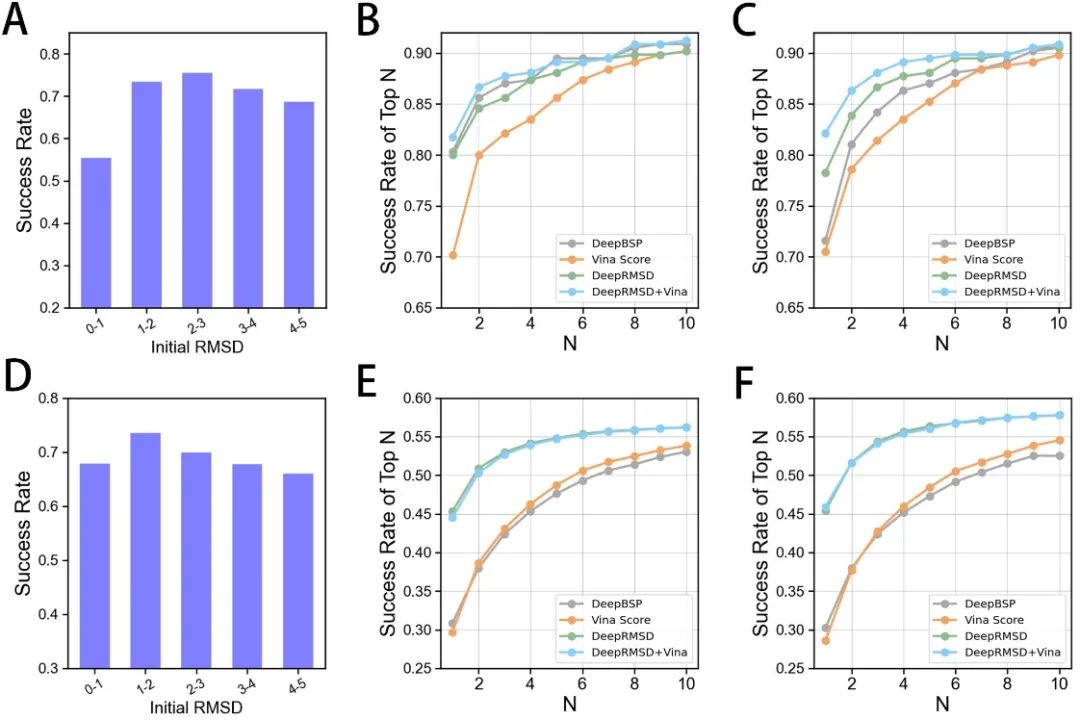
DeepRMSD+Vina的对接能力评估 在CASF-2016对接测试集上的结果表明,DeepRMSD在高RMSD值的结合姿势上表现较好,相反,Vina score在低RMSD的结合姿势上表现更好 (图3a)。分子对接的主要目的是区分出一个或几个近天然结合姿势,这些结合姿势通常具有较低的RMSD值,因此打分函数在低RMSD区间的准确性十分重要。有趣的是,DeepRMSD+Vina作为两者的结合,在低RMSD区间能够取得更理想的效果。在CASF-2016 docking power测试中,DeepRMSD+Vina实现的Top 1成功率达到94.4 %(图3b,包含天然结构)和91.6%(图3c,不包含天然结构)。
图3. 在CASF-2016上的对接能力测试
DeepRMSD+Vina和配体构象优化框架在实际应用场景中的评估 在实际分子对接场景下,通过分子对接软件将配体对接到靶体上产生一定数目的结合姿势,然后通过打分函数对这些结合姿势进行打分排序,挑选出排名靠前的结合姿势。通常情况下,这些排名靠前的结合姿势仍然与天然构象之间存在或大或小的差异。因此,这些结合姿势的质量仍然有进一步提高的空间。理想情况下,经过优化后的RMSD+Vina分数与真实RMSD值同时下降,代表优化成功。但是,受制于蛋白质结合口袋的形状与大小,很多情况下配体的活动范围有限,这使得优化后的RMSD+Vina值未必会下降。因此,作者只考虑优化后RMSD+Vina下降的样本来统计优化成功率。作者在重对接 (redocking) 和交叉对接 (cross-docking) 任务中测试了配体构象优化框架的优化成功率 (图4a和4d)。可以看出,对于RMSD在1-4之间的结合姿势,优化框架表现优异,有能力进一步提高结合姿势质量。在redocking(图4b,4c)和cross-docking(图4e和4f)对接成功率上,DeepRMSD+Vina显著优于基准打分函数Vina score和DeepBSP。
图4. DeepRMSD+Vina和配体构象优化框架在redocking和cross-docking任务中的表现
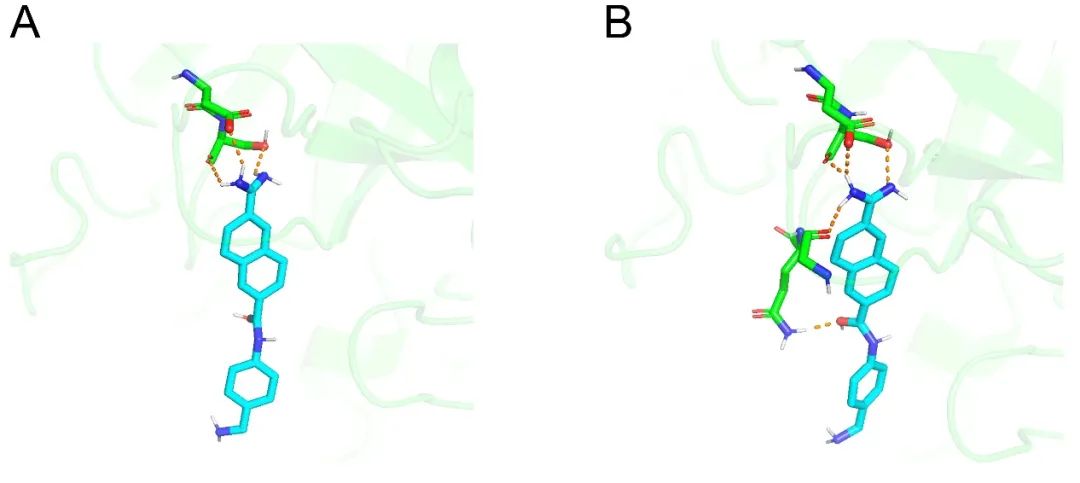
优化结构分析 理想情况下,成功优化的结构能更接近天然构象,并且有更强的分子间相互作用。这些高质量的结合姿势在优化过程中或许会受到一些关键相互作用的影响,比如氢键、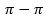
图5. 结合姿势 (PDB: 1OWH) 优化前后氢键形成情况。青色表示配体结合姿势,绿色与配体形成氢键的关键残基,橙色虚线表示氢键。
结语
该论文基于深度学习算法设计了一个预测配体结合姿势RMSD的打分函数DeepRMSD,通过与传统打分函数Vina score结合,DeepRMSD+Vina能够取得更高的对接成功率。论文中强调了对分子坐标可导的打分函数在未来分子对接和分子优化领域的重要性。作者基于DeepRMSD+Vina设计了一个配体构象优化框架来优化配体在受体结合口袋中的构象,这为深度学习算法在分子结构优化领域提供了范例,并有助于提高未来分子对接程序的性能,助力基于结构的虚拟筛选。
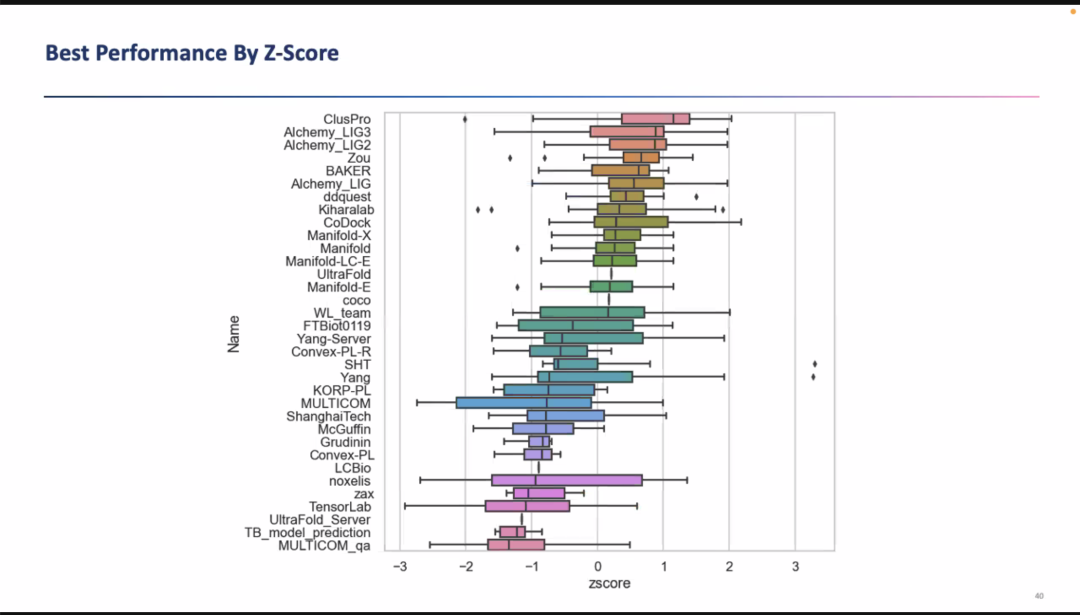
同时,基于transformer的小分子构象预测模型结合DeepRMSD+Vina的局部构象优化,助力了郑良振博士和王晟博士(智峪生科CEO)带领的智峪生科AIchemy-LIG系列算法在CASP15的蛋白质-小分子复合体预测竞赛上获得优秀成绩(如图6,按全部构象评分第二名,按第一构象评分第三名)。
图6. CASP15蛋白质-小分子复合体预测排名(按照全部构象)
参考资料 Wang, Z., Zheng, L., Wang, S., Lin, M., Wang, Z., Kong, A.W.K., Mu, Y., Wei, Y. and Li, W., A fully differentiable ligand pose optimization framework guided by deep learning and a traditional scoring function. Briefings in bioinformatics, p.bbac520.