项目名称: 面向蛋白质分子位点标记的多源特征提取和深度序列学习方法研究
项目编号: No.61462018
项目类型: 地区科学基金项目
立项/批准年度: 2015
项目学科: 计算机科学学科
项目作者: 樊永显
作者单位: 桂林电子科技大学
项目金额: 47万元
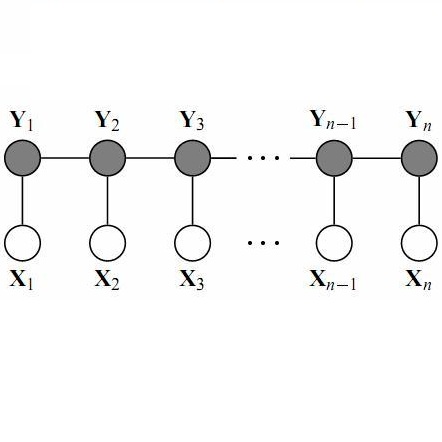
中文摘要: 在后基因组时代,越来越多的蛋白质分子序列被测定出来,如何确定蛋白质分子的功能位点是最重要的问题之一。传统生物实验的方法费时费力,在这种情况下,计算方法应运而生。本研究拟从蛋白质分子多源、异构、复杂的特征入手,首先研究这些特征的相似性度量问题;其次研究序列特征、结构特征、网络特征、共进化特征、进化踪迹特征、物理/生物化学属性特征等的提取计算分析方法,以寻找确定蛋白质分子位点在各种特征下的相似性模体,为蛋白质分子位点标记提供可解释的理论依据;再结合深度学习和条件随机场理论研究蛋白质分子的功能位点标记问题。最后整体形成有效的蛋白质分子位点标记的计算方法,并基于构建的模型进行个案研究和全基因组分析,为计算和生物学研究提供新的认知。
中文关键词: 蛋白质分子;位点标记;特征提取;条件随机场;深度学习
英文摘要: During the post-genomic era, a growing number of protein sequences are determined, how to identify their functional sites is one of the most important issues. The traditional biological experimental determination and validation on functional sites are usually laborious and time-consuming. To timely and effectively discover protein functional sites when facing with the avalanche of new protein sequences, computational methods are emerging. In this project, as protein molecules are multi-source, heterogeneous and complex, we will study from the following several aspects: firstly, different similarity measures for each features of protein molecules are presented. Secondly, to find different motifs of protein functional sites that provide an interpretable theoretical basis for protein functional sites labeling, sequence features, structure features, network features, co-evolution features, evolutionary trace features, physical/biological chemical property features are computed and analyzed. Furthermore, a novel method, deep sequential learning machine, is proposed for labeling protein molecule functional sites based on Deep Learning and Conditional Random Fields. Finally, an effective method for labeling sites of protein molecules is presented, and case studies and genome-wide analysis are carried out using the built prediction models to provide new insights into computation and biology.
英文关键词: Protein molecule;Sites labeling;Feature extraction;Conditional Random Fields;Deep Learning