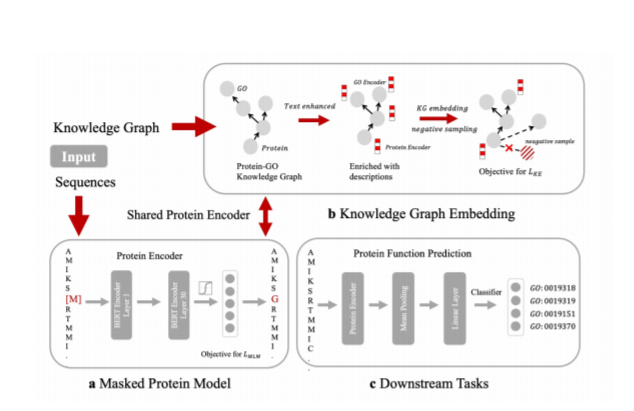
近年来,预训练模型以强大的算法效果,席卷了自然语言处理为代表的各大AI榜单与测试数据集。与自然语言类似,蛋白质的一级结构具有序列特性,这为将语言预训练模型引入蛋白质表示提供了有利条件。然而,蛋白质本质上不同于自然语言文本,其包含了大量预训练目标较难习得的生物学知识。事实上,人类科学家已经积累了海量的关于蛋白质结构功能的生物学知识。那么如何利用这些知识促进蛋白质预训练呢?本文将介绍被ICLR2022录用的新工作:OntoProtein,其提出一种新颖的融入知识图谱的蛋白质预训练方法。
蛋白质是控制生物和生命本身的基本大分子,对蛋白质的研究有助于理解人类健康和发展疾病疗法。蛋白质包含一级结构,二级结构和三级结构,其中一级结构与语言具有相似的序列特性。受到自然语言处理预训练模型的启发,诸多蛋白质预训练模型和工具被提出,包括MSA Transformer[1]、ProtTrans[2]、悟道 · 文溯[3]、百度的PaddleHelix等。大规模无监督蛋白质预训练甚至可以从训练语料中习得一定程度的蛋白质结构和功能。然而,蛋白质本质上不同于自然语言文本,其包含了诸多生物学特有的知识,较难直接通过预训练目标习得,且会受到数据分布影响低频长尾的蛋白质表示。为了解决这些问题,我们利用人类科学家积累的关于蛋白质结构功能的海量生物知识,首次提出融合知识图谱的蛋白质预训练方法。下面首先介绍知识图谱构建的方法。
当下蓬勃兴起的 AI for Science 正在促使以数据驱动的开普勒范式和以第一性原理驱动的牛顿范式的深度融合。基于“数据与知识双轮驱动”的学术思想,我们在本文中首次提出了融合知识图谱的蛋白质预训练方法OntoProtein,并在多个下游任务中验证了模型的效果。在未来,我们将维护好OntoProtein以供更多学者使用,并计划探索融合同源序列比对的知识图谱增强预训练方法以实现更优性能。