图谱实战 | 斯坦福黄柯鑫:图机器学习在生物图上的应用
转载公众号 | DataFunSummit

分享嘉宾:黄柯鑫 斯坦福大学 博士生
编辑整理:元玉蒲 西北大学
出品平台:DataFunTalk
导读:大家好,我叫黄柯鑫。我现在是斯坦福大学的计算机科学博士第一年级,研究方向是机器学习在生物医疗的实际应用场景方面的挑战。本次分享题目为分子网络中的图机器学习,主要介绍图机器学习在生物系统图上的应用。
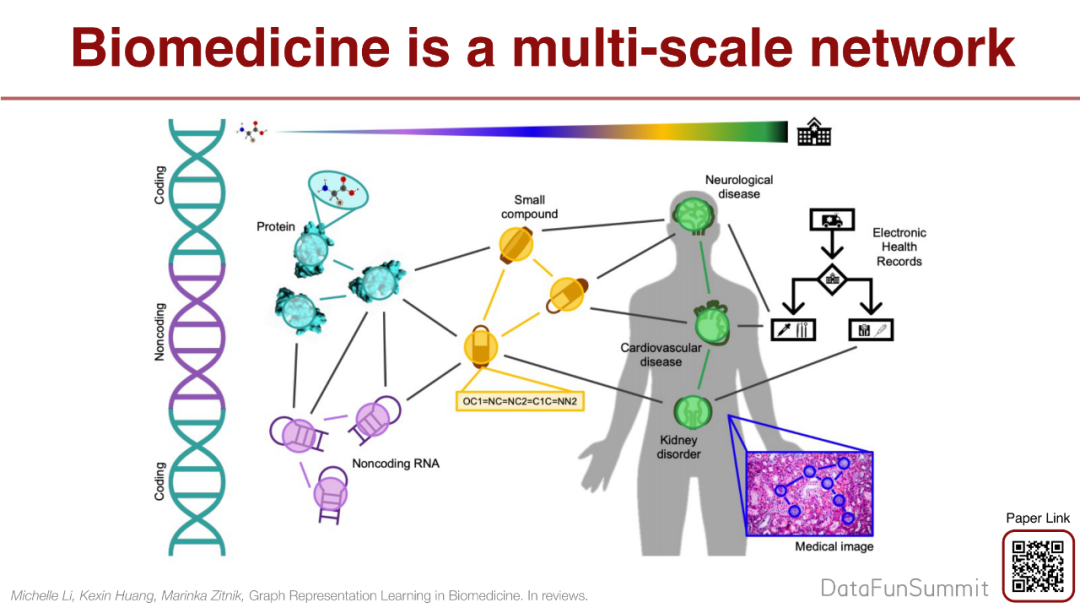
人体的很多功能是由复杂的小的生物实体进行反应,比如说话、吃东西的背后有各种细胞在工作。比如:在吃东西的时候,食物会引起一系列的信号到细胞,激发一系列的反应,翻译为蛋白质,不同的蛋白质合作形成功能,如果某些基因不工作会引起疾病。生物医疗领域是一个非常复杂的图,生物实体有蛋白质、RNA、药物、疾病等,实体之间的链接代表关系,设计药品解决有问题的蛋白质。
生物医学是一个多尺度的网络,现在有很多知识图可以回答一些生物问题。如果想知道这能不能解决病,即要预测药和疾病会不会有反应。如果预测是高质量的,就可以解决在传统的生物领域要花费很长时间的问题,达到加速解决过程的目的。
今天我会从下面的5个方面讲图机器学习在生物系统中的方向和实际应用。
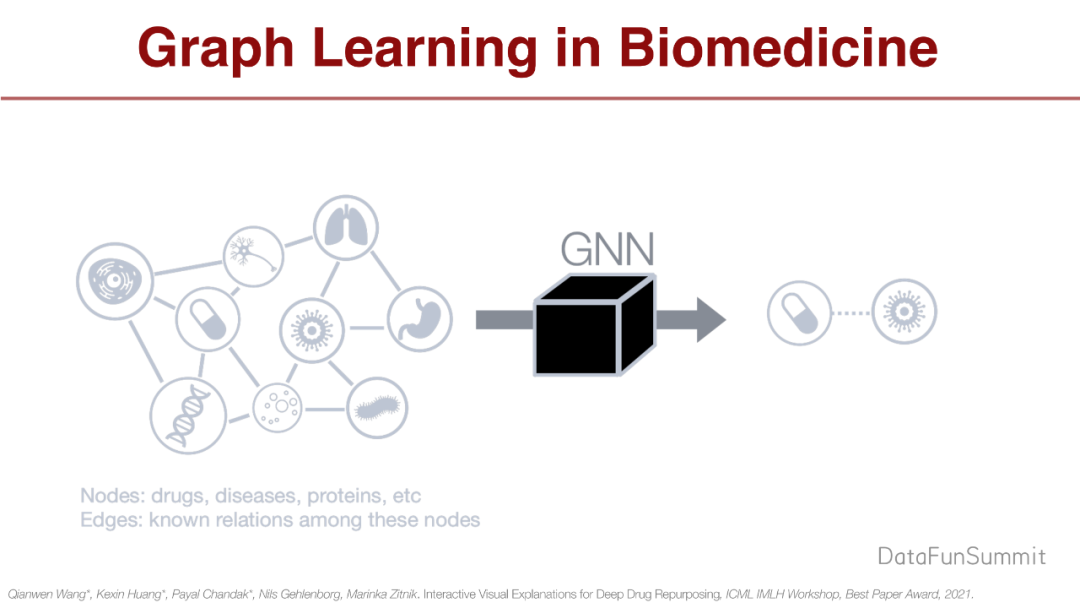
第一个我会讲GNN应用到生物系统上需要注意的地方。GNN开始训练的时候,利用homophily principle的性质,即相邻节点的性质相似。
把邻居节点传给中心目标节点,在信号处理角度就是促进节点的embedding保持一致。这个性质适合于social网络,但并不适合于molecular网络。
Direct similarity指的是social网络,节点之间的边代表similarity,但生物网络不是这样的。
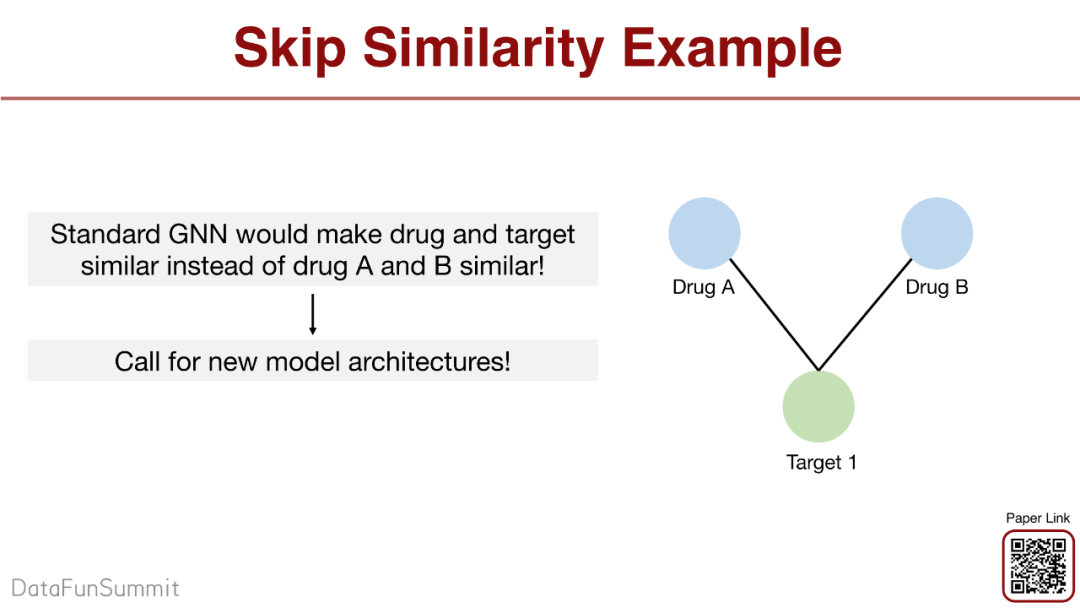
举个例子,药A和目标连接(会反应),但是药A和目标不是同一类型,所以药A和目标的embedding不同。假如药A和药B同时和目标反应,药A和药B不会反应但具有相似的embedding,标准GNN具有这样的特征。
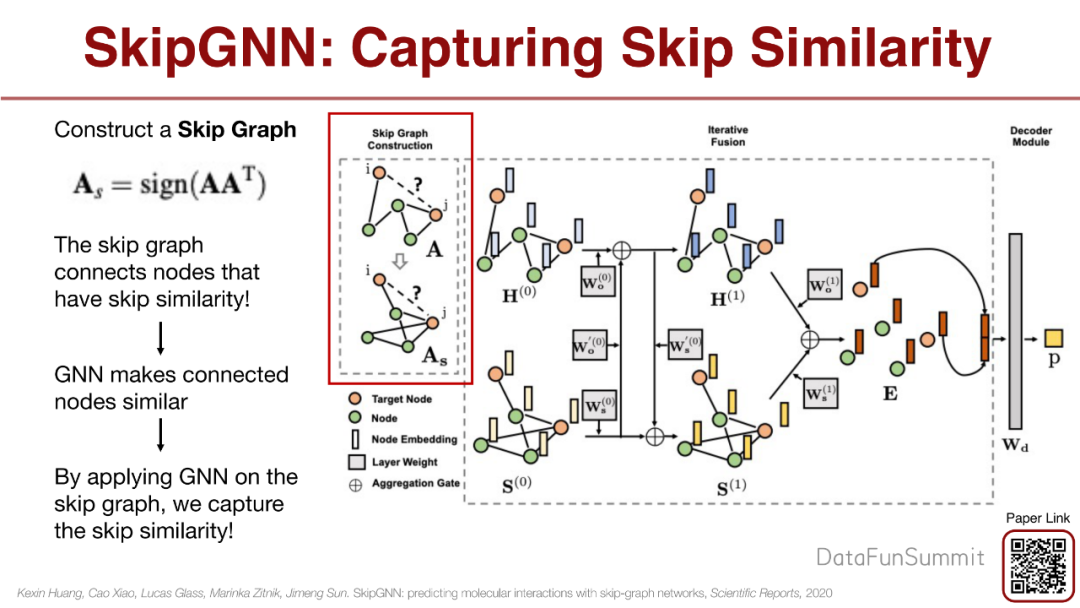
所以我们提出了SkipGNN,将药A和药B连接在一起,同时将药A、药B与目标的边断开。在新的图上做GNN,会促进连接的节点相似,间接达到了促进Skip similarity。
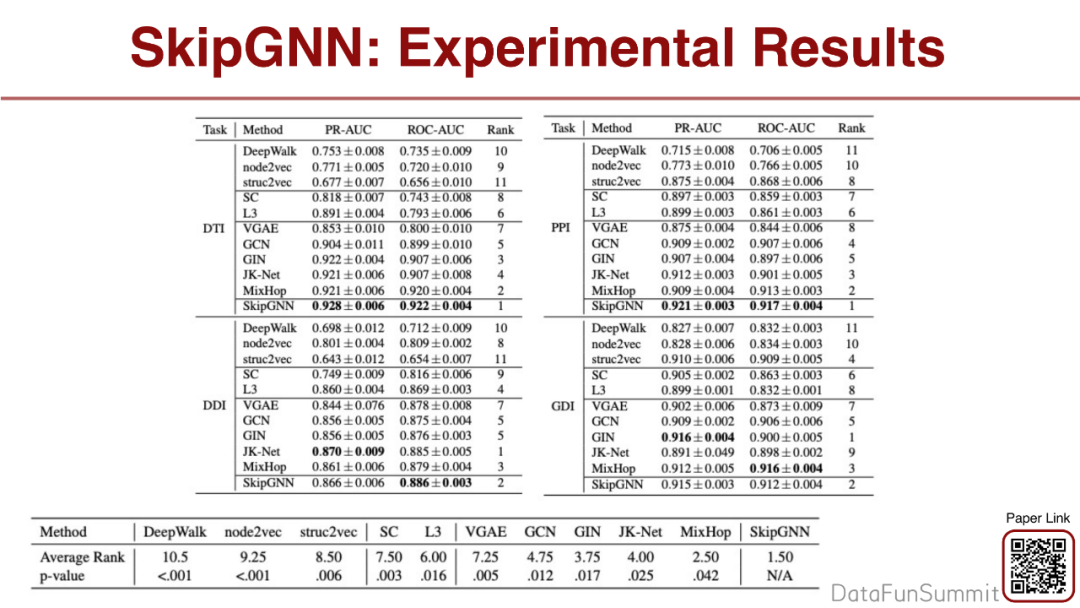
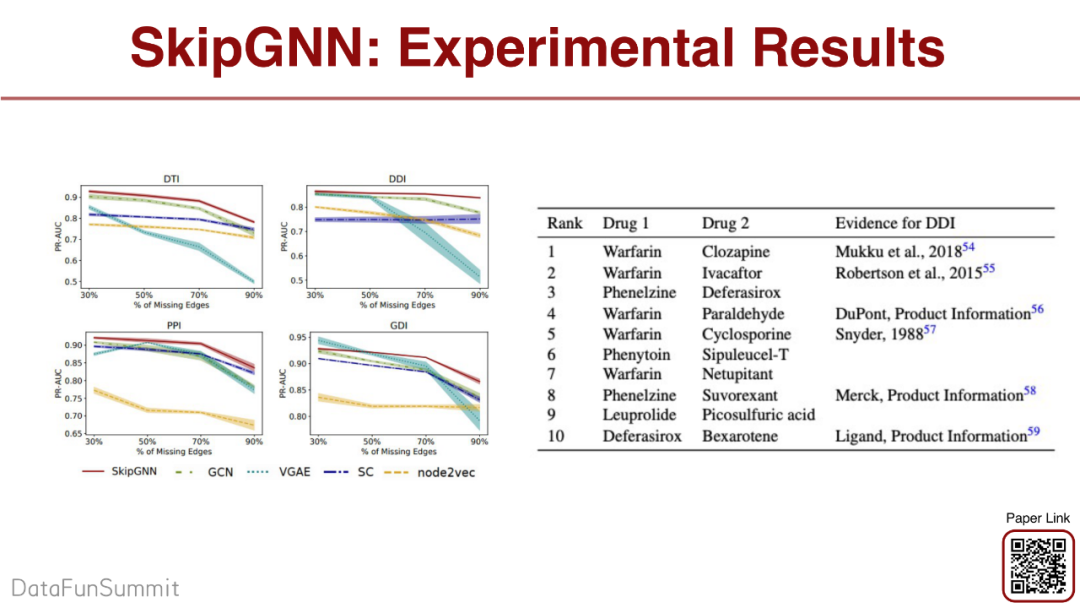
实验结果发现,SkipGNN的效果相比DTI、DDI、PPI、GDI得到提高。
我们还找到了一些基于SkipGNN的药,但无法用在其他方法。
在传统的GNN中,基因和药是混在一起的。但是我们想要分离基因和药,SkipGNN是可以达到这样的效果。
第一部分,我想强调的一点是,在从传统GNN到生物图,不能直接应用,因为生物系统图和其他网络相比有很多不同的性质。在这其中也有很多机会发现生物图的性质去设计新的图机器学习的方法。
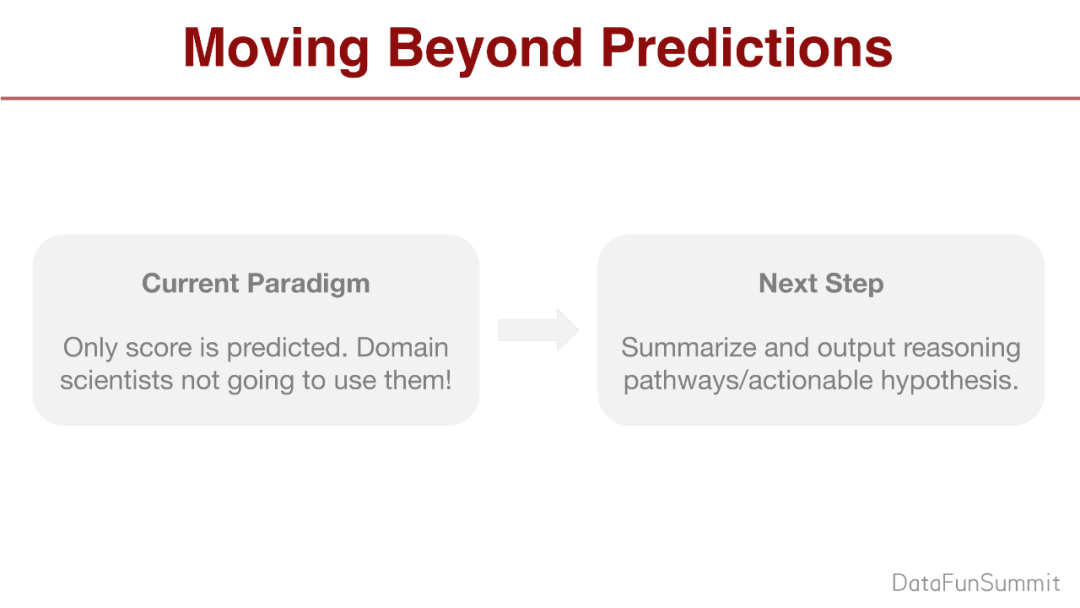
之前我们注重的是如何做预测,但更加重要的是,如果生物科学家不懂机器学习,是不会相信AI模型产生的东西,所以要产生更多的信息。生物学家需要的是一个假设,因为蛋白质和基因有关系,所以蛋白质和药可以有反应,这才是生物学家更加感兴趣的东西。
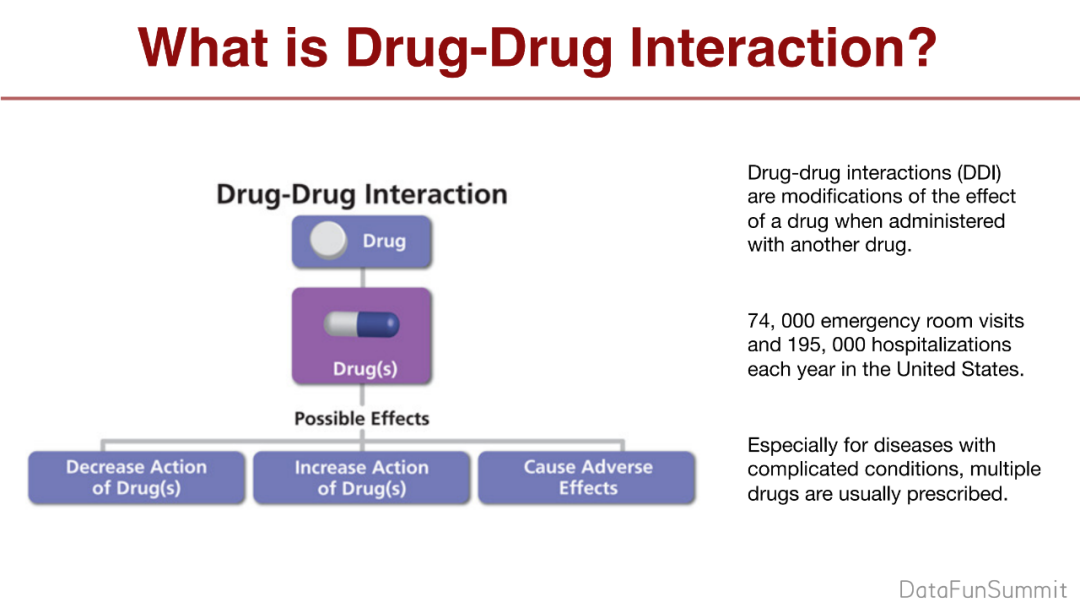
我们研究的一个问题就是Drug-Drug Interaction(DDI),DDI是两种药物共同起效时造成药效的改变。在美国,每年大概有20万次住院就诊。
为什么DDI会是一个问题?FDA批准3568种药,其中有超过100种相互作用的类型,2种药物的DDI有3568×3568×100=1273062400种,更不要说多种药物的DDI。不可能在体外实验中验证,更不用说严格的临床试验了!我们可以输入药物的信息到机器学习模型中,就可以计算出各种DDI类型。
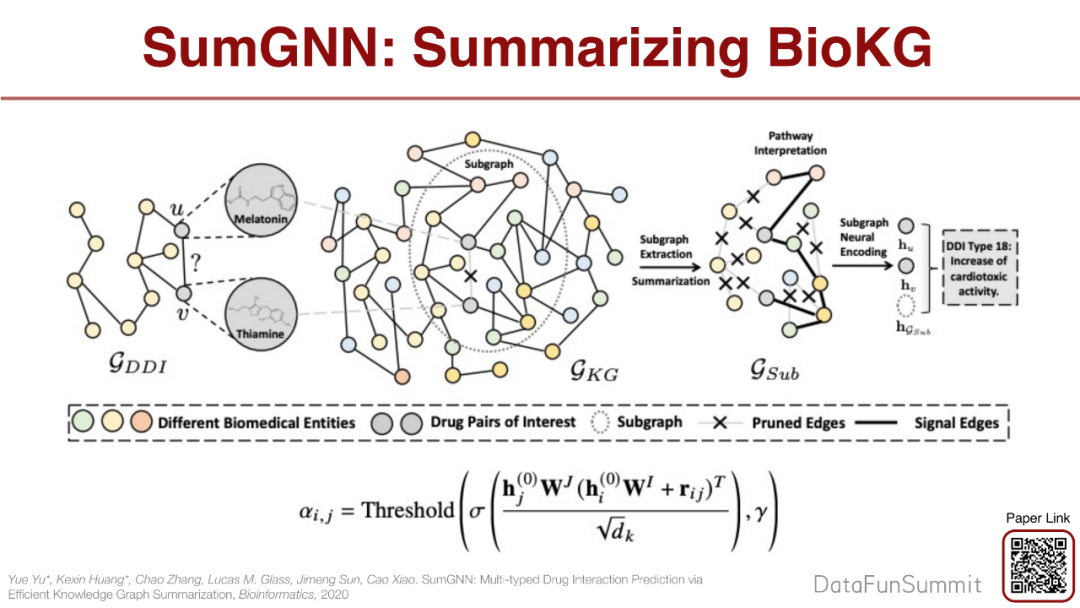
我们不仅仅想做预测,还想产生推理路径和可操作的假设。在系统生物图上聚焦到2种药物,这2种药物有很多连接节点,就可以找到节点的关系,就可以分析药物的反应。
这是非常好的图示,2个感兴趣的节点Melatonin和Thiamine输入到生物网络里,提取到子图。预测边对于prediction是否重要,若重要则予以保留,用保留的边做最后的预测,经过不断训练得到的边就是推理路径。
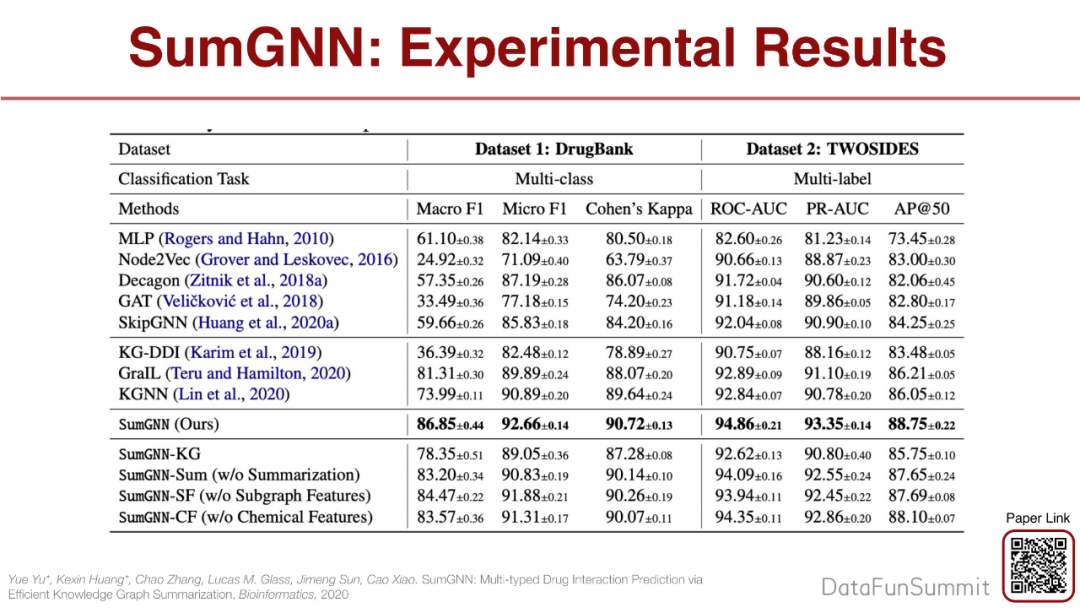
SumGNN的实验结果也非常不错。
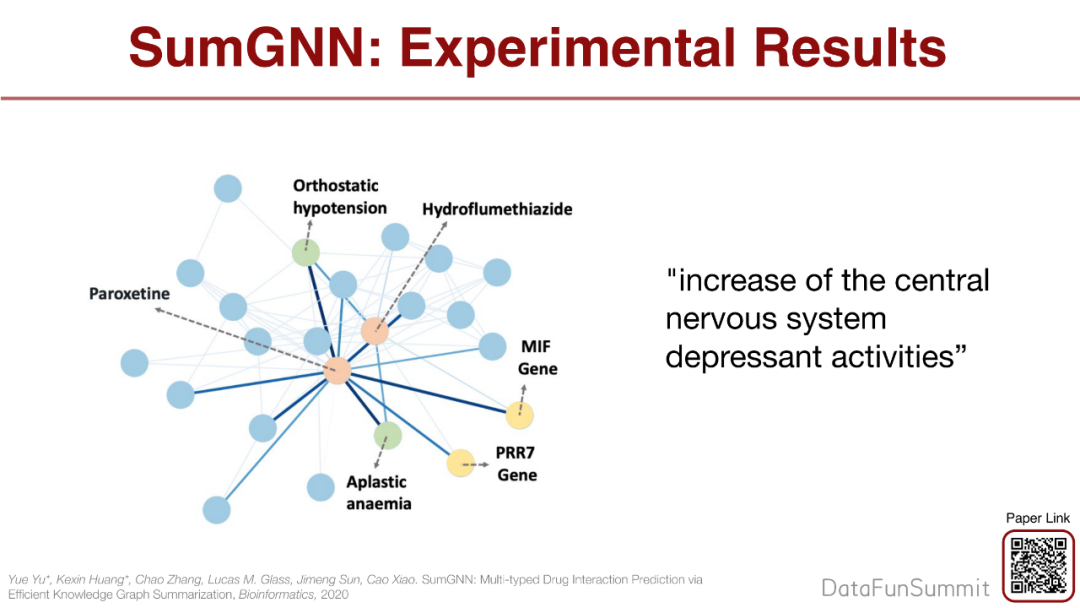
比如我们感兴趣的是Paroxetine和Hydroflumethiazide的2种节点,绝大部分边已经被去掉。当同时吃上面2种药的时候,Orthostatic hypotension和Aplastic anaemia这2种疾病的发病率会提高。
第3部分我们更加往领域科学家使用的角度去思考,不仅仅有解释、预测、可操作的假设,更重要的是接口是什么?
GNN在生物方面的目标是生成模型的预测和解释,让领域科学家更好地理解AI的解释结果。
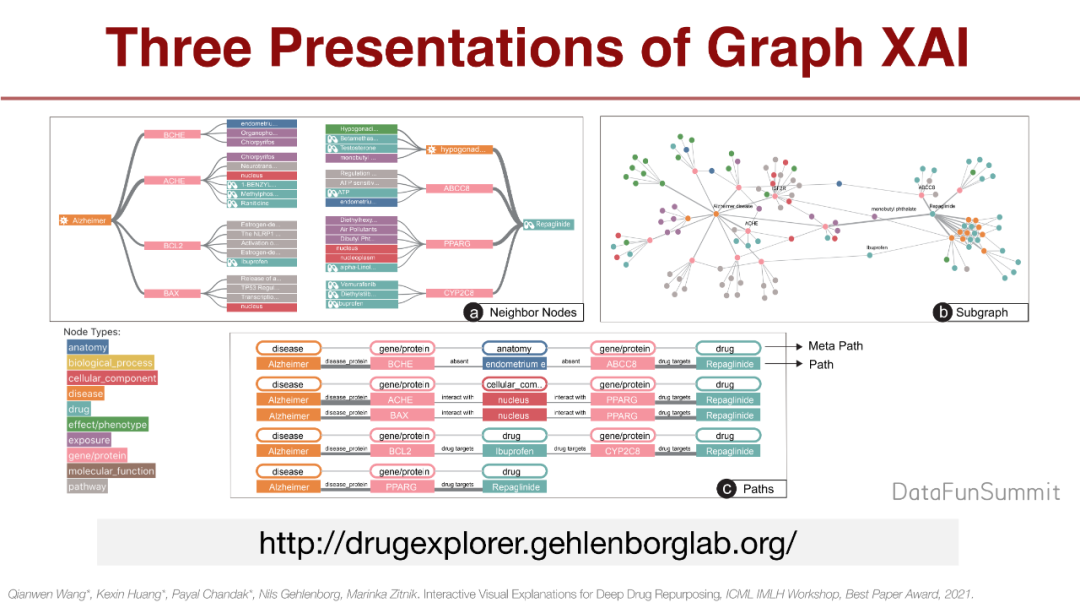
我们最近的工作是和一些专门做HCI(Human-Computer Interaction)的学者合作研究,根据不同的Graph XAI展示给用户。我们提出了3种方法:
Neighbor Nodes:分别提取疾病和药物中重要的基因,逐渐细分并构造树结构。
Subgraph:构造节点之间的知识图,通过去掉或保留边,找到节点之间的连接关系。
Paths:模拟领域科学家思考药物和疾病之间的关系,提取节点之间的路径,可以发现第3种方法更好。如何展示给领域科学家是非常重要的。
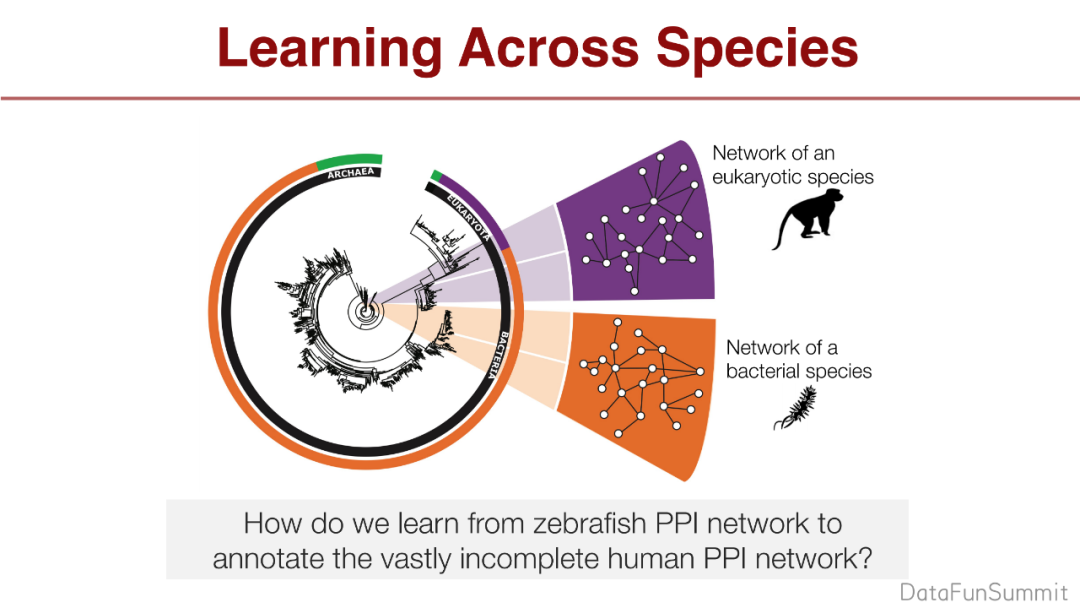
第4部分我想讲一个具体的应用,在少数据量的前提下用图的方法做转化生物医学。
我们最感兴趣的是人类的问题,但是我们在实验的时候不可能直接用于人类,而是使用小白鼠、猴子。如何从其他动物转换到人类身上就是一个重要的问题,因为动物模型和人类模型是存在差异的。
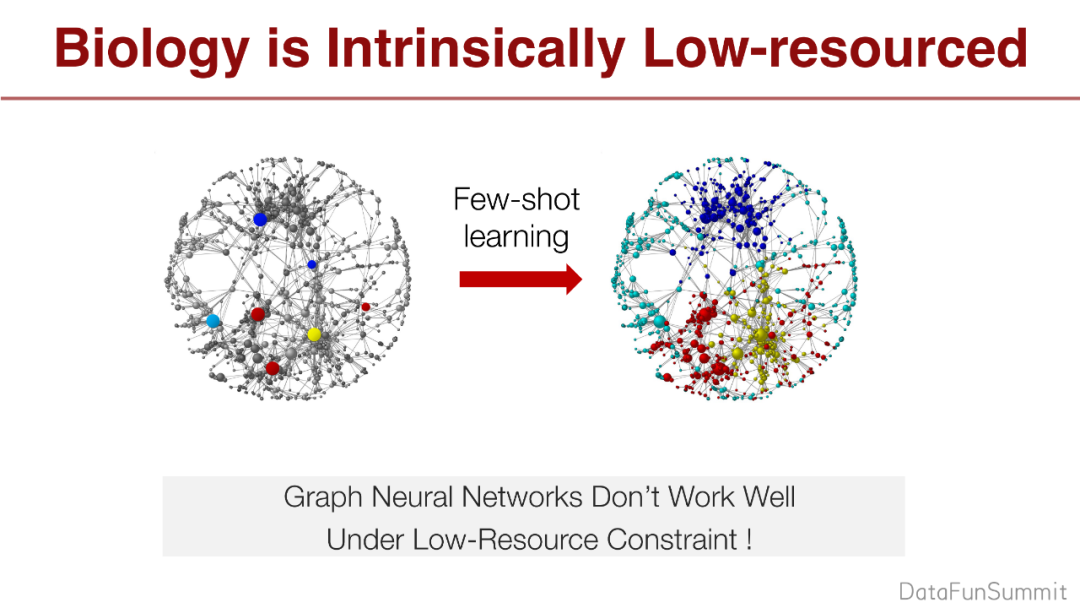
生物是一个少数据的问题,实验也是非常昂贵的,所以Few-shot learning是非常重要的事情。GNN可以获取20%~30%的节点,传统的GNN是无法学习的。
综合刚提到的2个挑战,如何根据少数的标签生成从未见过的图是一个问题。
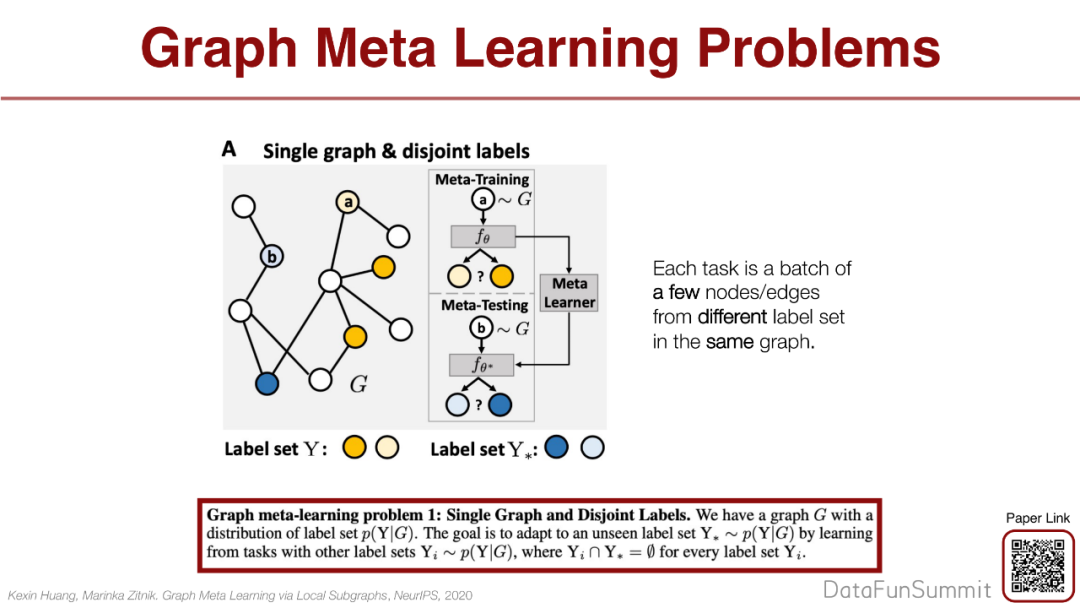
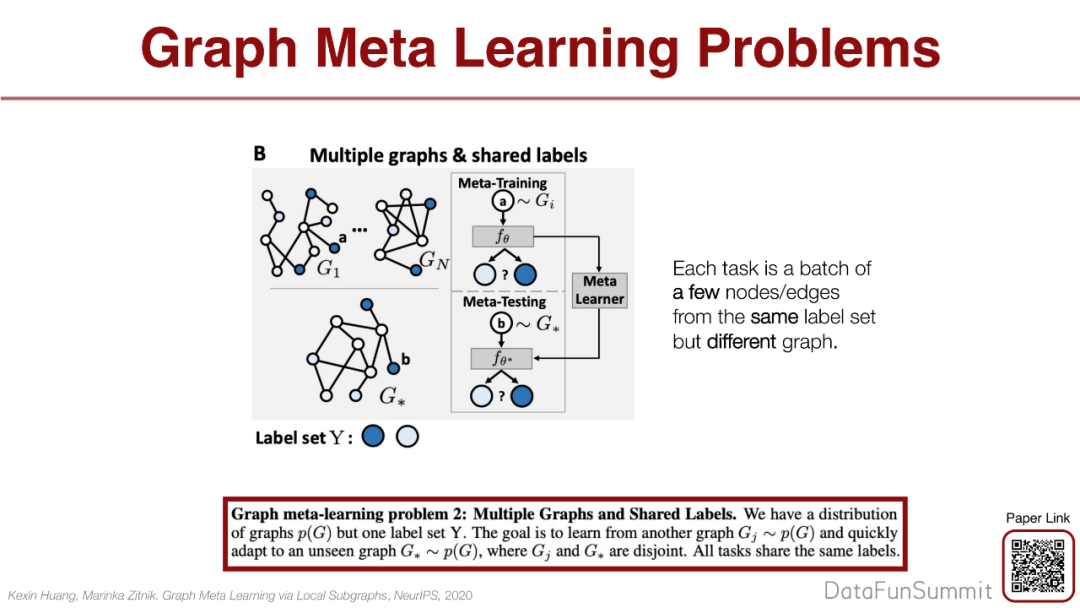
Meta Learning就可以很好地解决这个问题,我们把它抽象成3个问题:
第1个问题:有1个图和不同的label set,如何从已知的label得到未知的label是一个问题。
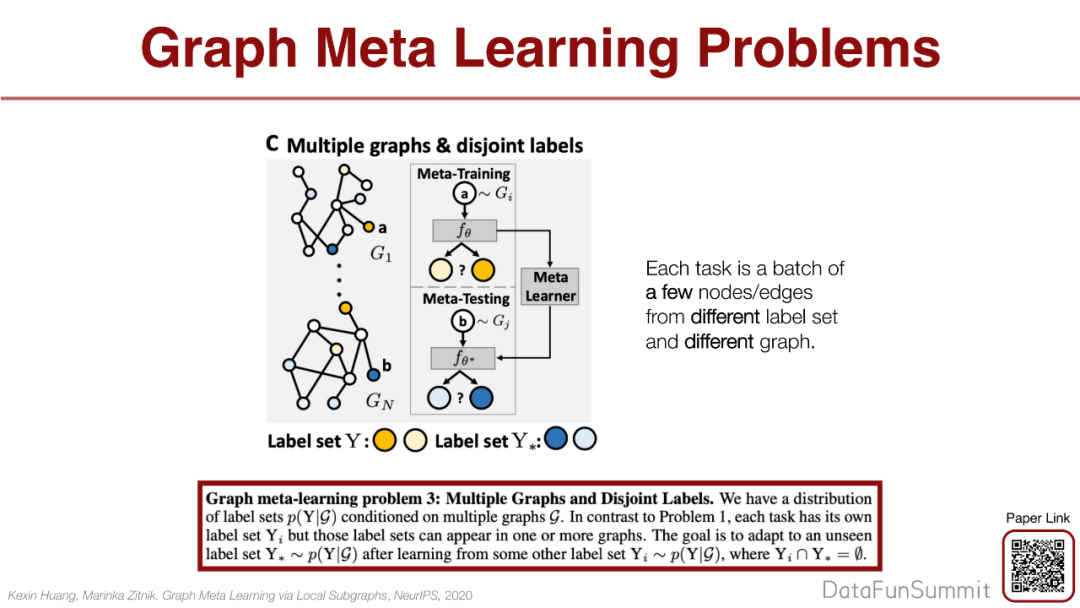
第2个问题:有相同的label和不同的图,如何根据某几种物种的图生成人类的图是一个问题。
第3个问题:是前2个问题的结合体,有不同的图和不同的label。
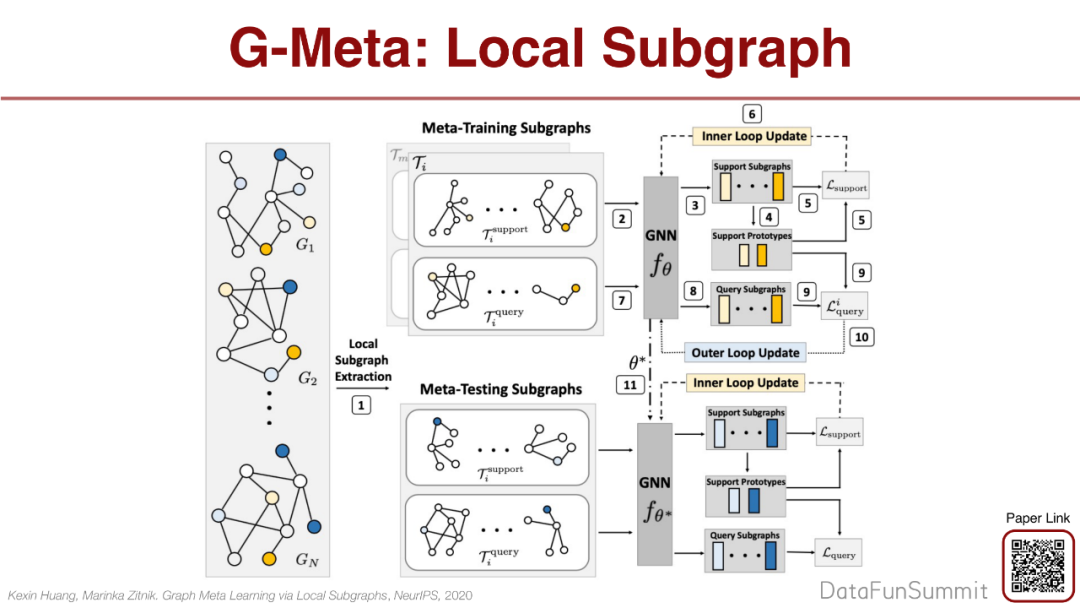
我们提出了一个方法叫G-Meta,用一个简单的思路有效地解决上面的3个问题。已知有不同的图,对于每个节点提取子图,利用子图之间的相似性进行GNN学习。
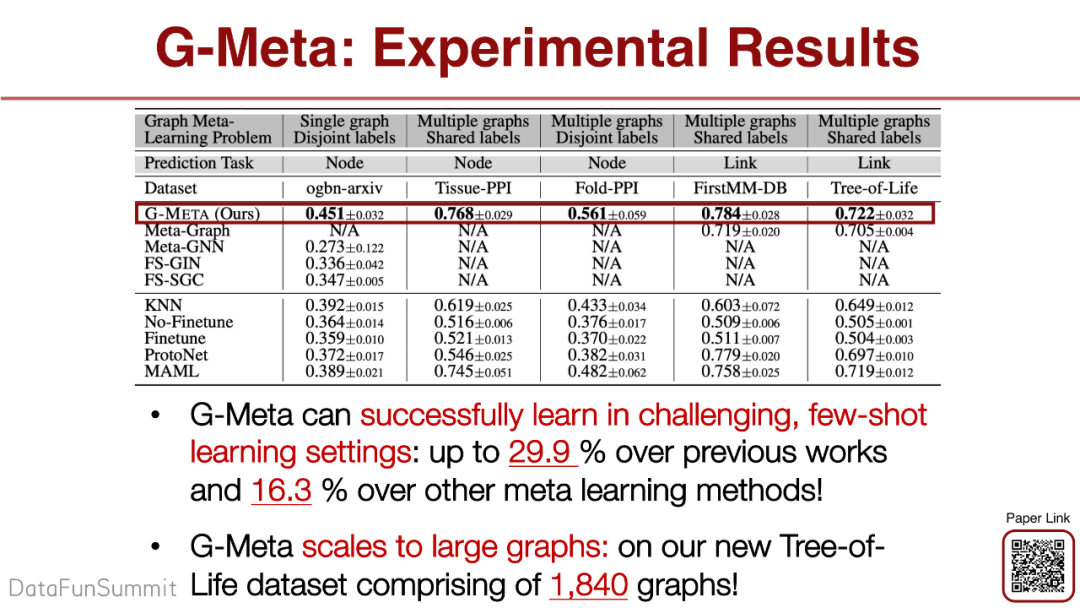
前人的一些方法只能解决一部分的settings,但是我们的G-Meta方法可以解决全部的settings。G-Meta可以适用于非常大的图,因为我们提取子图并且只需要考虑子图这部分。
最后我想讲一下在治疗发现方面我们团队的解决方法。
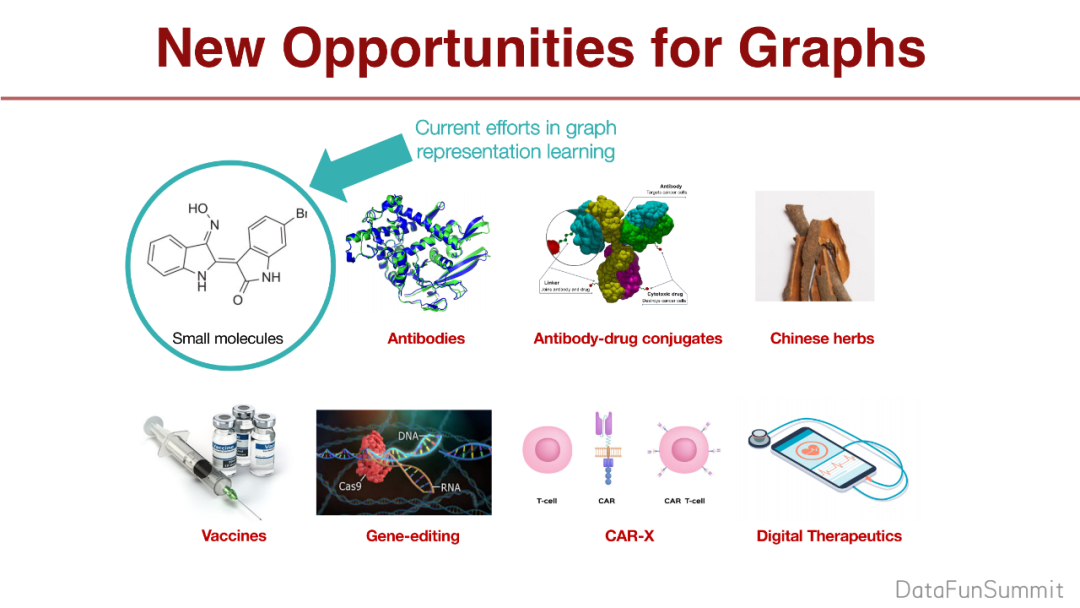
现在小分子药非常多,效果也比较好。但是小分子药的数量逐年在下降,有新的抗体药物能解决一些小分子药无法解决的问题,比如抗体药、中药、疫苗、基因编辑、CAR-X、数字治疗等。
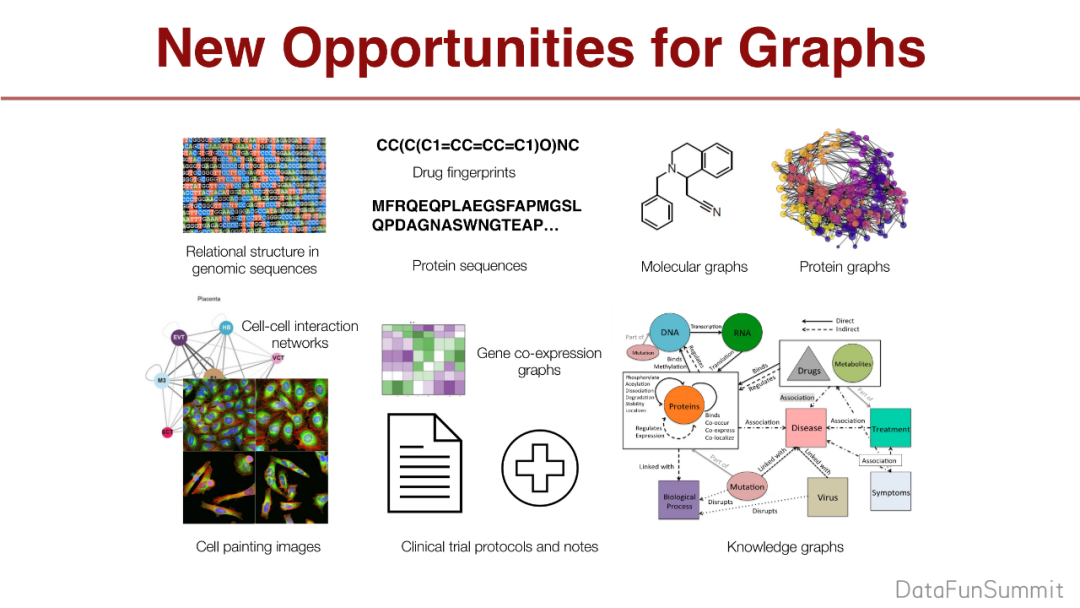
这里具体举一些图的例子,比如:分子图、蛋白质图、细胞关系网、知识图等,非常多的图被使用得很少。
找到有意义的治疗发现的数据集有助于使用上面的图。
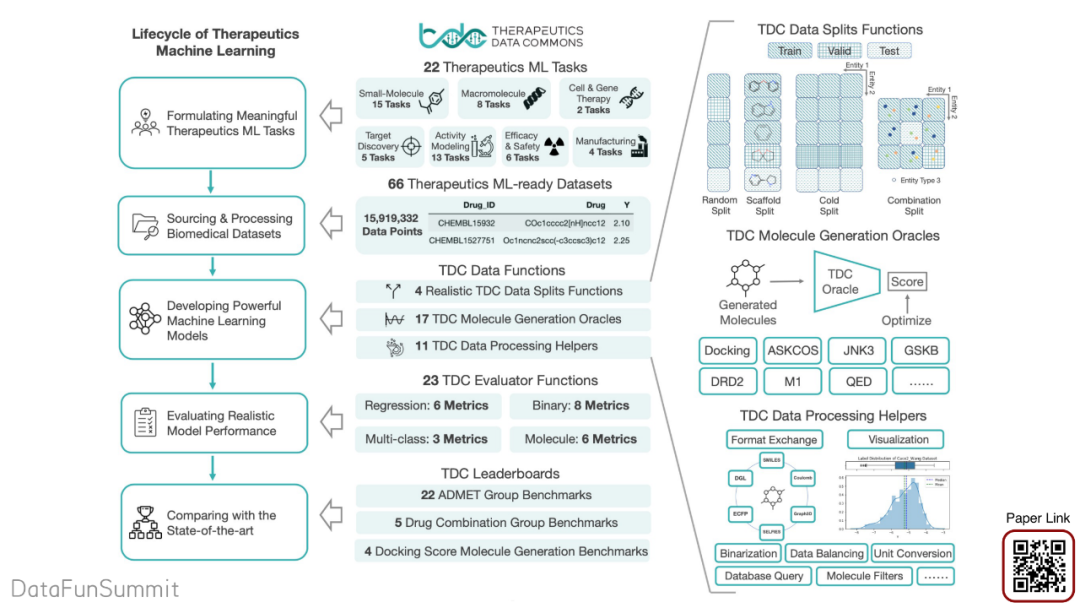
我们最近的工作是治疗学数据共享(TDC)来解决这个问题,TDC有基于22个学习任务的66个learning-ready数据集、开发工具、库、排行榜和社区资源的生态系统,包括数据方法、系统模型评估策略、有意义的数据分割、数据处理器和分子生成预测。
TDC覆盖了治疗学机器学习的生活圈。
我们提供了22个tasks从而解决了制定有效的治疗学机器学习任务。
66个ML-ready数据集包含15919332个数据点,可以直接使用处理好的数据。
训练机器学习模型,我们提供了一些数据方法,如:数据分割、分子生成预测、数据处理等。
评估模型性能。
与最先进的相比。
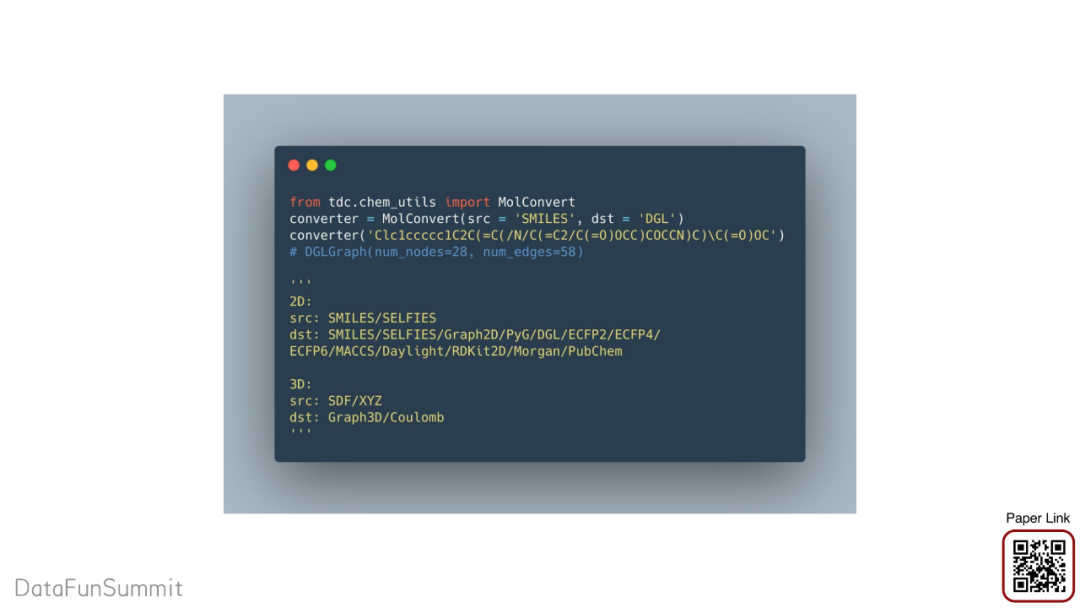
这是一个数据方法的例子,比如想从SMILES转换到DGL的图,用3行代码就可以实现转换。
这是TDC的简介和连接。
今天的分享就到这里,谢谢大家。
分享嘉宾:
OpenKG
OpenKG(中文开放知识图谱)旨在推动以中文为核心的知识图谱数据的开放、互联及众包,并促进知识图谱算法、工具及平台的开源开放。
点击阅读原文,进入 OpenKG 网站。