图预训练技术在生物计算领域的应用
预训练技术简介
图预训练学习化合物表示
图预训练在下游任务的应用
螺旋桨图预训练
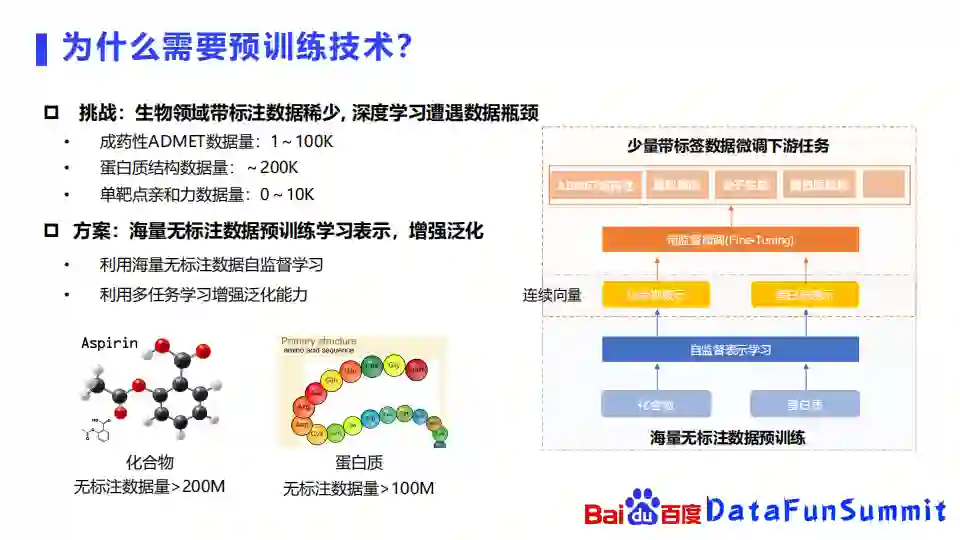
1. 为什么需要预训练技术?
我们发现生物计算领域,特别是药物发现领域,带标注的数据是十分稀少的,比如成药性ADMET的数据量就只有几万的水平,已知的蛋白质结构只有20万左右,靶点亲和力预测的数据大概也只有万级左右的水平,所以在生物领域数据十分稀少,也十分昂贵,这使得我们很难使用在AI领域常用的深度学习算法,尤其是很深的网络。因此我们就会自然而然地想到,能不能借鉴像自然语言处理或者图像处理里面已经比较成熟的预训练的方法,应用在生物计算的领域。恰好在生物领域,化合物无标注数据量超过200M,蛋白质无标注数据量也超过100M。因此,可以使用海量的无标注的生物领域数据去进行预训练,去学习化合物和蛋白质的表示,从而增强化合物和蛋白质的泛化能力。我们利用海量的无标注数据去构造自监督学习任务,通过多个自监督学习任务去同时学习化合物和蛋白质的表示。
上图中的右边这个图显示的是我们使用预训练的一个方式,首先使用海量的无标注数据去进行预训练,学习化合物和蛋白质的表示,然后在含有少量带标签数据的下游任务上进行微调,去学习ADMET,虚拟筛选等等任务。
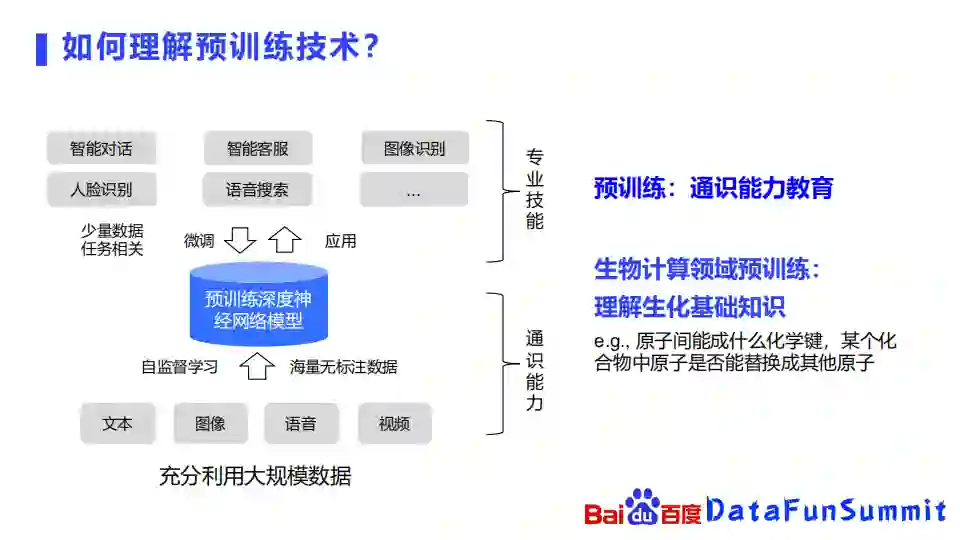
2. 如何理解预训练技术?
在除了生物的其他领域,预训练技术已经比较成熟,它会以文本,图像,语音,视频等作为输入,通过对这些海量的无标注数据去构造自监督的学习任务,去学习这些事物的表示,然后再在更为专业的一些任务上面去进行微调,比如智能对话、人脸识别等更专业的领域去进行学习。换句话说,可以将预训练学习看作通识能力的学习,比如一个小朋友在小学中学的时候,他会去先学语文、数学、英语等比较基础的课程,到了大学才会去学一些比较深的专业知识。在生物领域也是类似的,我们可以将生物计算领域的预训练认为是在理解生化的基础知识,比如两个原子之间,能形成什么样的化学键,或者某个化合物中一个原子是否能替换成其他原子等等。
3. 自监督学习
① 自监督学习
自监督学习任务,要解决的问题是如何去给一个无标注的数据构造label,或者说如何使用监督学习的方法去学习无监督的数据集。最常用的方法,就是用一部分信息去估算另外一部分信息。比如通过过去的信息去预估未来的信息,又或是根据当前的信息去估过去的信息,或者从下面的信息预估上面的信息等等。
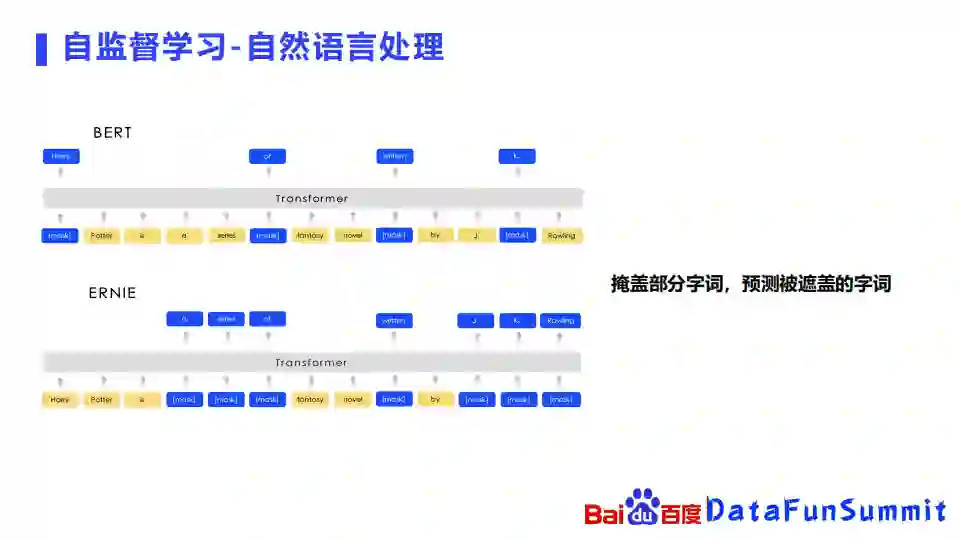
② 自监督学习-自然语言处理
自监督学习在自然语言处理里面用的最多就是masking的方式。自然语言处理里面的输入就是一个句子或者一个字词的序列。最经典的BERT就会将句子序列里面的一些字给遮盖掉,然后估出来被遮盖掉的字词是什么。ERNIE就是百度之前发布的,BERT的进一步延伸,它除了会mask掉一个字,也会mask一个词组,估算出这个词组是什么。
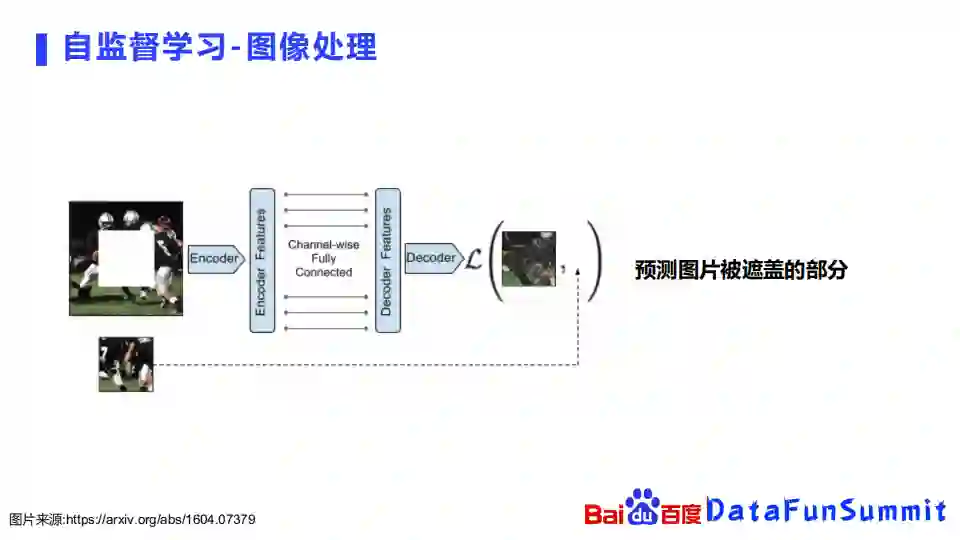
③ 自监督学习-图像处理
在图像处理领域,方法也是类似的。有一张图片,最简单的方式就是随机从图片里面挖空中间的一块,将这张图片给encode,再decode出来它中间被挖空的那一块内容是什么。通过这样的方式,可以构造自监督学习的任务去预测图片被遮盖的部分。
④ 自监督学习-生物计算
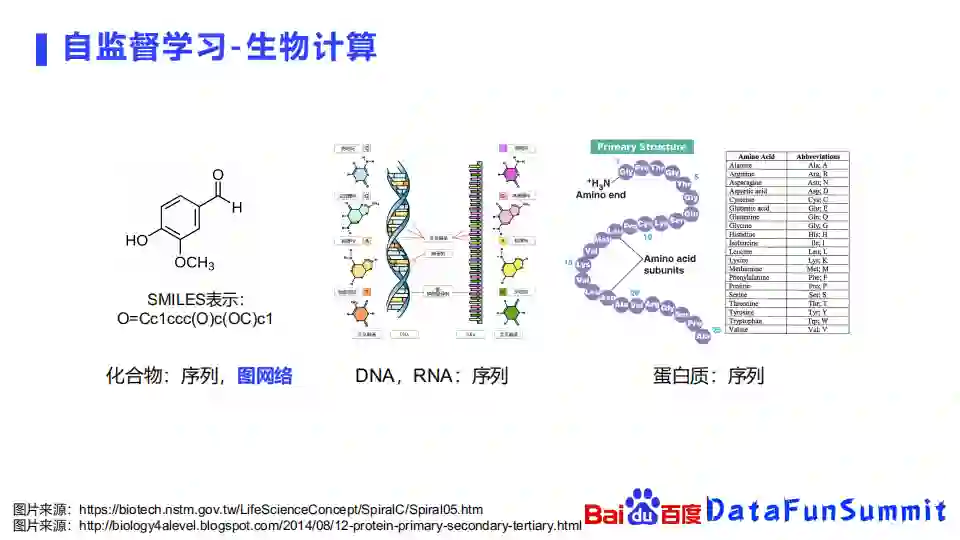
在生物领域,方法也是类似的。生物领域研究的对象,都可以被表示为序列或者图的形式,比如化合物,可以表示成为一个SMILES序列,也可以表示成一个图,DNA、RNA也可以表示成为一个序列的形式,像蛋白质天然就是一个氨基酸序列。我们今天就会重点介绍一下如何使用图网络去学习化合物的表示。
接下来重点介绍图预训练技术学习化合物的表示。
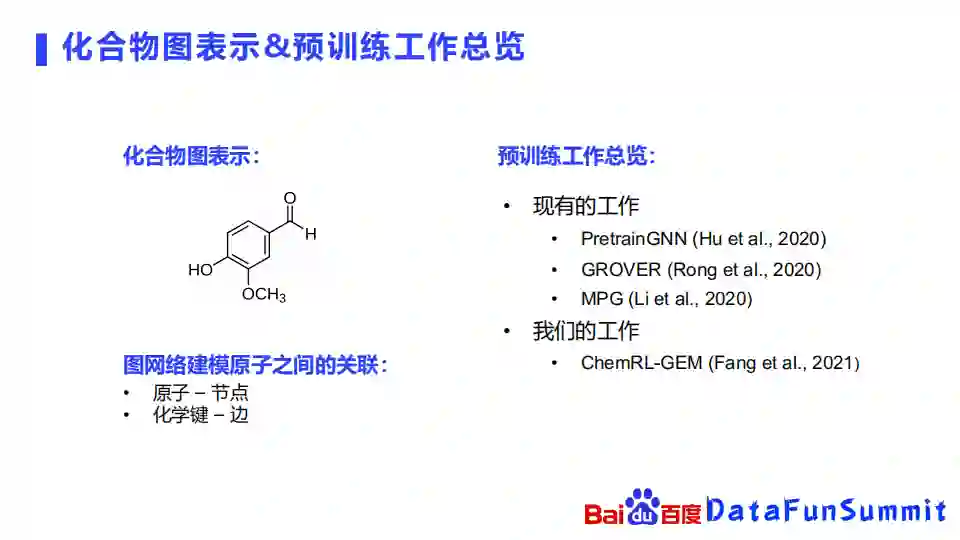
1. 化合物图表示&预训练工作总览
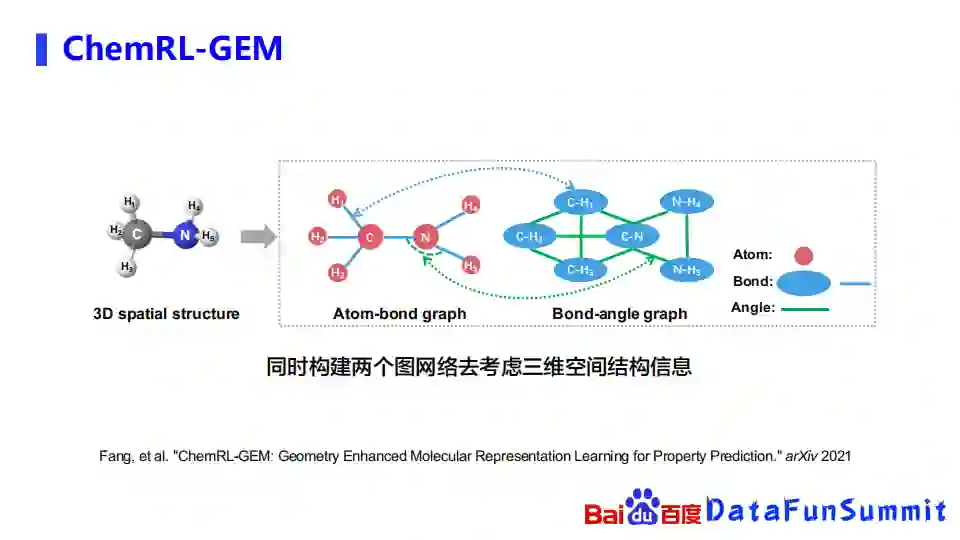
一个化合物,很天然的就可以被表示成一个图。我们可以把一个化合物里面的一些原子就当成是图网络里面的节点,连接这些原子之间的化学键就看成是图网络里面的一些边。通过这样的方式,我们就可以把一个化合物构成了一个图,不同的化合物就能构成非常多的不同的图。现有的预训练工作主要包括PretrainGNN(斯坦福),GROVER(腾讯),MPG(清华&平安)。我们最近也发布了一个叫ChemRL-GEM的模型,利用三维的空间结构去做预训练的表示。
2. 现有图预训练工作
① PretainGNN
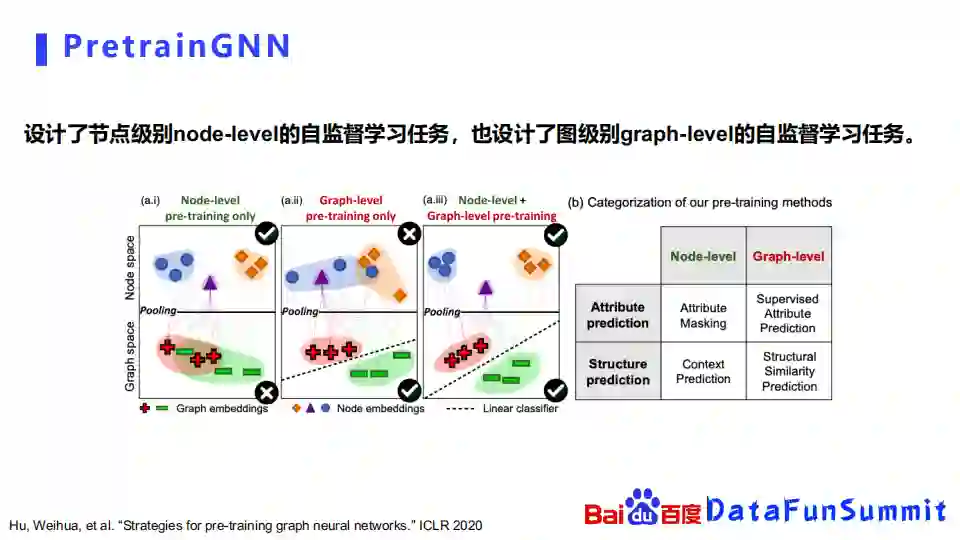
我们先回顾一下最经典的PretrianGNN是怎么做的。它是斯坦福的同学提出的一个方法,他最先提出将这个图预训练用在化合物上面,设计了节点级别node-level的自监督学习任务,也同时设计了图级别的graph-level的自监督学习任务。
如上图左半部分所示,他提出的观点是,如果我们只考虑节点级别的预训练的话,可能在节点级别或是原子级别,能把这些原子的embedding学习得很好,把相似的原子和不相似的原子区分开来,但是在graph embedding上,在整个化合物的表示上,它可能区分的就不是那么好。
比如说,如果我们要判断一个化合物是否具有毒性,我们可能在graphspace上面不太能区分开来。如果走另外一个极端的话,就考虑graph space的自监督学习任务,我们可能能够判断它是不是具有毒性,但是它在节点级别也就是原子级别的embedding,可能就会混在一起,比如说图中正方形和蓝色的圆形就会混在一起。因此他提出需要同时使用node-level和graph-level的自监督学习任务,去同时进行预训练学习。
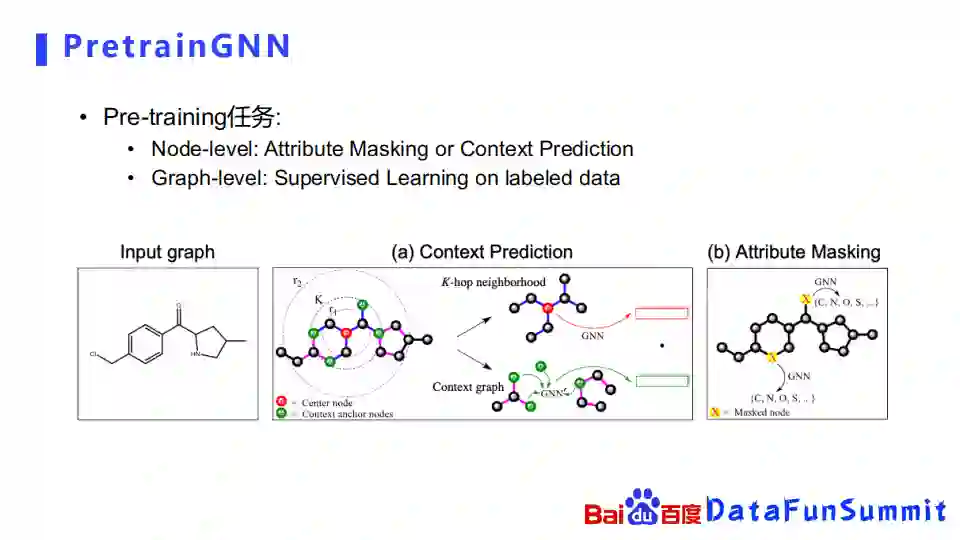
我们先从简单的Attribute Masking开始介绍,我们可以理解为它是直接借鉴了自然语言处理里面的,像BERT里面用到的masking的机制,我们可以把图网络里面的节点和边上面的属性进行masking,要预估它们被masking的属性是什么。比如原子这里有个交叉,我们就可以把它盖住,之后就问被盖住的原子是碳原子还是氮原子,还是氧原子。
他们提出的另外一种node级别的自监督学习任务叫Context Prediction,顾名思义,他是希望去考虑一些上下文的信息。它构造的方式比较特别,首先随便抽取了一个中心节点,要把中心节点附近的K-hop neighbor抽取出来,并且通过一个GNN去encode出它的表示,然后另外一方面,它把这些绿色的原子附近的原子也给一起拿出来了,并且通过GNN去学习它的表示,对比这两个表示的区别。通过这样的方式希望学习到化合物的一些上下文的信息。
在graph level的层面上,他们是通过从一些开源数据集上面找了一些带标签的数据,去构造了一个自监督学习任务。
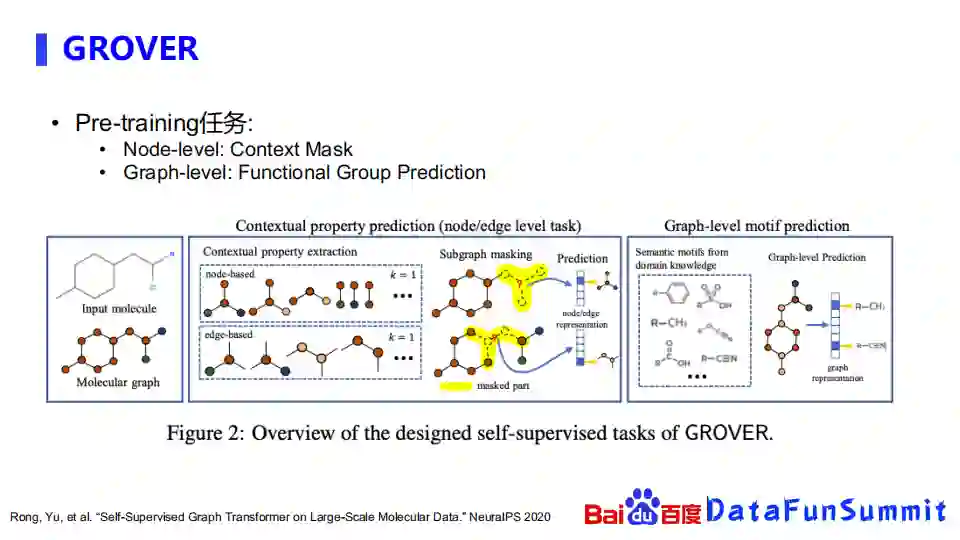
② GROVER
GROVER是腾讯之前的工作。他们是在PretrainGNN的基础上做了一些改进,在自监督学习任务上,从边或者是点的masking扩展到subgraph的masking。对于node-based, 首先随便抽取一个中间的节点,并且获取1-hop,例如邻居结点,然后把整个子图盖住,再去预估被盖住的这个子图是什么。edge-based也是类似的, 随机抽取了一条边,然后要把它周围的子图抽取出来,再去估算被盖住的子图是什么。在graph-level上,他们提供了一种比较新颖的方式,通过对graph进行表示之后,预测graph里面包含了哪些子结构,比如包含哪些功能团。
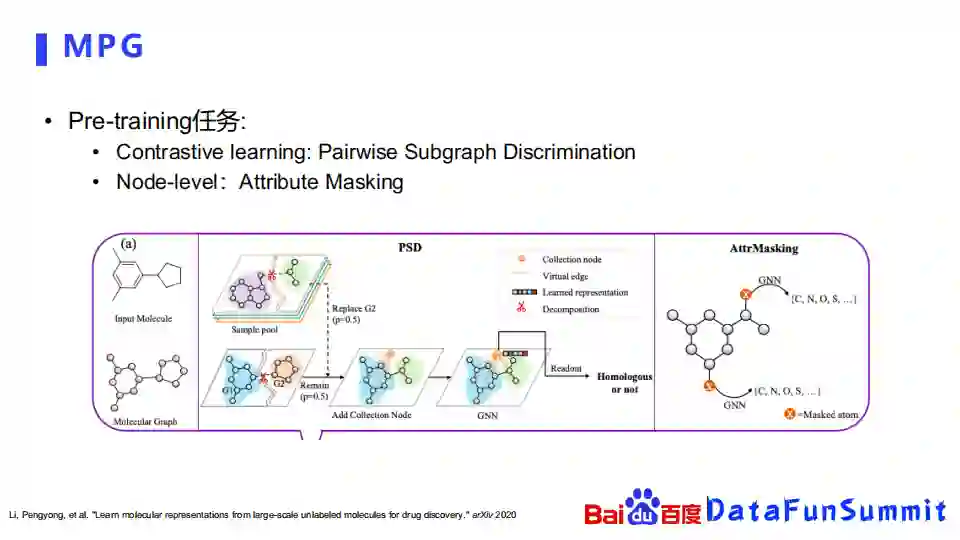
③ MPG
最后介绍一下清华和平安的工作,他们是通过一个对比学习的方式去学习化合物的表示。比如他们会随机的把一个化合物切成两块,像这里绿色的一块和紫色的一块,另外的化合物也是类似地剪成蓝色的一块橙色的一块,然后把有一定概率会来源于两个化合物的内容给拼接起来,再去判断,重新拼接出来化合物是不是由两个不同的化合物拼起来的,还是两个模块都是来源于同一化合物的,通过这样对比学习的方式去构造自监督学习任务。他们使用的另外一个自监督学习任务,也是之前介绍过的Attribute Masking,就是随机掉某一些属性,然后再去预估。
④ 现有工作的问题
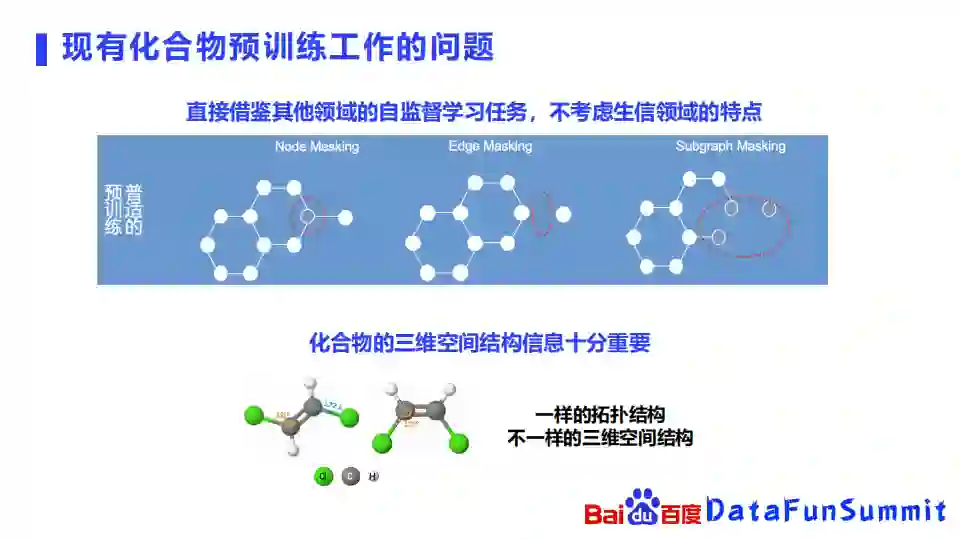
我们先总结一下我们刚刚介绍的三种化合物预训练工作的一些问题,我们可以发现其实他们都是直接借鉴了比较成熟的自然语言处理、图像处理领域的自监督学习任务的方法,但是没有太考虑生物领域的特点。像node masking、edge masking或者subgraph masking的方法,其实都是盖住了化合物里面的某个节点,或者一条边,或某个子图,然后估算出他们是什么。
但是在生物领域,三维空间结构信息是十分重要的。比如上图中的例子:包含了两个化合物,也就是两个分子,我们可以看到这两个分子在图的拓扑结构上是完全一样的,比如说它的中间骨架都是由两个碳原子连接而成的,每个碳原子都是连接的一个氢原子和一个氧原子。但是如果你从三维结构上去看,它们是不一样的,左边这个分子里面原子的角度是121度,右边的键角是125度。此外在左边这个分子中,两个氢原子的朝向是不一样的,而右边这两个氢原子朝向相同,因此即使是一模一样的图的拓扑结构,它们可能也会有着完全不一样的三维空间结构。
3. 我们的图预训练工作
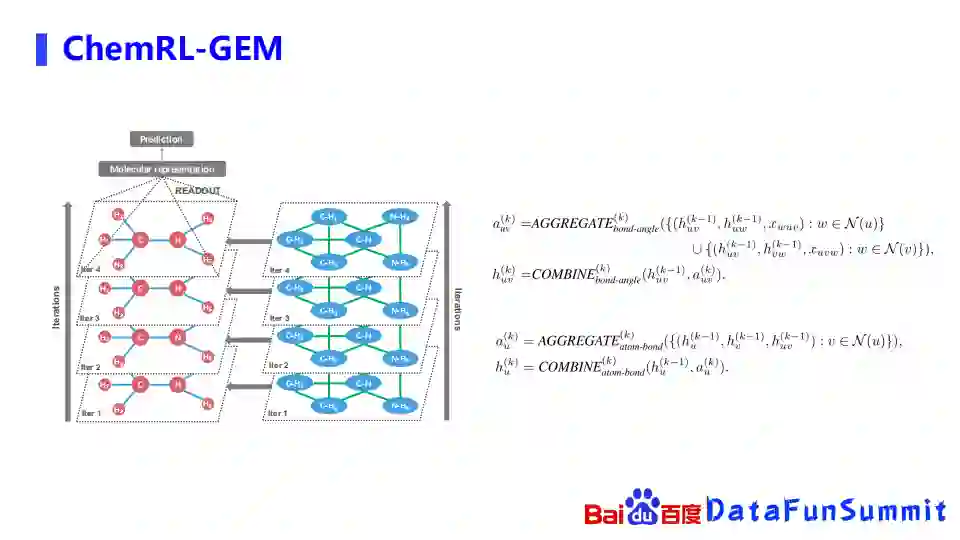
鉴于此,我们使用两个图网络去考虑三维空间结构的信息,比如在最左边的图中,假如我们先拿到化合物的空间结构,构造两个图,第一个图是经典的原子与化学键之间关系的图,原子之间用一个化学键相连。第二个图是我们新加的图,它的节点就不再是一个原子,而是一个化学键,比如蓝色边化学键到右边的图里面就成了一个节点,左边图里面化学键之间的键角,到右边的图里面就是一条边。通过这样的方式,我们希望从原子化学键和键角相互之间的关系,去捕获一些三维空间结构的信息。
在图网络上,我们先把右边这个化学键和键角组成的图网络进行消息的传播,信息赋给左边的以原子和化学键组成的图网络,每一层传递上去之后,再去左边的原子和化学键组成的图网络抽取信息,再进行下游任务或预训练任务的一些prediction。
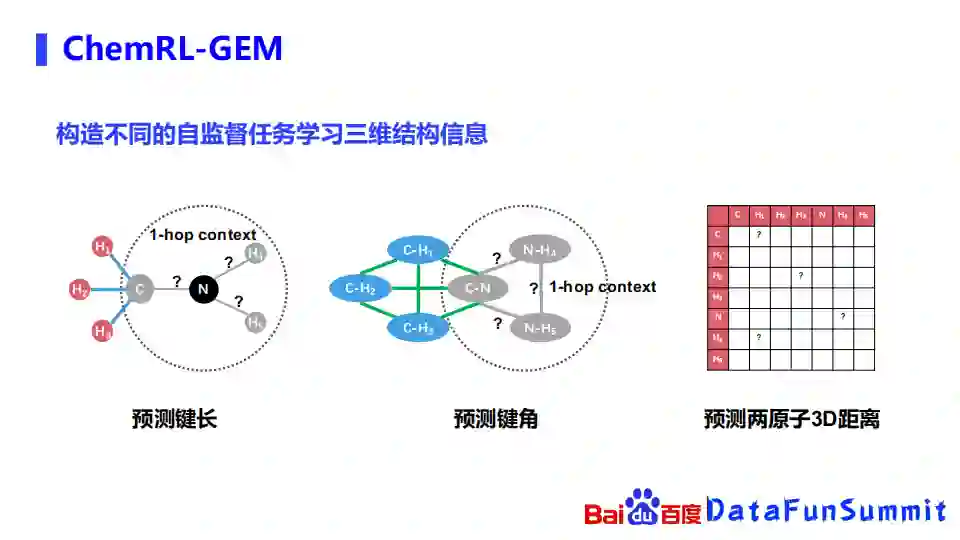
除了网络结构,最重要的是希望通过构造一些自监督学习任务,从海量无标注的数据里面学习到一些基础的化学知识。我们构造了不同的自监督任务去学习三维信息,比如左边的第一个任务,首先我们会随机的选取其中的一些原子,把它以及它的1-hop盖住,预估以这些原子为中心的键长是多少。第二个任务也是类似的,我们也是以某个原子为中心,把它相连的一些化学键和键角都盖住,预测这些键角是什么。左边这两个预测键长键角的任务,都是当成一个回归任务去进行预测。
右边这个任务是预测两个原子之间的3D距离,也就是说预测两两原子之间的距离是多少。因为存在两个分子是同分异构体的情况,即它虽然有同样的拓扑结构,但是他们的三维空间结构是不一样的,因此我们使用了分类的方式去进行学习。此外,如果两个原子的距离比较远,可能用回归的方式比较难以捕捉,因此我们也是使用分类的方式。
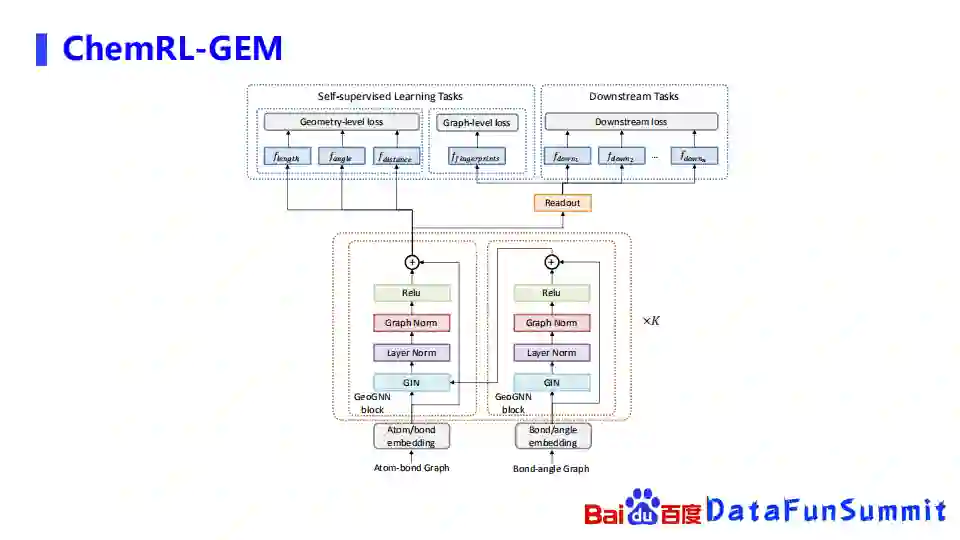
上图是ChemRL-GEM的总示意图,可以从下面开始看起。我们首先构造了原子和边的一个网络,还有边和键角的一个网络,左边是图网络的结构,右边是边和角度的图网络结构,右边的图网络的信息,在每一次聚合完以后就会传导到左边的图网络,然后左边的图网络在经过多层聚合好所有的信息以后,就会使用基于三维空间结构的自监督学习任务去进行学习,右边在Readout之后会进行下游任务的学习。
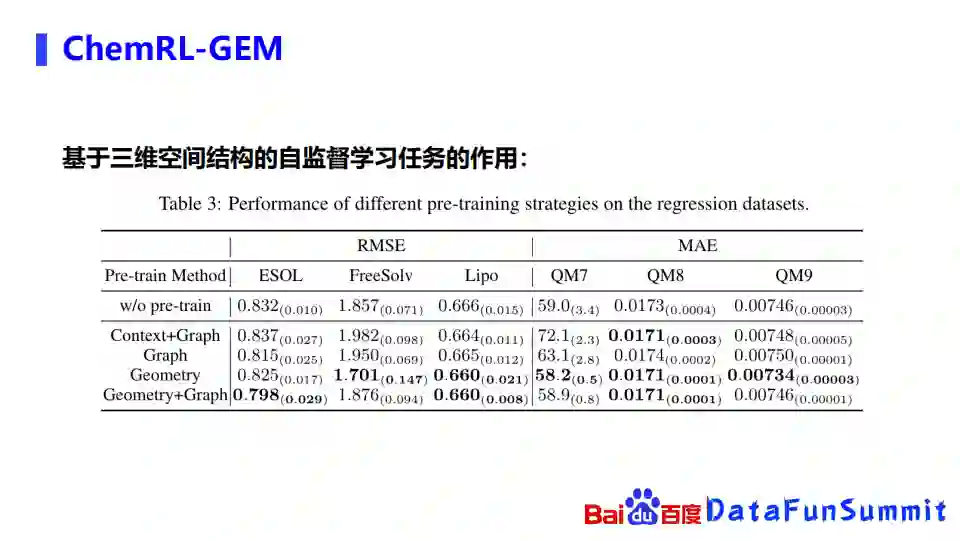
我们使用了molecular net里面的12个batch mark进行实验,发现ChemRL-GEM在11个数据集里面都是取得了SOTA的效果。
这些数据集包含的类型比较多,这里简单介绍一下,比如这里的数据集可能是一些水溶性的数据集,右边QM7到QM9是一些量子化学的数据集。下面这些分类任务的数据集,比如BBBP是一个血脑屏障的数据集。从整体上看,可以发现因为回归任务整体上是和三维的空间结构关系更为密切了,所以在这些任务上面的提升也更为明显。
针对基于三维空间结构的自监督学习任务,我们也分析了一下它的作用,做了一个performance study,发现基于空间结构的自监督的学习任务,在整体上也确实是比没有使用基于空间的自监督学习任务的方式取得了更好的效果。
首先是成药性ADMET的预测: 一个化合物,是不是能成为一款药物,其实是受到很多限制的,比如说这里的成药性。一款化合物被吃到体内以后,我们首先得确定它是不是能被胃吸收,如果能被胃吸收,我们还要确定它是不是能被运输到指定的器官脏器,以及它是不是能被代谢,能不能被排出体外等等。
最重要的就是它是不是具有毒性,如果有毒性,即使它具有生物活性也是不能成药的。我们是基于预训练技术去训练了一个ADMET模型,预估了多达50多项的指标,我们的评测准确率超越现有ADMET系统大概4%左右,我们的服务已经上线到了一个计算服务平台,用户除了可以直接去使用以外,也可以使用一些自由的数据去基于预训练模型进行一些finetune,得到一些定制化的模型。
此外,我们也在今年的三月份去参加了一个OGB的两个化合物属性相关的榜单的打榜,其中一个是化合物是否能够抑制HIV病毒,以及针对100多种疾病靶点的活性预测。当时是在榜单的第一名。在今年的六月份我们参加了KDDC的化合物的量子化学性质的任务,也取得了第二名,击败了DeepMind等众多团队。
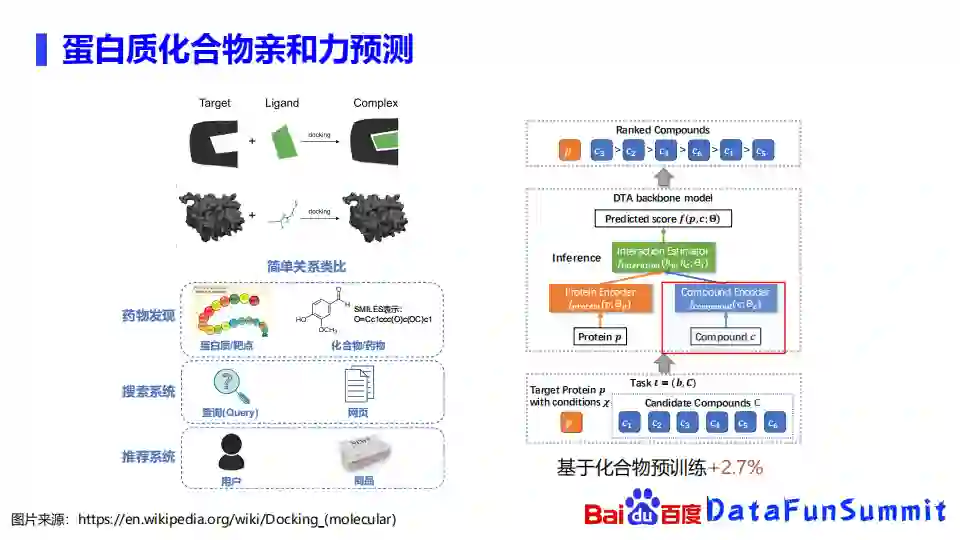
说完化合物的属性预测,我们再介绍一下第二块应用,就是蛋白质化合物的亲和力预测。大家都知道亲和力的预测对于药物发现是十分重要的,其实可以做虚拟筛选,那么什么是蛋白质化合物的亲和力预测呢?
我们可以把蛋白质靶点就当成是这一块拼图,或者说积木里面带凹槽的一块记录,而化合物或者说一个配体就当成是这样的一块绿色积木,我们要判断的就是这两块积木能不能被拼在一起,能不能被咬紧,或者说它能拼接的有多紧密。
如果我们把药物发现,和搜索、推荐系统去类比,就可以发现,其实药物发现里面的靶点蛋白质就和搜索系统里面的查询或推荐系统里面的用户是相似的,药物发现里面的化合物药物和搜索系统里面的网页或者推荐系统里面的商品也是类似的。简单来说就是能不能根据蛋白质靶点的信息,从海量化合物里面筛选出来合适的一些化合物作为潜在的药物。因为蛋白质化合物亲和力的预测涉及到蛋白质,那么化合物的表示部分,我们同样也可以用化合物的预训练去进行表示的增强。我们的实验也发现其化合物的预训练能提升大概2.7%的效果。
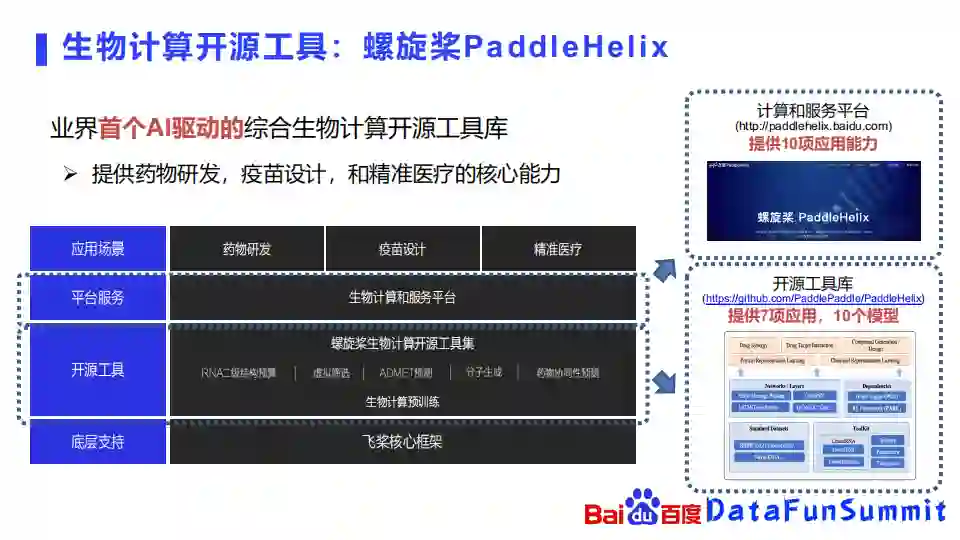
最后,来介绍一下螺旋桨图预训练技术的一些应用。PaddleHelix是一个以AI驱动的综合的生物计算的开源工具库,提供药物研发,疫苗设计和精准医疗的核心能力,主要包含了开源工具和平台服务两大块内容。开源工具已经在去年年底在github上线,目前启动了大概七项应用和十个模型,平台服务目前也提供了十项应用的网站,欢迎大家去体验。
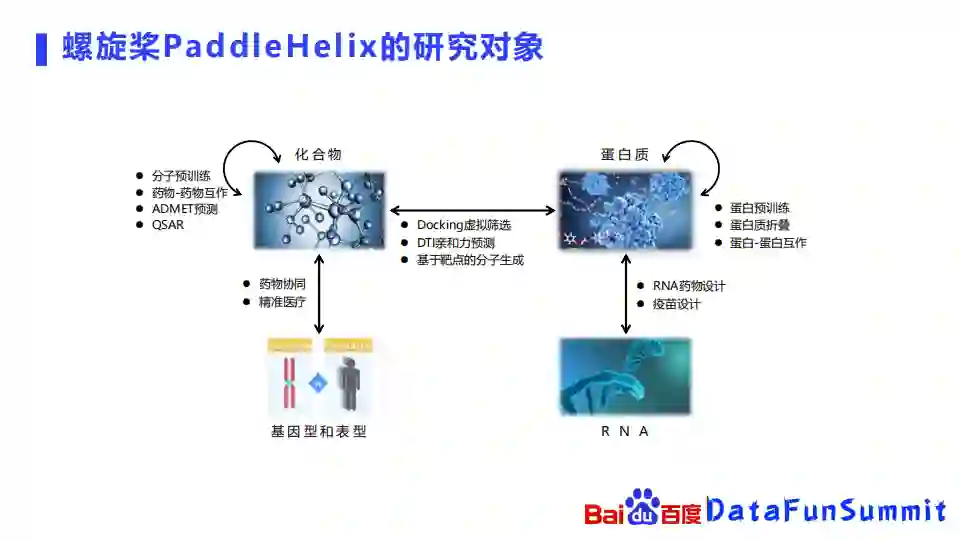
PaddleHelix的主要研究对象包括蛋白质化合物、基因型表型、RNA以及它们相互之间的一些关系。
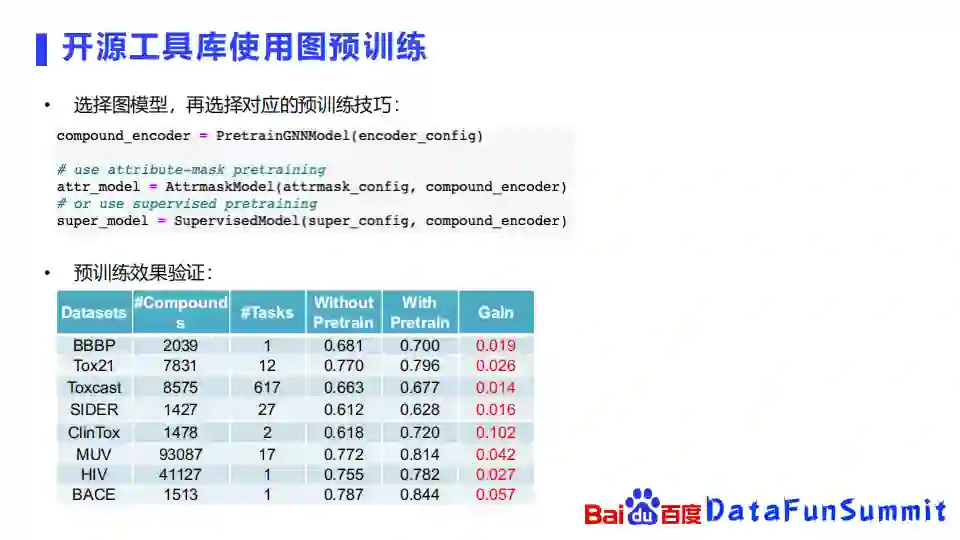
接下来介绍一下如何使用PaddleHelix预训练技术去学习化合物的表示。PaddleHelix提供了一些便捷的特征、抽取工具和模型预测的接口,能够把化合物直接转成图结构并进行预测。
比如上图中的例子,我们可以先定义需要抽取的原子和化学键的一些特征,塞到一个特征抽取器里面,然后就可以得到一个分子的图,再把这个图放到网络里面,就可以直接进行化合物的属性预测了。
此外,在组网方面,我们也提供了非常简易的组网工具,可以直接通过配置文件去快速组网,比如想选用gnn,只要在gnn_type选一下gin,然后dropout_rate选择0.5等等,就可以通过几个配置来快速组网。
有了这些网络以后,我们可以再选择对应的预训练技巧,比如刚刚提到的pretrainGNN上面的一些预训练技巧,我们可以选用不同的自监督学习任务去进行学习。我们的验证也发现,使用预训练的方式确实在许多的下游任务上取得了不错的表现。
在计算和复合平台上面,我们提供化合物属性预测的Finetune功能,可以在网站上面选择要训练新的模型,再选择使用回归算法还是分类算法,上传相应的一些数据,就可以用计算平台上的算力去进行训练,从而得到自己的一些属性预测的模型。
让我们来回顾一下本次分享的内容,首先介绍了预训练技术的概念及必要性,然后分别介绍了已有的PretainGNN、GROVER、MPG的特点和不足,针对这些问题,我们提出了ChemRL-GEM网络模型。接着介绍了图预训练在下游任务中的应用,最后展示了百度开发的生物计算开源工具PaddleHelix的具体应用。希望今天的分享能为大家带来一些启发和帮助。
Q:在蛋白质结构预测领域,图预训练有什么好的研究进展吗?
A:这个问题很好,其实之前在虚拟筛选领域,有一些工作已经是把蛋白质表示成图的形式,但是应用不多。但是已有许多把蛋白质用作序列的预训练的工作,比如由Berkley主导完成的TAPE,或者说像facebook的ESM-1b的工作,都是做蛋白质预训练的问题,但它是基于序列的,目前还没有基于图的方法。基于图的话会有一些问题,例如首先需要知道两个氨基酸之间的距离,但是目前无法知悉这个距离,因此基于图做预训练目前存在一些困难。
Q:ChemRL-GEM中的3D空间结构是怎么获取的?
A:现在是用RDKit去获取的,可能也有同学会问,RDKit获取到的空间信息其实是比较不准的,确实它也是跟真实的空间结构有一点差距,但我们也发现,即使是用这种不准的空间结构,在预训练上面或是在网络结构上面也有了不错的效果。此外,我们在paper里面重新做了一个实验,使用了准确的3D结构,模型效果更为明显。
在文末分享、点赞、在看,给个3连击呗~
分享嘉宾:
🧐分享、点赞、在看,给个3连击呗!👇