哈佛大学|构建知识图谱PrimeKG以实现精准医疗--数据与代码全部公开,帮你从零开始复现知识图谱
近日,哈佛大学团队在bioRxiv上发表了题为“Building a knowledge graph to ennable precision medicine"的文章,构建了PrimeKG知识图谱用于发现新的生物标志物、表征疾病过程、完善疾病分类、识别表型特征、预测生物学机制和药物重利用等,并公开了所有的数据与代码,帮你从零开始复现知识图谱。
1 PrimeKG的独特功能
疾病覆盖广:PrimeKG包含17000多种疾病。PrimeKG中的疾病节点与图中的其他节点紧密相连,并针对下游精准医学任务中的临床相关性进行了优化。
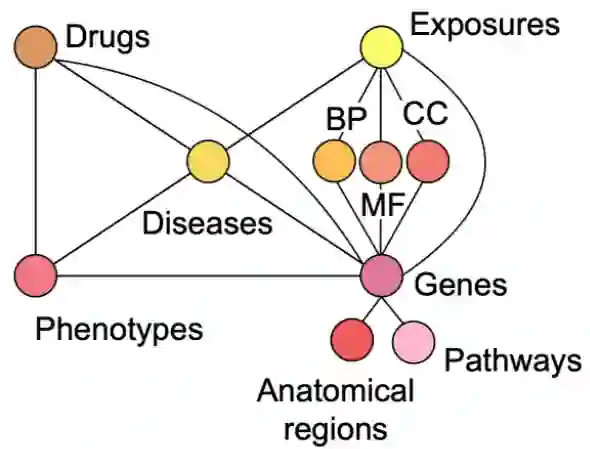
异构知识图谱:PrimeKG包含了分布在各种生物尺度上的超过100000个节点,如下图所示。PrimeKG还包含29种边,这些节点之间的关系超过400万个。
临床知识的多模式整合:PrimeKG中的疾病和药物节点增加了来自Mayo Clinic、Orphanet、DrugBank等医疗机构的临床描述。
即用型数据集:PrimeKG对外部包的依赖最低。知识图可以从Harvard Dataverse以即用型格式检索。
数据功能:PrimeKG提供了广泛的数据功能,包括用于主要资源的所有预处理包和用于构建更新的知识图谱的脚本,帮你完全复现知识图谱。
项目网站:https://zitniklab.hms.harvard.edu/projects/PrimeKG
代码地址:https://github.com/mims-harvard/PrimeKG
数据地址:https://doi.org/10.7910/DVN/IXA7BM
2 方法
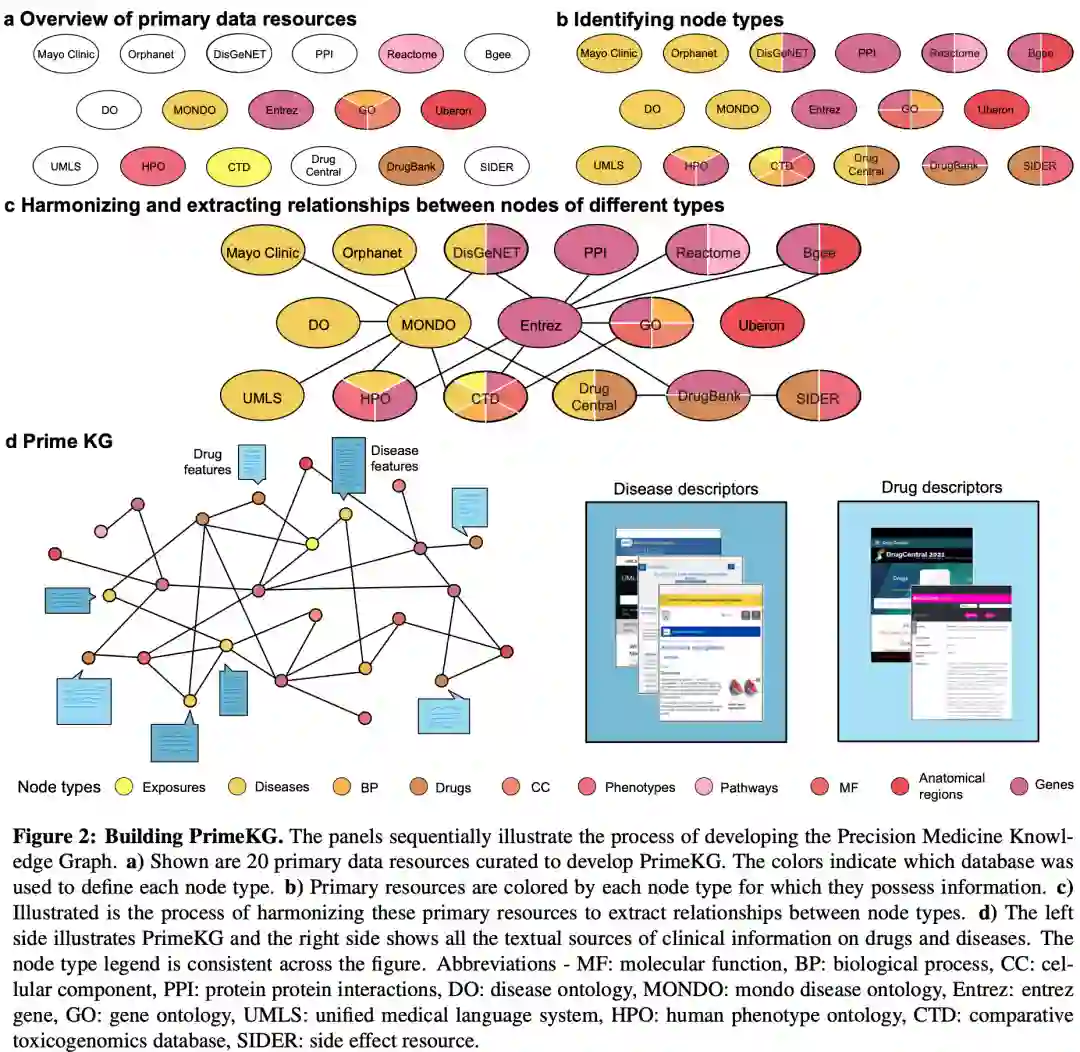
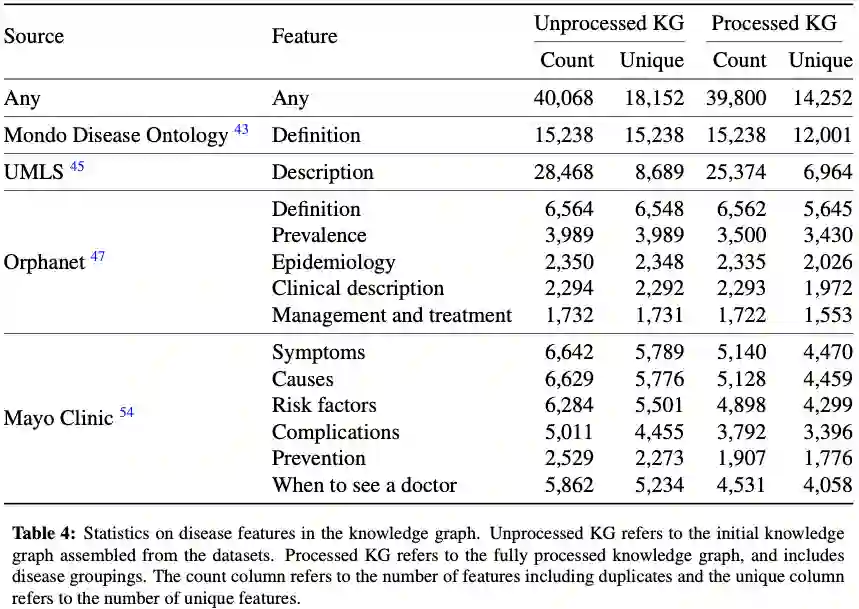
精准医学知识图谱(PrimeKG) 是异构的,有10种节点和30种无向边。为了开发PrimeKG,作者检索并整理了20种主要数据资源,如图a所示,确定了这些关系,图b和c所示,将它们协调成一个丰富的异构网络,如图c所示,增强了药物该网络中的疾病节点和文本描述如图d所示。
A 整理原始数据资源
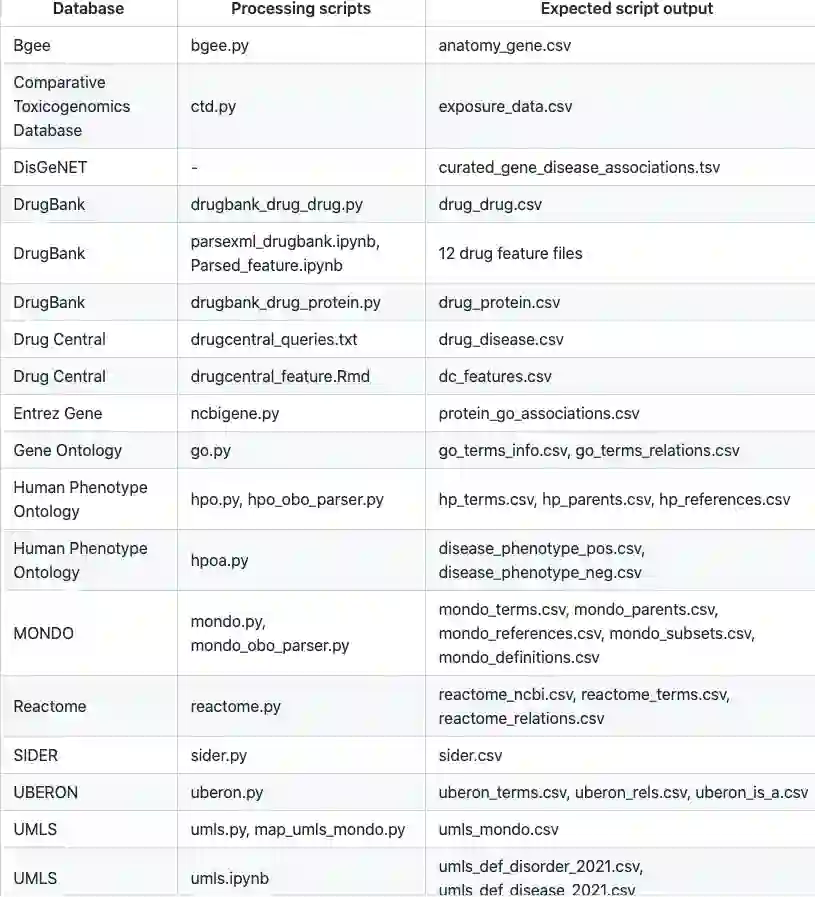
为了开发一个全面的知识图谱来研究疾病,作者考虑了20个主要资源和一些额外的生物和临床信息库。作者选择了广泛覆盖生物医学实体的数据集,包括蛋白质、基因、药物、疾病、解剖学、生物过程、细胞成分、分子功能、暴露、疾病表型和药物副作用。这些高质量的数据集,要么是专业策划的注释,例如DisGeNet和Mayo Clinic,要么是广泛使用的标准化本体,例如Mondo疾病本体,要么是直接读出的实验测量值,例如Bgee和DrugBank。数据记录部分列出了主要资源的完整列表及其处理步骤。所有的数据管理和处理方法都是透明的,完全可重复的,并且可以随着单个数据资源的发展和新数据的可用而不断调整。
B 标准化和统一数据资源
为了将这些主要数据资源整合到精准医学知识图谱中,作者为每种节点类型选择了本体,将数据集处理为标准化格式,并解决了本体之间重叠的问题。
定义节点类型和选择通用本体
作者的知识图由 10 种类型的节点组成。节点类型“药物”、“疾病”、“解剖”和“通路”分别用 DrugBank、Mondo、UBERON 和 Reactome 中的编码为术语。基因和蛋白质被视为单一节点类型,“基因/蛋白质”,并由 Entrez 基因 ID 识别。节点类型“生物过程”、“分子功能”和“细胞成分”是使用基因本体术语定义的。从 HPO 中提取的疾病表型和从 SIDER 中提取的药物副作用被合并成单个节点类型,即“效果/表型”,使用 HPO ID 进行编码。最后,使用 ExposureStressorID 字段定义“暴露”节点,该字段包含Comparative Toxicogenomics Database提供的 MeSH 标识符。
整合外部数据资源
作者映射了上述处理过的数据集,以确保所有节点都在其各自的通用本体中定义。接下来,作者为每种节点类型确定了跨不同主要资源的信息源,以最大化 PrimeKG 中的关系数量。然后作者重组数据集以遵循以下格式。对于知识图中的每个节点,作者提供“节点索引”,这是一个唯一索引,用于标识 KG 中的节点;'node_id' 表示节点在其本体中的标识符;“节点类型”表示知识图中定义的节点类型;'node name' 表示本体提供的节点名称;和“节点源”,它指示从中提取“node_id”和“节点名称”字段的本体。对于知识图中的每条边,提供“关系”,它是连接两个节点的边类型的名称;链接到“节点索引”字段的“x_index”;和“y_index”,它也链接到“node_index”。最后,为了保持一致性,重命名了列,删除了具有 NaN 值的行。
解决了表型和疾病节点之间的重叠
C 构建精准医学知识图谱(PrimeKG)
为了构建PrimeKG的网络结构,作者将整合后的原始数据资源合并到一个图中,并提取它的最大连通分量。整合了各种经过处理、策划的数据集,并通过删除 NaN 和重复边、添加反向边、再次删除重复和删除自循环来清理图形。此版本的知识图在我们哈佛数据宇宙中心以kg raw.csv获取。为了确保我们的知识图连接良好并且没有任何孤立的口袋,我们提取了它的最大连接组件。知识图的最大连接组件可在我们的哈佛数据宇宙中以“kg Giant.csv”的形式获得。
D 用临床信息补充药物节点
E 用临床信息补充疾病节点
3 技术验证
表征精准医学知识图谱
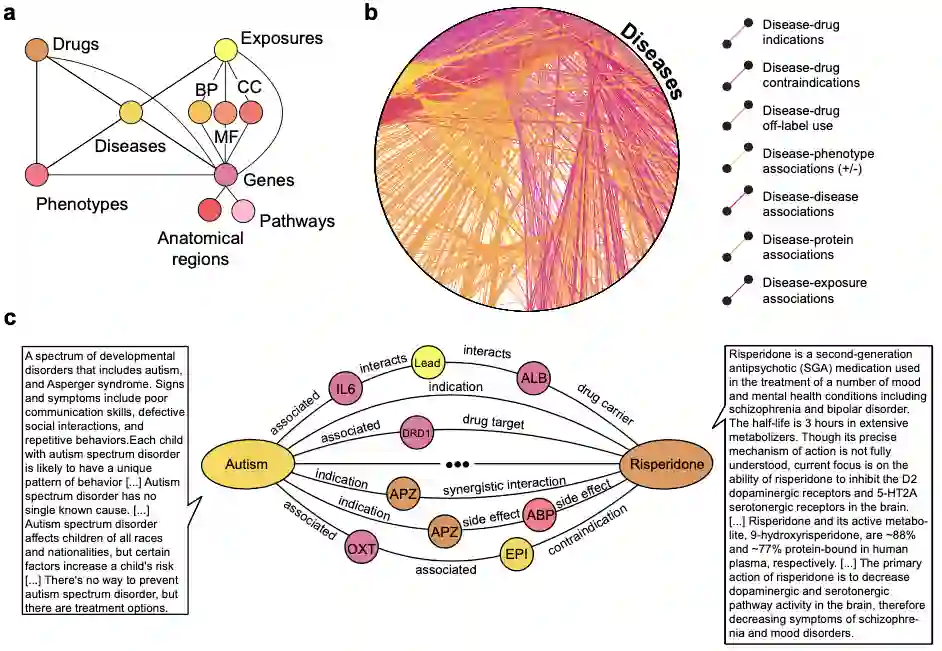
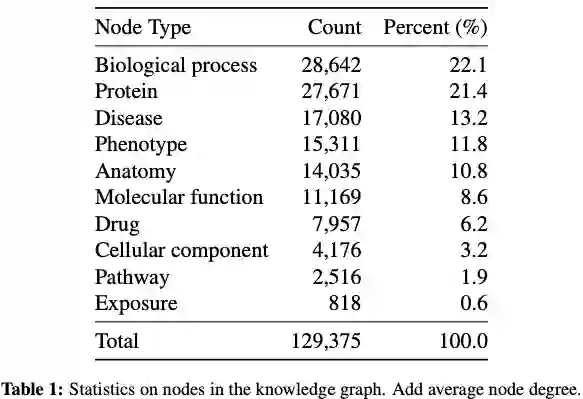
PrimeKG包含了129375个节点和8100498条边。图a显示了图结构的示意图,包含10种类型的节点和30种类型的边。表1提供了节点类型划分的节点数量,
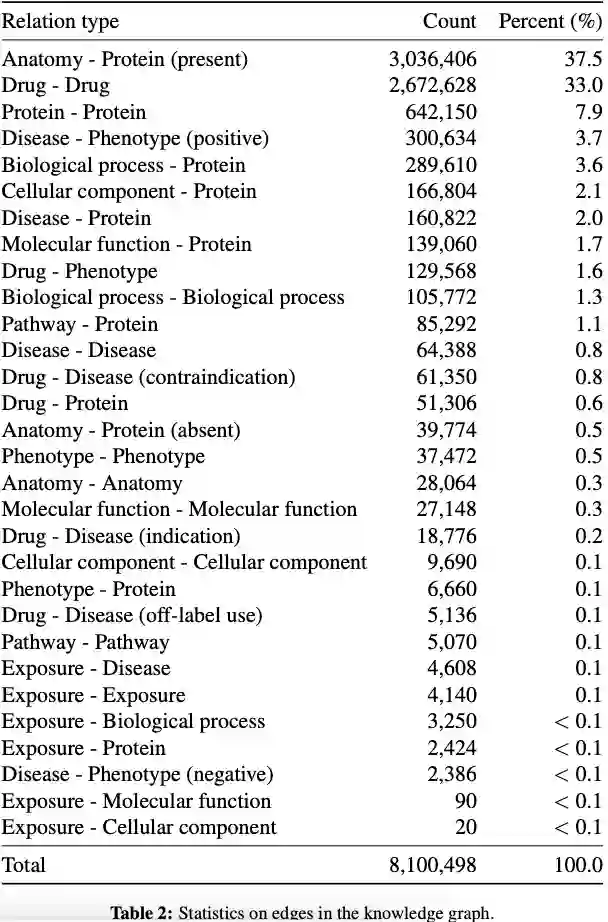
表2按边类型划分的边数的细分。
图b表明疾病节点与知识图中的其他节点类型紧密相连。
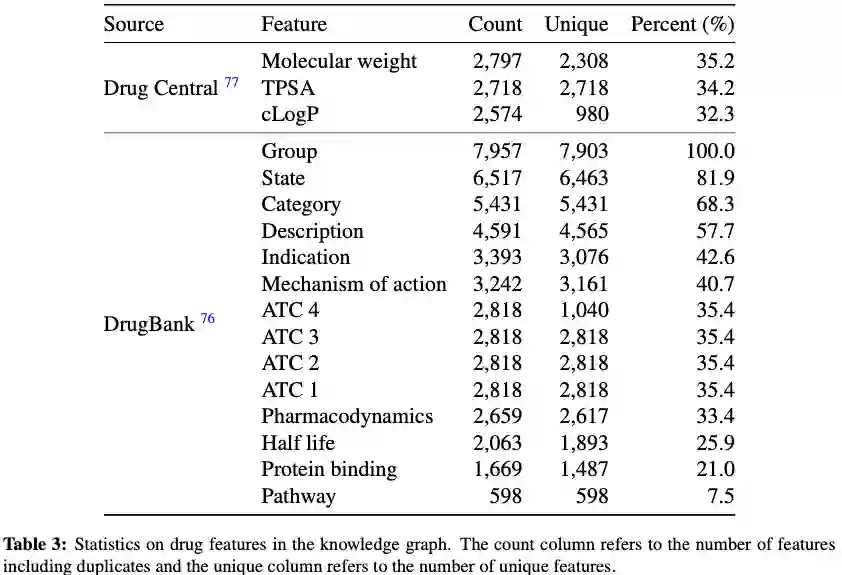
表3显示可用于药物节点的特征数量的统计信息。
表4显示可用于疾病节点的特征数量的统计信息。
疾病特征包括有关疾病流行、症状、原因、危险因素、流行病学、临床描述、管理和治疗、并发症、预防和何时就诊的信息。药物特征包括化合物的分子量、适应症、作用机制、药效学、蛋白质结合事件和信息通路等信息。这种描述整个药物和疾病范围的广泛临床信息是PrimeKG的独有特征,使PrimeKG在其同行知识图谱中脱颖而出。图c提供了跨这些功能可用的支持信息的示例。
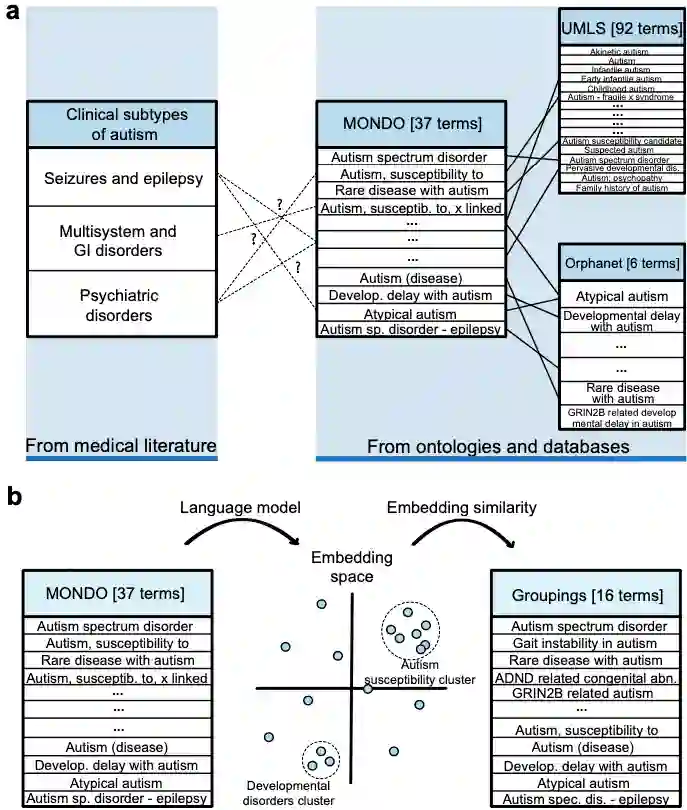
自闭症案例研究评估PrimeKG与自闭症临床表型的相关性
作者分两步评估PrimeKG中与自闭症相关的疾病节点相关性;首先,通过在所有相关的原始数据资源中对自闭症概念执行实体解析,其次,检查这些自闭症概念与自闭症临床亚型之间的关系。
将自闭症节点整合成更具医学相关性的实体
a)左侧显示了三种临床确定的自闭症亚型。右侧显示了三个本体中与自闭症相关的疾病术语:MONDO、UMLS和Orphanet。虽然可以识别跨本体的映射,但尚不清楚任何本体中的术语如何连接到临床亚型。b)说明如何使用语言模型ClinicalBERT将MONDO中术语映射到潜在嵌入空间。因为语言模型可以潜入空间中同义词进行分组,所以作者可以通过计算疾病概念嵌入之间Cosine相似度来对具有相似语义和医学意义的MONDO术语进行聚类。创建这些聚类是为了开发疾病分组,如b的右侧所示。
4 数据记录
详细描述用于构建PrimeKG的20个主要数据资源。
Begee动物基因表达知识库
Bgee73 包含跨多个动物物种的基因表达模式。我们于 2021 年 5 月 31 日从ftp://ftp.bgee.org/current/download/calls/expr_calls/Homo_sapiens_expr_advanced.tsv.gz检索了人类的基因表达数据。处理涉及仅保留黄金质量调用并确保解剖实体是使用 UBERON 本体编码。为了仅提取解剖实体中的高表达基因,我们根据经验过滤数据以保持表达等级小于 25,000 的数据。处理后,我们发现存在或不存在基因表达的 1,786,311 个解剖结构-蛋白质关联。
Comparative Toxicogenomics Database(CTD)
Comparative Toxicogenomics Database(CTD) 专注于环境暴露对人类健康的影响。作者于 2021 年 6 月 9 日从http://ctdbase.org/reports/CTD_exposure_events.csv.gz检索了有关expousures的信息(05/21 版)。处理涉及从 csv 文件中删除标题注释。处理后,数据包含 180,976 个暴露与蛋白质、疾病、其他暴露、生物过程、分子功能和细胞成分的关联。
DisGeNET基因疾病关联知识库
DisGeNET75 是由专家整理的关于基因与人类疾病之间关系的资源。作者于 2021 年 5 月 31 日从https://www.disgenet.org/static/disgeneLa_p1/files/downloads/curated_gene_disease_associations.tsv.gz检索了手动制作的疾病基因关联(7.0 版)数据。原始数据文件“curated gene disease associations.tsv“未经过处理,包含 84,038 个基因与疾病和表型的关联。
疾病本体
疾病本体通过使用临床相关特征将疾病分组在许多有意义的聚类中。例如,疾病按解剖实体分组。作者于 2021 年 6 月 29 日从https://raw.githubusercontent.com/DiseaseOntology/HumanDiseaseOntology/main/src/ontology/HumanDO.obo检索到本体。原始数据“HumanDO.obo”映射到我们知识图中的疾病节点。由于 Mondo 疾病本体没有按解剖学或临床专业进行分组,这将允许 PrimeKG 的用户以具有医学意义的格式探索疾病节点。
DrugBank
DrugBank 是一个包含药物知识的资源。作者于 2021 年 5 月 31 日从https://go.drugbank.com/releases/5-1-8/downloads/all-full-database检索了完整的数据库(版本 5.1.8),提取协同药物相互作用。处理后的数据包含 2,682,157 个关联。作者还从原始数据中提取了药物特征。对于超过 14,000 种药物,构建了 12 种药物特征,包括组、状态、描述、作用机制、ATC 编码、药效学、半衰期、蛋白质结合和通路。
作者于 2021 年 5 月 31 日还从https://go.drugbank.com/releases/5-1-8/downloads/target-all-polypeptide-ids检索到有关药物靶点的信息,从https://go.drugbank.com/检索有关药物酶的信息release/5-1-8/downloads/enzyme-all-polypeptide-ids ,关于来自https://go.drugbank.com/releases/5-1-8/downloads/carrier-all-polypeptide-ids的药物载体,关于来自https://go.drugbank.com/releases/5-1-8/downloads/transporter-all-polypeptide-ids的药物转运蛋白。处理涉及结合所有四种资源并映射来自 UniProt ID 的基因名称使用从 HNCG 基因名称https://www.genenames.org检索到的名词到 NCBI 基因 ID 。处理后的数据包含 26,118 种药物-蛋白质相互作用。
Drug Central
Drug Central 整理了有关药物与疾病相互作用的信息。作者于 2021 年 6 月 1 日从https://drugcentral.org/ActiveDownload检索了 Drug Central SQL 转储。数据库被加载到 Postgres SQL 中并提取了药物-疾病关系。处理后的数据包含 26,698 条indication edges、8,642 条contraindication edges和 1,917 条off-label use edge。作者还从“结构”和“结构类型”表的 Drug Central SQL 转储中提取药物特征。作者提取了超过 4500 种药物的特征,代表每种药物的特征包括拓扑极性表面积 (TPSA)、分子量和 cLogP。例如,阿托伐他汀的特点是:有机结构,分子量558.65,TPSA 111.79,ClogP值4.46。
Entrez Gene
Entrez Gene是由 NCBI 维护的资源,其中包含大量基因特异性信息。作者于 2021 年 5 月 31 日从https://ftp.ncbi.nlm.nih.gov/gene/DATA/gene2go.gz检索了有关基因和基因本体术语之间关系的数据。处理涉及使用 goatools 包提取基因和基因之间的关系基因本体论术语。处理后的数据包含 297,917 个基因与生物过程、分子功能和细胞成分的关联。
Gene Ontology
Gene Ontology网络描述了分子功能、细胞成分和生物过程。作者于 2021 年 5 月 31 日从http://purl.obolibrary.org/obo/go/go-basic.obo检索了本体。处理涉及使用 goatools 包 提取基因本体术语和 go 术语之间的关系的信息。处理后的数据包含 71,305 个生物过程、分子功能和细胞成分之间的层次关联。
Human Phenotype Ontology
Human Phenotype Ontology(版本 hpo-obo@2021-04-13)提供有关疾病中发现的表型异常的信息。作者于 2021 年 5 月 31 日从http://purl.obolibrary.org/obo/hp.obo检索到本体。处理涉及解析本体文件以提取本体中的表型术语、父子关系和对其他本体的交叉引用。处理后的数据包含疾病-表型、蛋白质表型和表型-表型边。作者还于 2021 年 5 月 31 日从http://purl.obolibrary.org/obo/hp/hpoa/phenotype.hpoa检索了经过精心标注的注释。此外,作者还提取了 218,128 条疾病与表型之间的精选正负关联。
Mayo Clinic
Mayo Clinic 是一家专注于综合医疗保健的非营利性学术医疗中心和生物医学研究机构。在其网站https://www.mayoclinic.org/diseases-conditions上,Mayo Clinic 收集了有关 2,227 种疾病和病症的症状、原因、风险因素、并发症和预防的信息。作者于 2021 年 3 月 28 日对这些数据进行了网络抓取,并使用mayo.py和disease.py 脚本提取了这些疾病和状况的描述。原始数据可在“mayo.csv”获得。
Mondo Disease Ontology
由于 Mondo 疾病本体 整理了来自广泛本体的疾病,包括 OMIM、SNOMED CT、ICD 和 MedDRA,它是作者定义疾病的首选本体。作者于 2021 年 5 月 31 日从http://purl.obolibrary.org/obo/mondo.obo检索了本体。处理涉及解析本体文件以提取本体中的疾病术语、亲子关系、疾病子集、交叉引用到其他本体,以及疾病术语的定义。处理后的数据包含 64,388 个疾病边。
Orphanet
Orphanet是一个专注于收集罕见疾病知识的数据库。Orphanet 网站https://www.orpha.net/consor/cgi-bin/Disease_Search._List.php?lng=EN整理了有关 9348 种罕见疾病的定义、流行、管理和治疗、流行病学和临床描述的信息。作者于 2021 年 5 月 10 日使用orpha.py上提供的代码对这些数据进行网络抓取并提取疾病特征。
四种物理蛋白质-蛋白质相互作用的综合资源
蛋白质-蛋白质相互作用由经实验验证的蛋白质之间的相互作用组成。作者考虑的相互作用在本质上是多种多样的,包括信号传导、调节、代谢途径、激酶-底物和蛋白质复合物相互作用,这些相互作用被认为是无权和无向的。
作者使用 Menche等人编制的人类 PPI 网络,作为起始资源。该资源集成了多个蛋白质-蛋白质相互作用数据库,包括用于调节相互作用的 TRANSFAC 、用于酵母与混合二元相互作用的MINT 和 IntAct ,以及用于蛋白质复合物相互作用的CORUM。此外,作者从 BioGRID 和 STRING 数据库中检索蛋白质-蛋白质相互作用信息。作者还考虑了 Luck等人生成的人类参考相互作用组 (HuRI) 。具体来说,作者使用 HI-union,结合了 HuRI 和几个相关的努力来系统地筛选蛋白质-蛋白质相互作用。处理后的数据包含 642,150 条边。
Reactome pathway database
Reactome 是一个开源的、精选的通路数据库。作者于2021年 5 月 31 日从 https://reactome.org/download/current/ReactomePathways.txt 检索到有关通路的信息,从https://reactome.org/download/current/ReactomePathwaysRelation.txt检索通路之间的关系,从https ://reactomePathwaysRelation.txt检索通路-蛋白质关系。/reactome.org/download/current/NCBI2Reactome.txt。处理涉及提取本体信息,例如层次关系和提取通路-蛋白质相互作用。处理后的数据包含 5,070 个通路-通路和 85,292 个蛋白质-通路边。
Side effect knowledgebases
Side effect knowledgebases(SIDER)包含有关药物不良反应的数据。作者于2021年 5 月 31 日从http://sideeffects.embl.de/media/download/meddra_all_se.tsv.gz检索副作用数据(SIDER 4.1 版本),并从http://sideeffects/embl.de/media/download/drug_atc.tsv
检索SIDER 的药物到解剖治疗化学(ATC)分类映射。处理涉及提取 MedDRA 术语在“PT”或首选术语级别编码的所有副作用,然后将药物从 STITCH ID 映射到 ATC ID。处理后的数据 202,736 包含药物-表型关联。
Uberon multi-species anatomy ontology
Uberon是一个包含人体解剖信息的本体。作者于 2021 年 5 月 31 日从http://purl.obolibrary.org/obo/uberon/ext.obo检索到本体。处理涉及提取有关解剖节点及其之间关系的信息。处理数据 28,064 解剖节点之间的层次关系。
UMLS 知识库
统一医学语言系统 (UMLS) 知识源包含有关生物医学和健康相关概念的信息。作者于 2021 年 5 月 31 日从https://download.nlm.nih.gov/umls/kss/2021AA/umls-2021AA-metat hesaurus.zip 以“.RRF”格式检索了完整的 UMLS Metathesauras 。为了将 UMLS CUI 术语映射到 Mondo 疾病本体,作者使用“MRCONSO.RRF”来提取英语中的 UMLS 概念唯一标识符 (CUI) 术语。作者以两种方式将 UMLS CUI 术语映射到 Mondo 术语。首先,作者直接从Mondo本体中提取了两者之间的交叉引用。其次,作者使用 OMIM、NCIT、MESH、MedDRA、ICD 10 和 SNOMED CT 作为中间本体,将 UMLS 间接映射到 Mondo。
此外,作者使用“MRSTY.RRF”和“MRDEF.RRF”文件来提取 UMLS 术语的定义。在“MRSTY.RRF”文件中存在的 127 种语义类型中,作者以与先前工作一致的方式选择了属于 Disorder 语义组的 11 种。这些语义类型是先天性异常、后天异常、损伤或中毒、病理功能、疾病或综合征、精神或行为功能障碍、细胞或分子功能障碍、疾病实验模型、体征和症状、解剖异常和肿瘤过程。然后,作者使用“MRDEF.RRF”文件从英文来源中提取 CUI 术语的定义。
Additional vocabularies
作者于 2021 年 5 月 31 日从https://www.genenames.org/download/custom/检索了NCBI Entrez ID 和 UniProt ID 之间的基因名称和映射。作者从https://go.drugbank.com/检索了 DrugBank 药物词汇表2021 年 5 月 31 日发布/5-1-8/downloads/all-drugbank-vocabulary。这些用于将知识图中的节点映射到一致的本体。
代码与数据地址
项目网站:https://zitniklab.hms.harvard.edu/projects/PrimeKG
代码地址:https://github.com/mims-harvard/PrimeKG
数据地址:https://doi.org/10.7910/DVN/IXA7BM