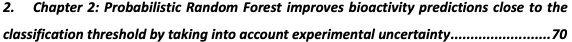机器学习用于化合物的作用机制、安全性(包括PROTACs)预测|剑桥大学249页博士论文
该论文回答两个问题:使用哪些数据和方法来理解化合物的MOA;如何探索新数据的安全性如蛋白质水解靶向嵌合体(PROTACs)等模式。
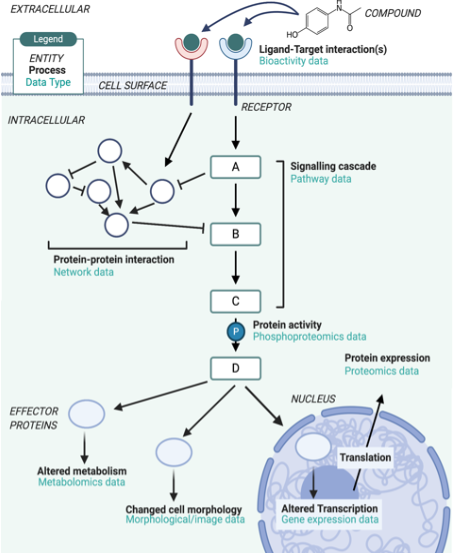
Figure:MoA研究中使用不同类型的数据/信息可以定义不同类型的MoA(Trapotsi et al[1])
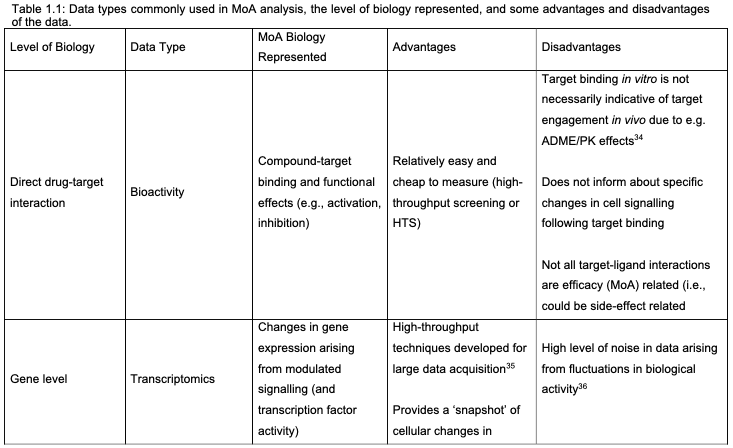
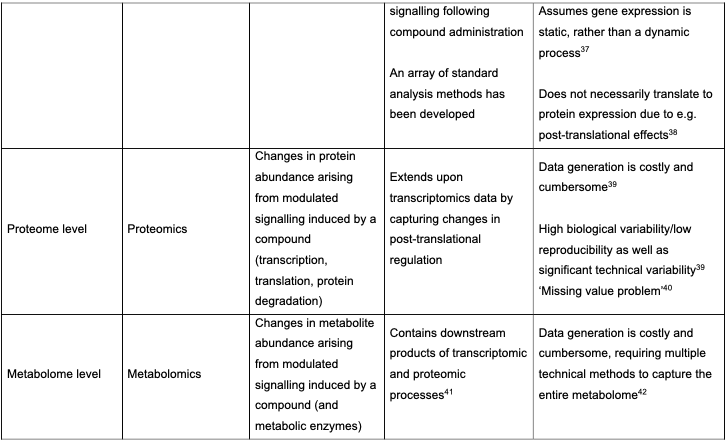
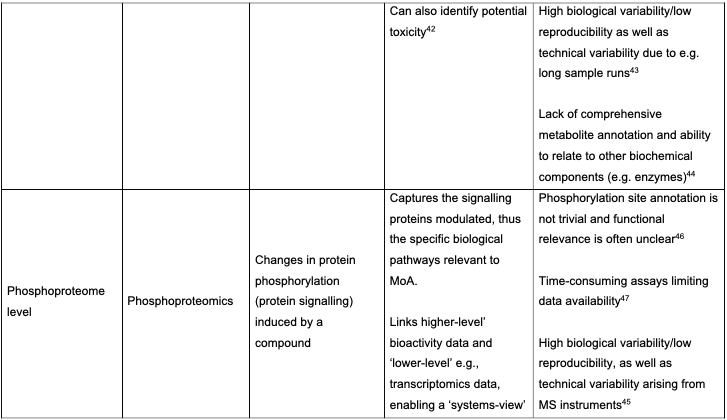
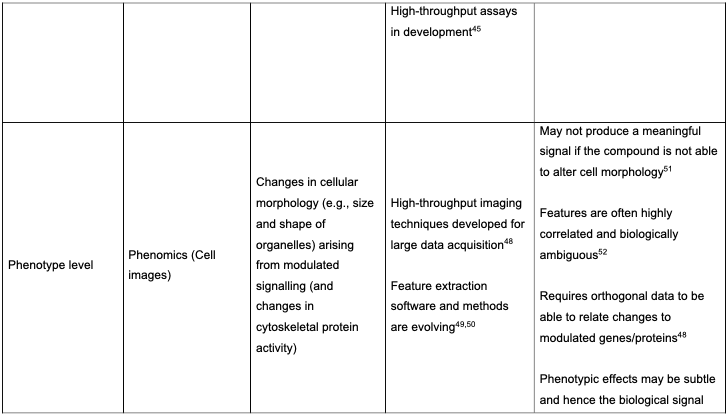
分析的生物学层次包括Direct drug-target、Gene level、Proteome level、Metabolome level、Phosphoproteome level、Phenotype level、Biological pathway level,如下表。论文对这些点都进行了具体讨论,包括机器学习在这一块的应用。
特别的,作者对新数据与新作用机制也进行了分析,包括PROTACs的数据、安全性与MoA。
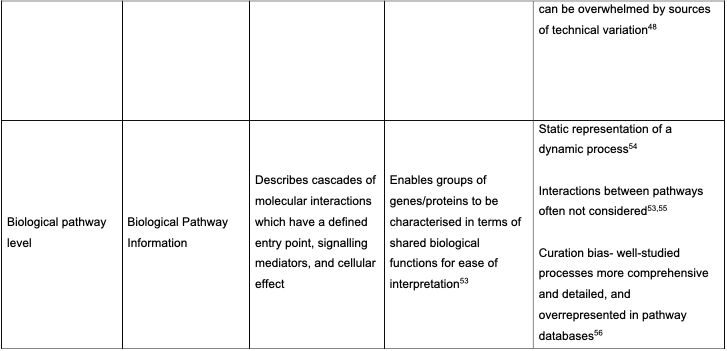
第二章:“通过考虑实验不确定性,使用概率随机森林(Probabilistic Random Forest)改善生物活性的二分类预测”。
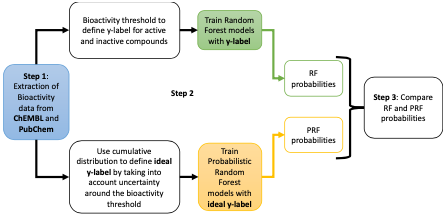
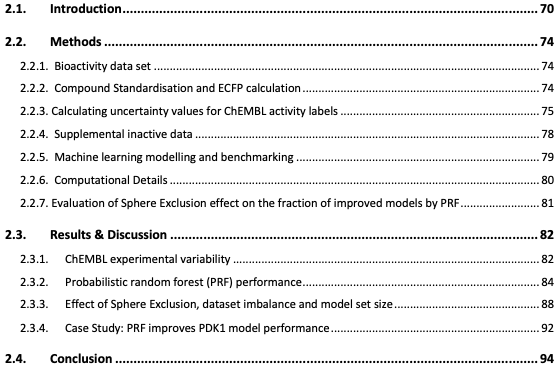
第三章:“通过比较结构化学和细胞形态信息,使用多任务学习对生物活性预测“。利用细胞涂饰分析中的细胞形态信息(以CellProfiler特征的形式)和化学结构信息(以Extend connectivity Fingerprints的形式)进行比较。比较表明,有一些靶点可以通过细胞形态学信息可以更好地预测,如b-catenin,还有一些靶点可以通过化学结构信息更好地预测,如属于G-蛋白偶联受体1家族的蛋白质。
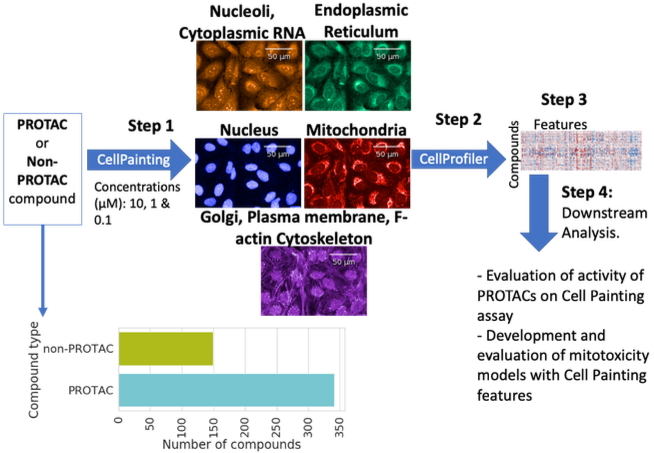
第四章:“在PROTACs数据集上使用细胞绘制分析预测线粒体毒性”,探讨了使用细胞绘制分析成功分析新数据模式(PROTACs)的方法,并评估了该分析方法是否可用于理解这些新化合物的安全性。在PROTACs数据集中,细胞形态特征(以CellProfiler特征的形式)成功预测了线粒体毒性。这项工作产生了第一个使用基于细胞绘制的特征预测PROTACs线粒体毒性的ML模型,并扩展了我们对PROTACs安全性预测的知识。
图:分析与总结。PROTACs和非PROTACs化合物通过细胞涂饰分析进行分析,然后进行数据归一化和下游分析。
往期PROTACs相关文章
BioRxiv|机器学习结合高内涵预测PROTAC有丝分裂毒性
JCIM | 深度图生成模型用于设计PROTAC、FBDD、骨架跃迁的linker
Nucleic Acids | PROTAC-DB:PROTAC在线数据库
回台回复: MoA 获取博士论文