Nucleic Acids | PROTAC-DB:PROTAC在线数据库
浙江大学人工智能药学侯廷军团队在Nucleic Acids Research上发表了一篇题为“PROTAC-DB:an online database of PROTACs“的文章。
该PROTAC数据库:PROTAC-DB,集成了结构信息和实验数据。
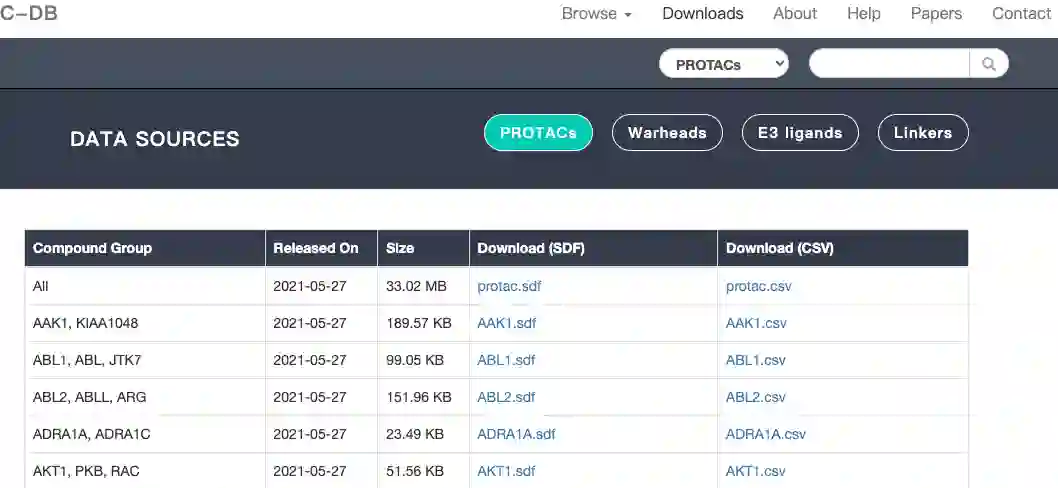
目前PROTAC-DB由1662个PROTAC、202个warhead(靶向目标蛋白的小分子)、65个E3配体(能够招募E3连接酶的小分子)和806个linker组成,以及它们的化学结构、生物活性和物理化学性质。
除了warhead和E3配体的生物活性外,PROTAC-DB还提供PROTACs降解能力、结合亲和力和细胞活性。
提供了数据下载链接:
PROTAC-DB可以使用两种搜索方法进行查询:基于文本(target name,compund name or ID)和基于结构的。此外,为方便用户,还提供了基于化合物理化性质的检索过滤工具。
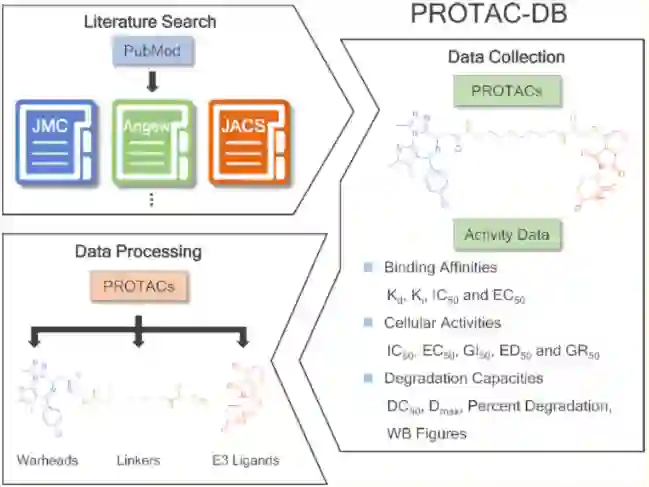
构建PROTAC-DB三个步骤:(1)文献检索,(2)数据收集,(3)将PROTACs分类为warheads, E3 ligands和linkers做预处理。
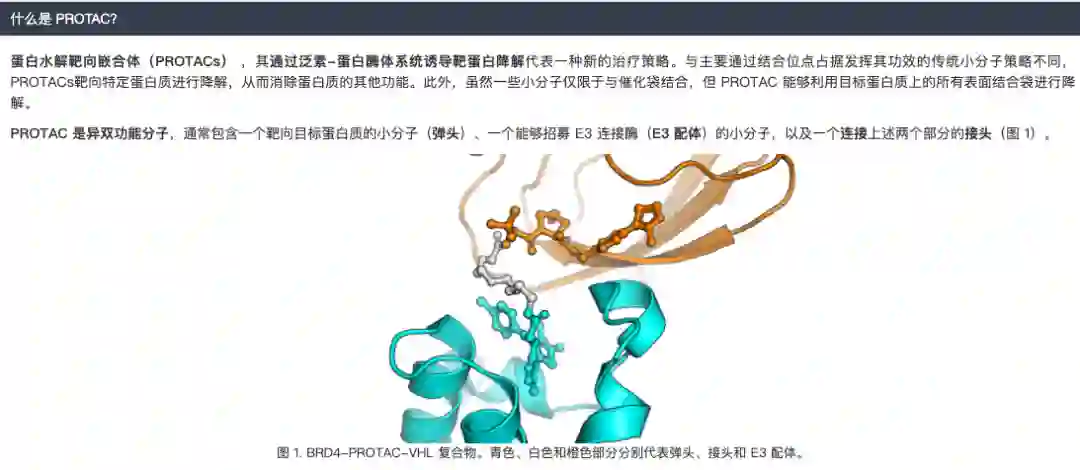
PROTAC是异双功能分子,它包含一个靶向靶蛋白的小分子(warhead)、一个能够招募E3连接酶(E3配体)的小分子以及一个连接上述两个部分的linker。
尽管在过去十年汇总取得了巨大进步,但设计具有理想物理化学、吸收、分布、代谢和排泄特性的PROTAC仍然是一个巨大的挑战。
与传统小分子药物不同,PROTAC由于其高分子量可能不符合Lipinski的“五原则”,这将限制它们的细胞渗透性和其他类似药物的特性。虽然PROTAC的优化应该更多的关注整个分子而不是单个分子,但在初步PROTAC设计中单独考虑它们是有用的。
在这方面,Linker设计被认为是PROTAC设计的最关键领域。此外,积累的证据表明,linker和PROTAC的熵、选择性、活性、水溶性、渗透性等有关。然而,对linker的设计的探索是无止境的。为了促进PROTACs的合理设计,收集和注释PROTACs的实验数据和结构信息是非常必要的。
虽然有一些综合数据库也收集了一些PROTACs信息,例如GtoPdb,PubChem和ChEMBL,这些数据库中PROTAC的数量和实验信息仍然相当有限。
PROTAC-DB 是一个公共的、可通过网络访问的数据库。这些化合物的化学结构、生物活性、理化性质是人工从文献中提取或通过一些程序计算得出的。在这里,PROTACs 的生物活性包括降解能力、结合亲和力和细胞活性。
详细信息如下:
降解能力:一般而言,DC50(导致 50% 靶向蛋白质降解的浓度)和Dmax(蛋白质降解的最大水平)用于量化 PROTACs 的靶向蛋白质降解能力。然而,由于大量 PROTACs 缺乏上述数据,如果至少用两个浓度评估并且每个浓度用至少两个独立实验测量,则将降解百分比整合到数据库中。
此外,我们还收集了蛋白质印迹 (WB) 数据以显示 PROTAC 的降解能力。但是WB数字只显示在PROTAC的详细信息页面上,而不是在其列表页面上。
结合亲和力:PROTACs 与靶蛋白、PROTACs 和 E3 连接酶之间的结合亲和力 ,三元复合物的形成被收集到 PROTAC-DB 中。三元复合物形成的结合亲和力用于评估 PROTAC 诱导的 E3 连接酶和靶向蛋白质形成复合物的能力。
它可以通过 E3 连接酶(靶向蛋白)和 PROTAC 和靶向蛋白的复合物(E3 连接酶)之间的一些测定来确定。有四种类型的值,包括 Kd、Ki、IC50 和 EC50。只有 Kd 和 IC50 将显示在列表页面上,其他显示在详细信息页面上。
此外,对于生物物理结合数据,ΔG、ΔH、-TΔS、t1/2、kon和koff也被收集到数据库中,仅显示在详细信息页面上。
细胞活性: IC50、EC50、GI50、ED50 和 GR50 被收集到 PROTAC-DB 中。同样,ED50 和 GR50 仅显示在详细信息页面上,而不显示在列表页面上。
此外,还收集了弹头和E3配体的生物活性。我们将不断添加新数据并提高界面的可用性。
数据库:
http://cadd.zju.edu.cn/protacdb/