蛋白质大模型
·

尽管现有的综述文章侧重于特定的方面或应用,本研究提供了首个关于蛋白质大语言模型的全面综述,涵盖了其架构、训练数据集、评估指标和多样化的应用。通过对超过100篇文章的系统分析,我们提出了一个结构化的分类法,系统总结了最前沿的蛋白质大语言模型,分析了它们如何利用大规模蛋白质序列数据提高准确性,并探讨了其在推动蛋白质工程和生物医学研究中的潜力。此外,我们还讨论了关键挑战和未来方向,将蛋白质大语言模型定位为蛋白质科学中科学发现的关键工具。相关资源可在 https://github.com/Yijia-Xiao/Protein-LLM-Survey 获取。
1 引言
“蛋白质是生命的机械,理解它们的语言将揭开生物学的秘密。” — 大卫·贝克(2024年诺贝尔奖获得者)蛋白质是重要的生物分子,推动着诸如催化生化反应、维持细胞结构和实现细胞间通信等功能。理解蛋白质的序列-结构-功能关系是生物学研究的核心。然而,传统的实验方法,如X射线晶体学、核磁共振(NMR)光谱学和冷冻电镜(cryo-EM),不仅耗时且劳动密集,成为大规模应用的瓶颈。近年来,语言建模的进展彻底改变了计算生物学,提供了强大的蛋白质分析工具。蛋白质大语言模型(Protein LLMs)与传统的大语言模型(LLMs)在多个基础方面有相似之处:
- 训练目标和学习范式:蛋白质LLMs和LLMs都通过自监督方式,在大规模数据集上进行训练,使用如掩码语言建模(Devlin等,2019)、自回归建模(Luo等,2022)或句子重排(Lewis等,2020;Yuan等,2022)等目标,学习预测序列中缺失或下一个元素。LLMs预测文本数据中缺失的单词或短语(Reimers和Gurevych,2019;Liu等,2019;Touvron等,2023),而蛋白质LLMs则预测蛋白质序列中的氨基酸或子序列。
- 预训练数据:蛋白质LLMs采用数据驱动的范式,直接从大规模的蛋白质数据集中学习(Liu等,2024b;Jones等,2024)。用于训练蛋白质LLMs的数据集由庞大的蛋白质序列集合构成,类似于LLMs用于训练的文本语料库。这消除了显式特征工程的需要,使得蛋白质LLMs能够学习复杂的模式,如结构模体、进化关系和功能信息,类似于LLMs在语言中捕获语义和句法结构。 这一范式的转变催生了高效的模型,能够预测蛋白质折叠、注释生物学功能,甚至设计具有特定特性的全新蛋白质。除了预测能力,蛋白质LLMs还提供了交互式界面,允许用户上传蛋白质序列或结构文件(如PDB格式),提出问题并以对话形式与模型互动(Liu等,2024c;Xiao等,2024b,c),深入理解蛋白质的结构、功能和设计。我们首次进行专门的蛋白质LLMs综述,分析其独特的架构、训练方法和在蛋白质研究中的实际应用。虽然之前的研究探讨了各种计算方法在蛋白质研究中的应用(Chen等,2024c;Wu等,2022),或讨论了语言模型在生物医学(Wang等,2023a)和化学(Liao等,2024)等科学领域的作用,但本综述特别聚焦于蛋白质LLMs——这一在计算生物学和自然语言处理交叉领域快速发展的研究领域。主要贡献如下:
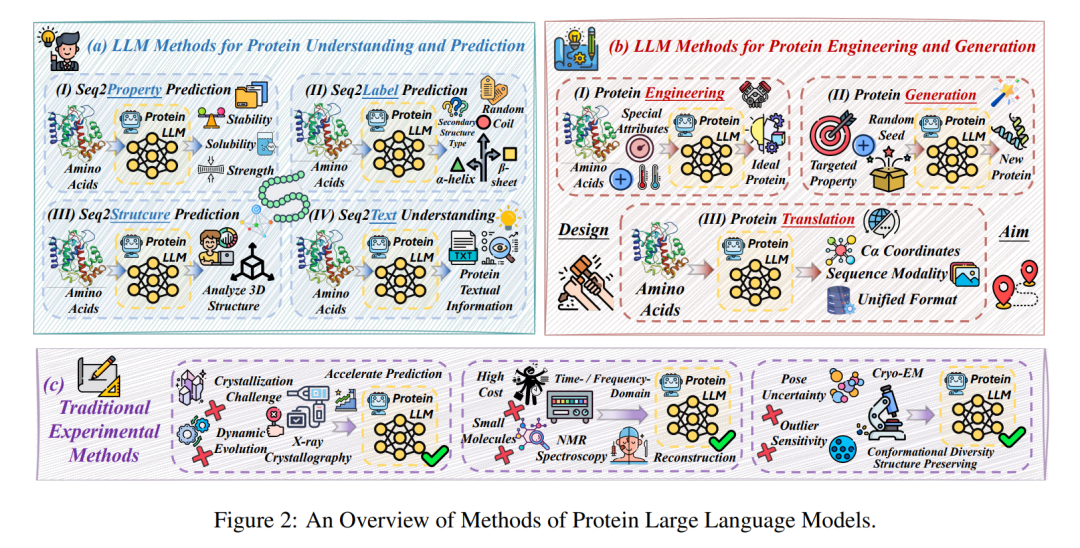
- 架构概述:提出了最前沿的蛋白质LLMs的结构化分类法(图3),详细介绍了它们在蛋白质理解(§2)和生成(§3)中的独特架构,突出这些模型在效率和准确性上如何超越传统实验方法(附录§A)。
- 数据洞察:对用于预训练、微调和基准测试的蛋白质LLMs数据集进行全面总结,提供了数据整理策略及其对模型性能影响的关键洞察(§4)。
- 评估协议:深入讨论了评估蛋白质LLMs性能和影响的方法,包括全新的基准测试策略(§5和附录§B)。
- 应用:详细探索蛋白质预测、注释和设计中的实际应用,特别强调了最近的创新进展,并展示了蛋白质LLMs在推动生物医学研究方面的变革潜力。
成为VIP会员查看完整内容
相关内容
Arxiv
40+阅读 · 2023年4月19日
Arxiv
211+阅读 · 2023年4月7日
Arxiv
79+阅读 · 2023年4月4日
Arxiv
144+阅读 · 2023年3月29日