摘要
在对真实世界的数据进行建模之前,所做的假设极大地影响了机器学习的性能任务。因此,为了成功开发机器学习模型,找到一个好的数据表示方法是最重要的。当数据不存在相当大的先验假设时,数值会直接被表示在一个 "扁平化"的一维矢量空间中。然而,可以更进一步,感知更复杂的关系模式:例如,使用图维空间来说明表示数据的更结构化的方式及其关系归纳偏差。
本论文主要关注人类生物学两个尺度上的计算数据维度:使用基因表达数据的分子生物学微观尺度和使用神经影像数据的神经科学宏观尺度。我们将探索不同的建模方法,以了解如何在这两个尺度的应用领域中对高维度的大脑数据进行建模和表示。具体来说,对于图维数据,将开发两种方法。首先,通过在49个人体组织中应用多层共表达网络,将提取可普遍适用于外部数据集的特定和共享的基因谱。然后,将引入一个新的深度学习模型,以利用整个静息状态fMRI数据(即空间和时间动态),与之前文献中简化和浓缩这类数据的方法不同,同时说明其在外部多模态数据集中的稳健性和可解释能力。对于一维数据,将开发一个可解释的模型,以利用多模态大脑数据理解认知因素。
总的来说,本论文采用的研究探讨了可解释的数据驱动的表征和建模方法,涉及机器学习、分子生物学和神经科学等多学科的科学领域。它还有助于突出这些领域在为大脑及其在人体中的内部和相互动态建模时的贡献。
引言
1.1 数据表示的复杂性
科学已经非常成功地推进了我们对世界的理解,通过在许多领域建立模型,同时关注不同的数量级。例如,我们可以关注天文尺度(即10^8和10^24米左右),一直到亚原子(即10^-18米以下)。这个迷人的尺度范围说明了在试图对世界进行建模时涉及的内在复杂性。找到一个好的数据表示,对于使用计算方法成功建立科学的某些方面的模型是最重要的。在机器学习领域,这通常被称为表示学习(或表征学习)[31],它的正确使用可以大大影响后续的学习任务。
本论文认为,在对现实世界的案例进行建模时,有两种主要的方式来看待特定的尺度,这影响了信息的获取和表达。最简单的方式是当人们直接感知到简单的特征或数值时;例如,在人的尺度中,这可能是人的高度,甚至是简单的颜色感知(例如,这张桌子是棕色的,这个杯子是黄色的)。从技术上讲,这些数值以 "扁平化"表示,或者换句话说,以一维矢量空间表示;我们不对数据做任何预先假设,只是直接感知观察到的东西。然而,我们可以更进一步,在这些更简单、直接的模式之外感知更复杂的模式。例如,除了棕色的桌子和黄色的杯子,人们可以看到一些关系:杯子在桌子上,人在看杯子。一个更典型的关系例子存在于社会网络中,除了我们对每个人的特征之外,我们还有连接他们的友谊关系。我把这种表示方法称为图-维空间,以说明代表数据的更加结构化的方式。
图维空间是考虑关系性归纳偏见的结果,在这里,我们不仅试图感知扁平的、简单的特征,而且还感知它们的任意关系。关系性归纳偏见在分子生物学和神经科学领域普遍存在。在观察大脑结构扫描时(见第2.3节),人们可以感知到扁平化的特征,如右半球前额皮质的厚度,或左半球的灰色体积。这两个区域可能有不同的关系:它们的神经元在一段时期内是以类似的模式发射,还是以非同步的方式?同样,如果取一个血样并测量其基因表达谱(见第2.2节),我们可以看到特定的基因如何更多或更少地表达(即扁平化表示);如果这种表达谱与身体的其他部分共享,那么血液就与这些身体部分有关。
乔治-博克斯在他的一些工作中著名地提到:"所有的模型都是错的,但有些是有用的"[42]。本论文正是在这个庞大而复杂的现实世界下进行的:在开发 "错误"的模型时,仍然可以在某些方面 "有用"。我将把注意力缩小到人脑生物学的两个迷人的尺度上:首先是使用基因表达数据的分子生物学的微观尺度,然后是使用神经影像数据的神经科学的宏观尺度。
1.2 研究问题
上一节说明了如何通过使用扁平化的特征(即一维表征)或以图形形式的相关实体的特征(即图-维表征)来影响表征学习。本论文旨在探索脑科学中使用1-D和Graph-D表示的不同建模方法,重点是基因表达和神经影像领域。鉴于这一背景,在本论文中我将寻求探索三个主要的研究问题(在括号中加入了一个名字,以便进行文字识别)。
-
问题1.(表征)我们如何根据分子生物学和神经科学应用领域的具体需要,对非常复杂和高维的大脑数据进行建模和表征?
-
问题2.(解释)是否有可能向应用研究人员提供模型,对如何做出决策提供解释,即使是学习复杂的非线性模式?
-
问题3.(图)我们如何整合基于图的数据,以便更好地理解大脑的神经和遗传机制?
机器学习在众多领域利用不同的数据结构所取得的历史和近期的成功[86, 153],使得这一计算子领域成为回答这些研究问题的明显候选者。不可能开发出一个放之四海而皆准的解决方案;因此,我将研究深度学习和更传统的机器学习方法对研究问题1(表征)中每个应用领域的适合性。就分子生物学而言,将探索基因表达数据,而就神经科学而言,我将专注于神经影像数据。虽然在神经影像领域,分析通常只集中在大脑上,但在基因表达领域,我们可以获得与大脑相同格式的人体其他部位的数据。因此,我将利用基因表达数据的这一特殊特点,使大脑模型的开发能够利用其他身体组织的信息。
使用机器学习方法来探索数据的错综复杂的非线性模式可能会导致该领域众所周知的问题。在过去,科学家们开发的尖端模型打败了基准,却不太了解模型实际在学习什么[53]。虽然这些黑箱模型可能非常有帮助[140],但如果不了解模型在学习什么,就会导致对抗性攻击[8,99]或在分布之外的数据中无法归纳[261]。这些问题一旦发生在安全关键环境中,就会对人类产生严重的后果,也证明了研究问题2(解释)的相当重要性(见第2.1.7节)。
最后,研究问题3(图形)集中在使用Graph-D空间的数据的具体情况,以及与使用扁平化数据表示法相比,这个空间带来了哪些额外的机械学优势(见第2.1.8节)。
总而言之,鉴于分子生物学和神经科学等现实领域的内在复杂性,我的目的是通过不同的、但互补的视角来发展可能的答案。具体来说,我将探索数据的表示方法(即在一维空间和图维空间)和机器学习方法(即监督和非监督)。
1.3 论文结构
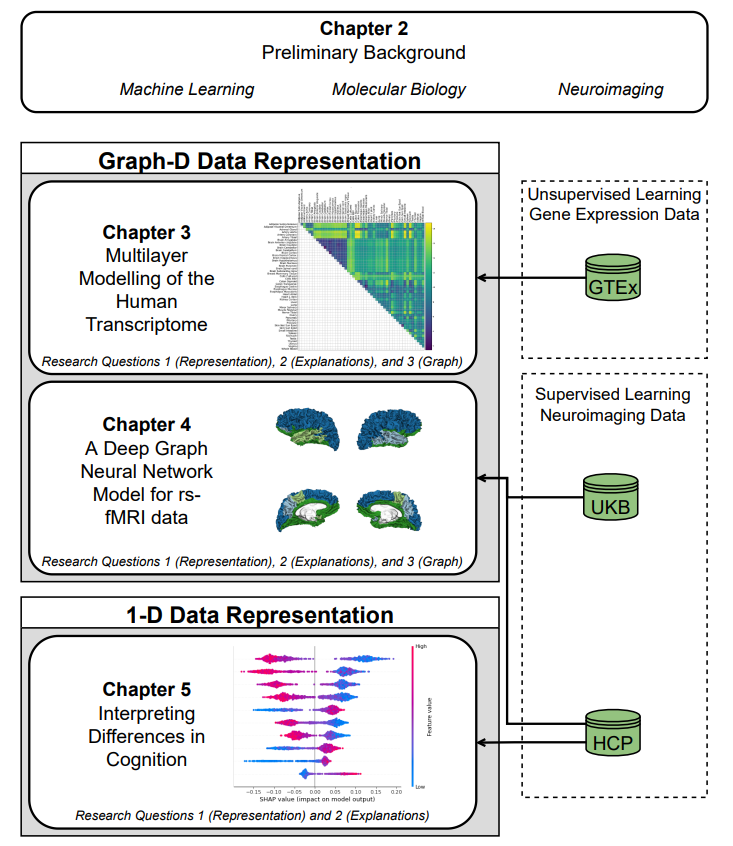
图1.1: 主要章节的论文结构。不同的观点将在不同的数据表示(即1-D和Graph-D空间)、机器学习方法(即监督和非监督)、数据领域(即分子生物学和神经科学)以及由基因表达和神经影像数据组成的各自的主要数据集中进行探索和整合。每一章所解决的研究问题都在第1.2节中显示和解释。每个数据集在其各自的章节中都有详细介绍。第二章提供了在主要章节中探讨的机器学习、分子生物学和神经影像学的基本概念。
论文结构如图1.1所示,以解决上一节中提出的三个研究问题。本论文分为两个主要部分。第一部分探讨了图-维数据表示法,由两章组成。第二部分探讨一维数据表示法,由一章组成。前两个研究问题将在这三个主要章节中进行探讨,而第三个研究问题将只在前两个主要章节中明确探讨。
为了帮助回答研究问题1(表征),我将在这三个主要章节中分别处理不同的挑战。在第三章中,我将着重于使用多层方法对13种脑组织和36种其他人体组织的共同表达网络进行建模,旨在提取特定的和共享的基因图谱,这些图谱可以被推广到外部数据集。第四章将介绍一个新的深度学习模型,它可以利用整个静止状态的fMRI数据(见第2.3节),而不是以前文献中简化和浓缩这类数据的方法。最后,在第五章中,我将重点讨论如何利用特征工程从大脑数据中提取更多可解释性信息,以更好地理解认知因素。
我将设法在所有三章中提供关于每个模型所学的具体见解,以解决关于可解释模型的研究问题2(解释)。事实上,在第三章中,提供了所有的代码和跨人体组织的每个社区的信息,所以任何人都可以分析每个基因特征是如何在人体组织中共享的。第四章将利用跨样本形成的聚类来理解模型中出现的哪些模式,在第五章中,我将为手头的每项任务提供一份信息量最大的大脑特征的排名表。
最后,研究问题3(图)只在第3章和第4章进行探讨。在这些章节中,数据被表示在Graph-D空间中,也就是被建模为图。事实上,在前一章中,我采用了一种多层方法来表示共表达网络的关系(即图),而在后一章中,我提出了一种深度学习架构,能够专门利用数据的图结构。
现在是研究这些课题的一个特别激动人心的时刻[65, 74, 81, 287],因为有高质量的、经过精心整理的数据集被发布给研究人员使用。在这篇论文的开发过程中,我有机会接触到第4章和第5章中探讨的近千张神经影像扫描,以及第4章中超过3.5万张神经影像扫描(见2.3.3节)。GTEx数据集的最后一个版本是在接近本论文结束时公开发布的[4];事实上,我能够在第三章探索这个最后的版本。本论文中使用的所有数据集的进一步细节,如图1.1所示,将在各章中提供。
除了本论文的三个主要章节之外,在第二章中我将概述本论文所探讨的主题。具体来说,我将总结关于机器学习训练程序的基本概念,以及分子生物学和神经影像学的背景概念。在第六章中,我将强调本论文的主要局限性和贡献,并提出我对源自其局限性的有趣的未来研究方向的想法。