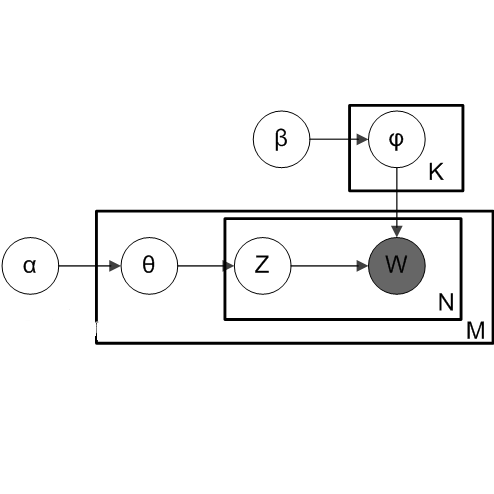
Mixed-membership (MM) models such as Latent Dirichlet Allocation (LDA) have been applied to microbiome compositional data to identify latent subcommunities of microbial species. These subcommunities are informative for understanding the biological interplay of microbes and for predicting health outcomes. However, microbiome compositions typically display substantial cross-sample heterogeneities in subcommunity compositions -- that is, the variability in the proportions of microbes in shared subcommunities across samples -- which is not accounted for in prior analyses. To address this limitation, we incorporate the logistic-tree normal (LTN) model into LDA to form a new MM model. This model allows cross-sample variation in the composition of each subcommunity around some "centroid" composition that defines the subcommunity. Incorporation of auxiliary P\'olya-Gamma variables enables a computationally efficient collapsed blocked Gibbs sampler to carry out Bayesian inference under this model. We compare the LDA and the new model and show that in the presence of large cross-sample heterogeneity, LDA can produce inference which is sensitive to the specification of the number of subcommunities. As such, the popular strategy of overspecifying the number of subcommunities and hoping that some meaningful subcommunities will emerge alongside artificial ones can lead to misleading conclusions. In contrast, by accounting for such heterogeneity, our new model restores the robustness of the inference in the specification of the number of subcommunities and allows meaningful subcommunities to be identified.
翻译:混合成员(MM) 模型,如Lentant Dirichlet 分配(LDA) 模型,已经应用到微生物构成数据中,以确定微生物物种的潜在亚群落。这些亚群群是了解微生物的生物相互作用和预测健康结果的丰富信息。然而,微生物组成通常在亚群落构成中表现出大量的跨相模异性 -- -- 也就是说,在样本之间共享子群落中微生物比例的变化性 -- -- 在先前的分析中没有考虑到这一点。为了应对这一限制,我们将物流-树正常(LTN)模型纳入LDA,以形成一个新的MM模型。这个模型可以使每个亚群落的构成围绕某些“centroid”组成进行交叉抽样变异性,从而界定亚群落。结合辅助的P\'olya-Gamma变量,可以计算高效地阻断Gibbbs取样器,从而根据这个模型将Bayesales的推断结果传播出去。我们比较LADA和新模型,并表明,在大型跨树群群群群群群域(LTAA)的分类和亚群落的精确度定义中,可以使这些亚群落的亚群落的亚群落的亚群落的亚群落的亚群落能够将我们的次群落变成变成变变变变变。