魔改nnU-Net夺冠!2021 BraTS 脑肿瘤分割竞赛第一名解决方案
点击下方卡片,关注“CVer”公众号
AI/CV重磅干货,第一时间送达

代码(刚刚开源):
https://github.com/rixez/Brats21_KAIST_MRI_Lab
本文描述了基于nn-UNet试验了几种改进,包括使用更大的网络、用GN替换BN以及在解码器中使用Axial Attention。与Baseline相比,量化指标略有改进。在unseen test data的最终排名中,nn-UNet赢得了第一名的好成绩。
1改进策略
1.1 数据方面
BraTS2021包括了来自2000名患者的多参数MRI扫描结果,其中1251人的图像提供了分割标签给参与者来开发算法,其中219人在验证阶段被用于公共排行榜,其余530个案例用于私人排行榜和参与者的最终排名。
MRI扫描有4种对比:
-
原生T1加权图像 -
对比后T1加权(T1GD) -
T2加权 -
T2流体衰减反转恢复(T2-Flair)
注释由1-4个评分员手工完成,最终得到有经验的神经放射学家的批准。这些标签包括gd增强肿瘤(ET)、瘤周水肿/侵袭组织(ED)和坏死肿瘤核心(NCR)区域。所有MRI扫描均通过同解剖模板配准、各向同性1mm3分辨率插值和颅骨剥离进行预处理。所有MRI扫描及相关标记的图像大小为240×240×155。
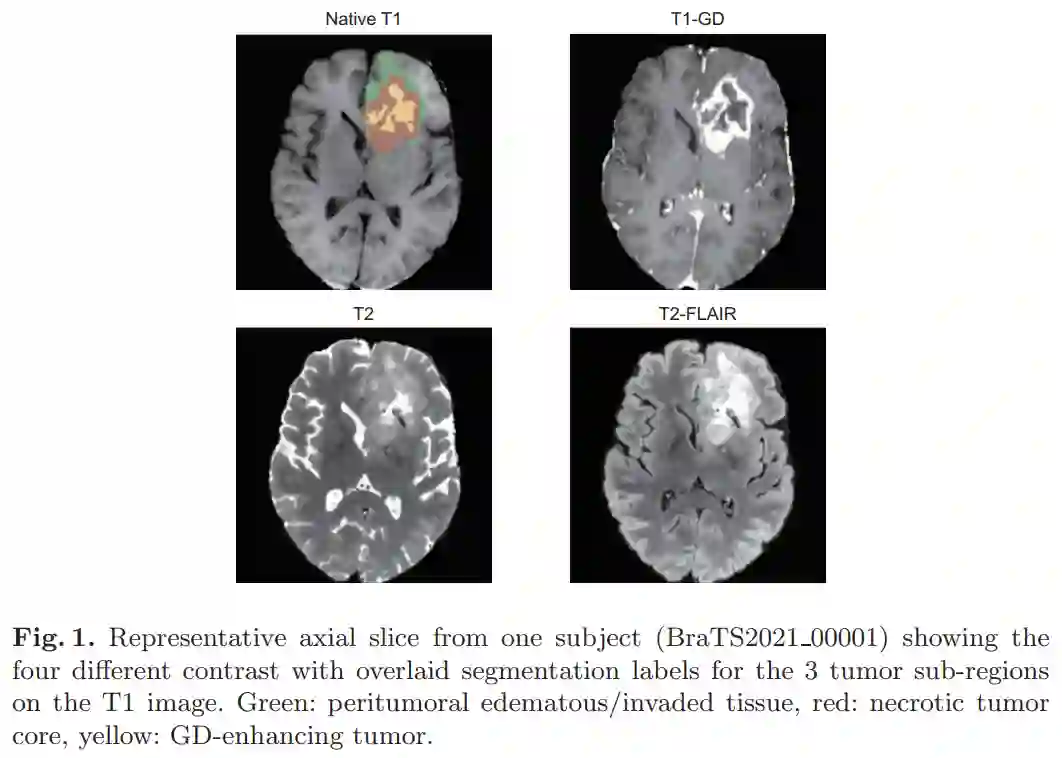
图1显示了这4种对比与分割的代表性切片。在输入网络之前,对提供的数据进行了进一步的处理。为了减少计算量,将volumes裁剪为non-zero voxels。由于MR图像的强度是定性的,因此根据其均值和标准差对voxels进行归一化。
1.2 模型方面
1、Baseline nnU-Net
nnU-Net核心是一个在128×128×128大小的Patch上运行的3D U-Net。该网络具有编码-解码器结构,并带有Skip Connection,将两条路径连接起来。
该编码器由5个相同分辨率的卷积层组成,具有卷积下采样功能。该解码器遵循相同的结构,使用转置卷积上采样和卷积操作在同一级别上的编码器分支的串联Skip特征。每次卷积操作后,采用斜率为0.01的Leaky ReLU(lReLU)和批归一化处理。mpMRI volumes被连接并作为4通道输入。
nnU-Net应用Region-Based训练,而不是预测3个相互排斥的肿瘤子区域,而不是预测3个互斥肿瘤分区,与提供的分割标签一样,该网络预测的是增强肿瘤的3个重叠区域如加强肿瘤(ET,original region),肿瘤核心或TC(ET+necrotic tumor),和整个肿瘤或WT(ET+NT+ED)。
网络的最后一层的softmax被sigmoid所取代,将每个voxels作为一个多类分类问题。
由于公共和私人排行榜的计算指标是基于这些区域的,这种基于区域的训练可以提高表现。额外的sigmoid输出添加到每个分辨率除了2个最低的水平,应用深度监督和改善梯度传播到早期层。卷积滤波器的数量被初始化为32个,并且分辨率每降低一倍,最大可达320个。
2、更大的网络和GN
第一个修改是,通过将编码器中的kernel数量加倍,同时在解码器中保持相同的kernel,非对称地增加了网络的大小。由于训练数据的数量是前一年的4倍,增加网络的容量将有助于它能够建模更大的数据种类。kernel的最大数量也增加到512个,改进后的网络结构如图2所示:

第二个修改是用GN代替所有BN。即使使用混合精度训练,3D卷积网络也需要大量的GPU内存,这限制了在训练中可以使用的Batch-Size。
3、Axial attention解码器
最后添加的是在解码器中使用Axial attention。Self-Attention或Transformer是一个突破性的想法,允许学习一个输入序列的自适应注意力仅仅基于它自己。Self-Attention最初是在自然语言处理中,现在已经慢慢被计算机视觉研究所采用。当试图将Self-Attention应用于视觉问题时,主要的障碍之一是注意力机制的计算复杂度与输入的大小成二次方,这使得它不可能适合或训练网络在一个标准的工作站设置。当处理带有额外维度的3D数据时,这是一个更大的问题。
Axial attention最近被提出作为将注意力应用于多维数据时的一种有效解决方案。通过将Self-Attention独立地应用于输入的每一个轴上,计算只与图像大小成线性比例,使注意力机制即使与3D数据整合成为可能。
本文将Axial attention应用到网络的解码器上,将其运行在转置卷积上采样的输出上,然后将它们相加。

显示了Axial attention解码器块的示意图。即使有了更有效的注意力,作者发现这种方法也不可能应用于最高分辨率的特征(128×128×128),因此只选择了4个较低分辨率的特征。注意力头的数量和每个头的尺寸随着分辨率的降低而增加一倍,分别从4和16(64×64×64分辨率)开始。
1.3 训练策略
这里遵循nnU-Net训练方法。每个网络都接受了5倍交叉验证的训练。在训练过程中,动态地应用数据增强来提高泛化能力。数据增强包括随机旋转和缩放、弹性变形、附加亮度增强和伽玛缩放。
优化的目标是binary entropy loss和Dice loss的总和,计算在最终的全分辨率输出以及在低分辨率的辅助输出。使用Batch Dice loss代替sample Dice loss,将整个批次作为一个样本来计算损失,而不是平均每个样本在小批中的Dice。批量Dice帮助稳定训练通过减少来自样本的错误,少量的注释样本。网络采用Nesterov动量为0.99的随机梯度下降法进行优化。初始学习率为0.01,并按照多项式计划衰减:

每次训练运行持续1000个epoch,每个epoch包含250个小批量。当前折叠验证集上的Dice Score用于监视训练进度。所有实验都是在24GB VRAM的NVIDIA RTX 3090 GPU上使用Pytorch 1.9进行的。开发了以下模型:
-
BL:Baseline nnUNet -
BL+L:Baseline with Large nnUNet -
BL+GN:Baseline with Group Normalization -
BL+AA:Baseline with axial attention, batch normalization -
BL+L+GN:nnUNet with larger Unet, group normalization
2比赛结果


上面论文代码下载
后台回复:冠军方案,即可下载上述论文和代码
后台回复:CVPR2021,即可下载CVPR 2021论文和代码开源的论文合集
后台回复:ICCV2021,即可下载ICCV 2021论文和代码开源的论文合集
后台回复:Transformer综述,即可下载最新的3篇Transformer综述PDF
重磅!医疗影像交流群成立
扫码添加CVer助手,可申请加入CVer-医疗影像 微信交流群,方向已涵盖:目标检测、图像分割、目标跟踪、人脸检测&识别、OCR、姿态估计、超分辨率、SLAM、医疗影像、Re-ID、GAN、NAS、深度估计、自动驾驶、强化学习、车道线检测、模型剪枝&压缩、去噪、去雾、去雨、风格迁移、遥感图像、行为识别、视频理解、图像融合、图像检索、论文投稿&交流、Transformer、PyTorch和TensorFlow等群。
一定要备注:研究方向+地点+学校/公司+昵称(如医疗影像+上海+上交+卡卡),根据格式备注,可更快被通过且邀请进群
▲长按加小助手微信,进交流群
▲点击上方卡片,关注CVer公众号
整理不易,请点赞和在看



