近来,大语言模型在自然语言处理方面展示了令人瞩目的理解和推理能力,促使不同领域纷纷探索这些模型在自己专业领域内的应用潜力。
在生化分子理解领域,分子结构,尤其是3D空间结构,对于理解分子动力学、蛋白质-配体相互作用、酶功能等一系列生化分子性质至关重要。虽然现有的大语言模型通过在广泛的生化文献上的预训练,具有了一定的基于文本的分子理解能力,但是受限于文本输入类型的限制,模型仅能将分子建模为以SMILES式为代表的1D文本序列。因此,理解分子结构信息对于现有语言模型是个较大的挑战。如果能将更多的分子3D结构信息注入现有的语言模型中,可以提升其分子结构理解和性质预测能力。
总结来说,现有的分子-文本多模态研究在以下两方面存在缺失:
·3D 分子-文本对齐。这是为了将 3D 分子表示对齐到 1D文本空间,使得在 1D 文本上训练的大语言模型能理解 3D 空间中的分子。
·3D 分子指令微调。这是为了使大语言模型服从人类的指引,完成 3D 分子相关的多种任务。
近日,来自中国科学技术大学,新加坡国立大学,以及华为的研究团队,针对现有语言模型缺乏理解生化分子3D空间结构能力和专业领域指令遵循能力较差的研究现状,通过设计 3D 分子语言模型,构建3D分子-文本预训练和指令微调数据集,对语言模型进行跨模态对齐和指令微调训练,增强语言模型对分子3D空间结构的理解,同时提升其指令遵循能力。
该论文发表于ICLR 2024: 论文地址:https://arxiv.org/abs/2401.13923 项目地址:https://lsh0520.github.io/3D-MoLM/ 模型简介
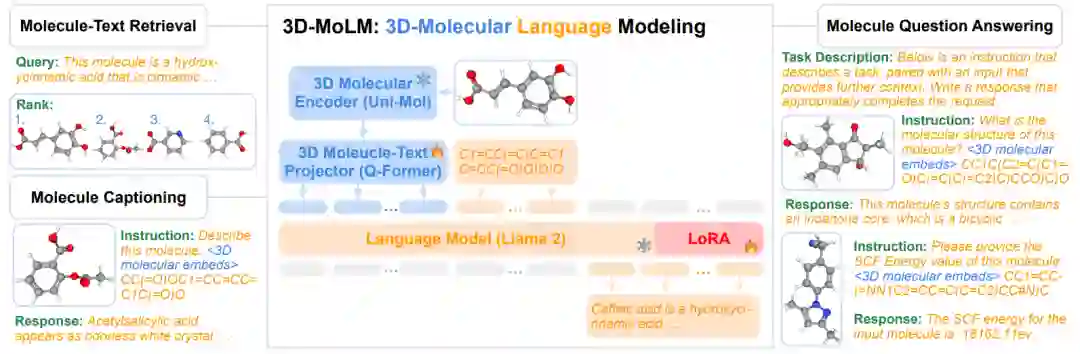
图1:3D-MoLM模型结构
虽然大语言模型已对各个研究领域产生了巨大影响,它们在理解3D分子结构方面存在局限性: 单模态语言模型只能理解1D文本序列。这一局限性极大地限制了大语言模型在生物分子领域的潜力。为了弥补这一差距,我们提出了3D-MoLM: (1)3D-MoLM是一个多功能的3D分子语言模型,可用于分子-文本检索(Molecule-Text Retrieval)、分子描述生成(Molecule Captioning)和分子问答任务(Molecule QA)。 (2)3D-MoLM采用 3D分子-文本projector来弥合3D分子编码器与语言模型之间的模态差异,使得语言模型能够感知 3D分子结构。
训练数据及流程
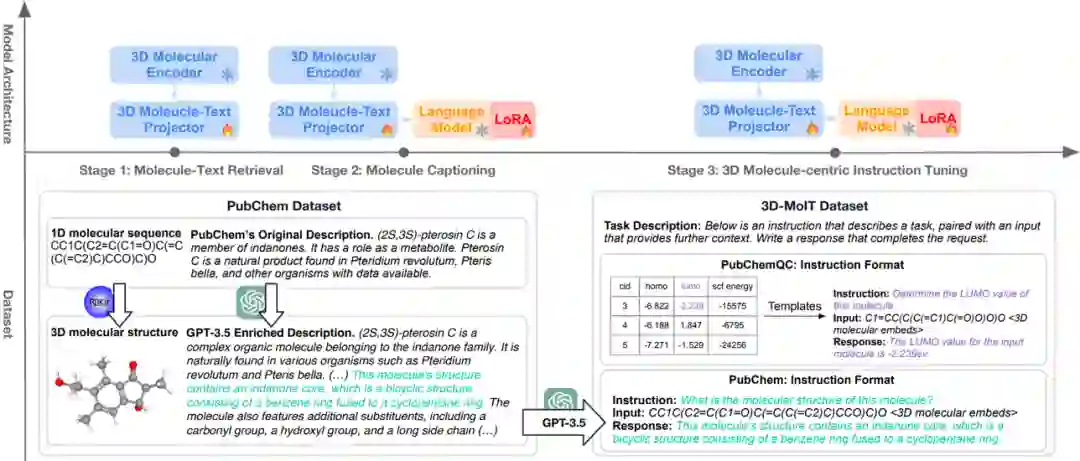
图2:3D-MoLM训练数据与训练流程
为了应对3D分子-文本对齐和以3D分子指令微调这两大挑战,3D-MoLM进行三阶段训练,包括1) 3D分子-文本表示学习,2) 通过文本生成进行3D分子-文本对齐,以及3) 3D分子指令微调。
1)3D分子-文本对齐
现有3D分子编码器通过在3D分子数据上进行预训练,具备了提取具有丰富3D空间特征的分子表征的能力,并且在分子属性预测等下游任务中获得了出色的性能。在这一小节, 我们将其与一个3D分子-文本projector连接, 用作语言模型的 3D 分子感知模块。通过分子-文本对齐训练,我们将3D 分子编码器输出的带有 3D空间结构信息的分子表征经过projector对齐到大语言模型的输入空间,从而加强大语言模型对分子 3D空间结构的感知、理解能力。
数据准备 – PubChem:大量的 3D分子-文本对是有效对齐分子表征空间和语言模型输入空间的关键。我们从PubChem收集分子SMILES和对应的性质描述文本。由于PubChem记录中大量分子的性质描述文本缺乏有效信息,我们使用GPT进行recaption来对文本记录进行增强。另一方面,我们通过在RDKit中运行MMFF算法获得分子的3D结构来构成3D分子-文本对用于对齐训练。
模型结构:我们采用Query Former (Q-Former) 作为 3D分子-文本projector。给定一个分子的 3D分子表征,Q-Former将其转换为与语言模型的输入空间对齐的令牌(tokens)作为软提示词(soft prompt)。这种转换使得语言模型可以理解3D分子结构。Q-Former进行了两个训练阶段——第一阶段专注于分子-文本检索(Retrieval),而第二阶段优化分子的描述生成(Captioning)。

阶段1:3D分子-文本表示学习。在第一阶段,我们将Q-Former与3D分子编码器一起在分子-文本对上进行联合预训练。这个阶段旨在培养Q-Former提取与对应文本相关的分子特征的能力。我们进行多任务训练,包括分子-文本对比学习,分子-文本匹配,和分子的描述生成。具体来说,在分子-文本对比学习中,分子与其对应文本(即正对)之间的相似性与负对的相似性进行对比,目标是最大化分子和对应文本表示之间的互信息。分子-文本匹配任务使用 Q-Former 的 Self-Attention 模块区分匹配和不匹配的分子-文本对,增强跨模态表示的细粒度对齐。在分子的描述生成任务中,Q-Former被训练为基于给定的分子输入生成文本描述。
图4:3D-MoLM 在阶段 2 和阶段 3 的结构图。
阶段2:3D分子的描述生成。在这个阶段,我们将集成了3D分子编码器的Q-Former与语言模型连接起来,通过分子的描述生成任务使Q-Former输出对齐到语言模型的输入空间。Q-Former提取的分子表征令牌作为语言模型可以理解的软提示来指导语言模型生成相应的文本描述。这个阶段联合优化3D分子编码器,3D分子-文本projector,和语言模型,将3D分子表征与语言模型的输入空间对齐,促进跨模态连接。
2)3D分子指令微调
在将3D分子表征与语言模型文本输入空间对齐后,我们进一步对其进行3D分子专业指令微调。它一方面可以强化模型遵循指令的能力,另一方面也准确地激活了它对分子3D结构的感知能力和对应3D属性的理解。
指令微调的核心是构建高质量的指令数据集。我们从两个公开数据库搜集数据来构建指令数据集: 1) PubChem,提供了广泛的分子属性、起源和应用等文本描述; 2) PubChemQC,提供了大量依赖分子3D结构的属性,如 HOMO,LUMO,SCF energy 值等。具体来说,来自 PubChem 的指令数据可以分为两种类型: 数值型属性和描述性文本。其中计算出的分子属性是数值,描述性文本以自由文本的形式描述分子属性。我们选择以下数值型属性: 分子量 (Molecular Weight),疏水常数 (LogP),拓扑极性表面积(TPSA) 和分子复杂性 (Complexity)。我们使用预定义的一组文本模板将这些数值型属性转换为指令格式。对于描述性文本,我们采用 GPT-3.5 阅读分子描述,并为每个分子生成问题-答案对。如此,源自 PubChem 的指令包括各种各样的分子属性。
为了有效激活模型的3D结构感知能力,我们进一步包含了来自PubChemQC 的数值型分子属性。PubChemQC 包含与分子3D结构有强关联的属性,如最高占据分子轨道(HOMO),最低未占据分子轨道 (LUMO),HOMO-LUMO 间隙,和 SCF 能量,这些属性大多不能从 1D或2D分子表示中推断出来。PubChemQC 还包括通常被视为 3D分子结构的准确估计的 DFT 计算得出的3D分子结构数据。
实验结果
为了评估3D-MoLM的有效性,我们在分子-文本检索(Molecule-Text Retrieval)、分子描述生成(Molecule Captioning)和分子问答任务(Molecule QA)上测试3D-MoLM的能力:
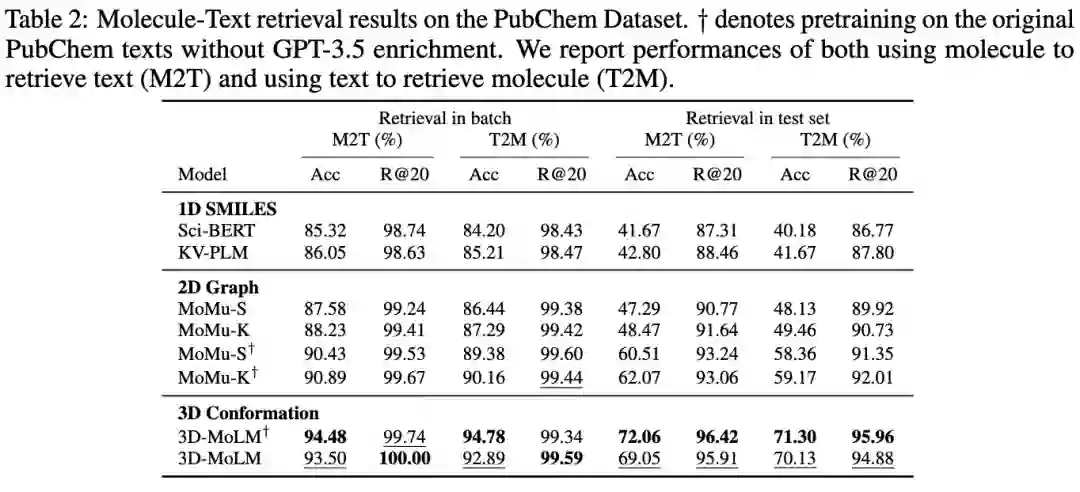
表1:分子-文本检索(Molecule-Text Retrieval)
表2:分子描述生成(Molecule Captioning)
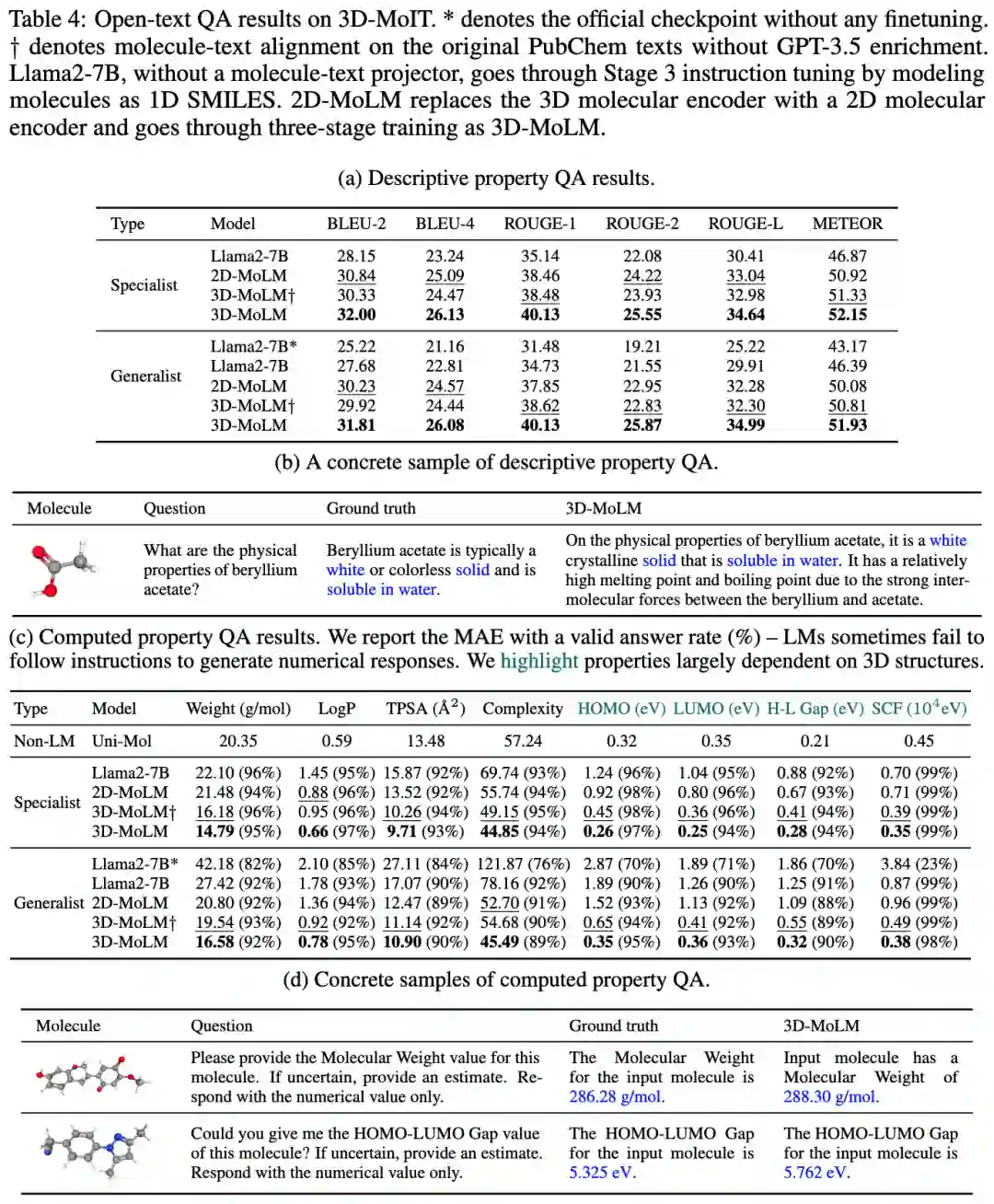
表3:分子问答任务(Molecule QA)
未来展望
在本文中,我们提出了3D-MoLM,旨在促进大语言模型对 3D分子表示的跨模态理解。尽管所提出的方法在各种任务上展示了有效性,我们仍然指出以下局限性和可能的未来探索方向:(1)扩展到更多的分子-文本建模任务,(2)细粒度的 3D分子-文本对齐,(3)数据集规模。更详细的讨论请见论文原文。