我们探讨了如何最优地训练蛋白质语言模型,这一领域在生物研究中备受关注,但有关最佳实践的指导较为有限。大多数模型在训练时耗费大量计算资源,直到性能增益趋于平稳,主要集中在增加模型规模,而非优化平衡性能和计算预算的高效计算边界。我们的研究基于包含 9.39 亿条蛋白质序列的大规模数据集。我们训练了超过 300 个模型,参数规模从 350 万到 107 亿不等,训练的独特标记数从 50 亿到 2000 亿,以探讨模型规模、训练标记数量和目标之间的关系。
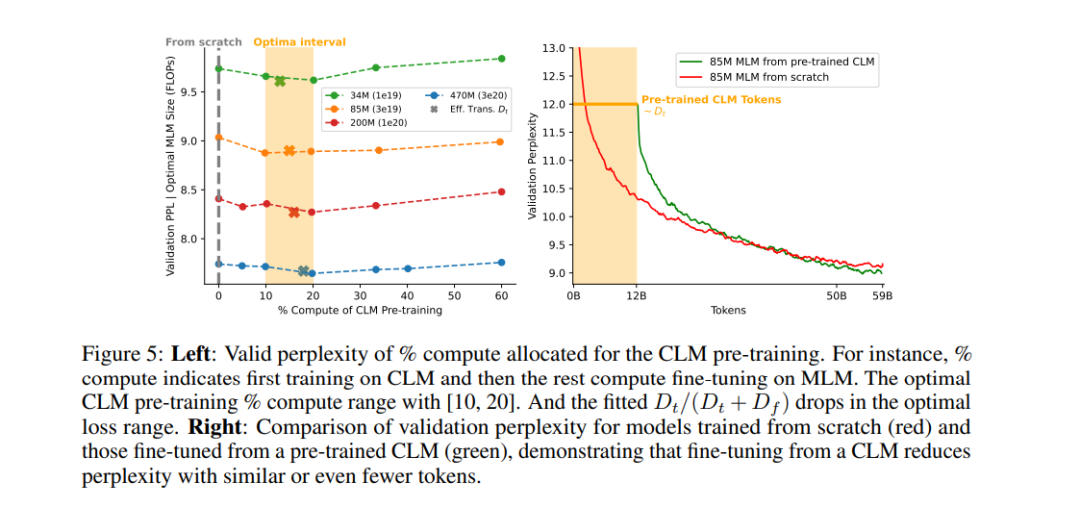
首先,我们观察到因果语言模型(CLM)的收益递减效应,以及在重复使用常见的 Uniref 数据库时,掩码语言模型(MLM)的过拟合现象。为了解决这一问题,我们在训练集中加入了宏基因组蛋白质序列,以增加多样性,避免性能的平稳或过拟合现象。其次,我们获得了基于 Transformer 的 CLM 和 MLM 的扩展规律,这些规律针对蛋白质序列数据的特定特性进行了优化。第三,我们观察到从 CLM 到 MLM 的迁移扩展现象,通过基于有效迁移标记数量的扩展行为进一步证明了迁移的有效性。
最后,为验证我们的扩展规律,我们将大规模版本的 ESM-2 和 PROGEN2 在下游任务上进行比较,涵盖蛋白质生成以及与结构和功能相关的任务评估,所有测试均在不超过或相等的预训练计算预算内完成。
成为VIP会员查看完整内容
相关内容
Arxiv
1+阅读 · 2024年12月18日
Arxiv
42+阅读 · 2023年4月19日
Arxiv
219+阅读 · 2023年4月7日