没数据,没SPSS,如何轻松获得生存分析结果?| 推广
生存分析,顾名思义是用来研究个体的存活概率与时间的关系的方法。通俗来讲,“一个人可以活多久”这个问题,纵是扁鹊华佗在世亦不能做解,而利用现有样本的生存资料,通过生存分析即可推断样本活过一定时间的概率。
生存分析本质上就是个回归分析【回归分析?简单来说,就是给定一些已知样本的自变量X和对应的结果变量Y,回归分析算法会计算出Y和X之间的关系公式。比如最简单的一元线性回归算法,公式是这个样子:Y=aX+b。再来一个新的样本,得知其自变量X的情况,结果Y自然就预测出来了】。那么问题来了,为什么不用普通的回归算法推断生存概率呢?因为生存分析中的结果变量有点不一样,它不仅考察生存事件的结果,同时还要考察事件出现的时间。也就是说,生存分析的结果变量有两个,一个是事件状态,一个是时间。
临床研究中通常指患者死亡或疾病复发等。敲黑板!SPSS分析时定义事件要选死亡!
普通回归算法只能hold住完全数据,即发病到死亡的时间完整记录;由于失访或因其他原因导致的死亡(删失数据),或随访结束时观察对象仍存活而无法获得其生存时间(截尾数据),都是不完全数据,这时候普通回归算法就搞不定啦
生存分析常用的方法有寿命表法、Kaplan-Meier法和Cox回归。在仅考虑单因素的情况下,通常多用Kaplan-Meier法(如图1);若需要综合分析多个因素对生存时间分布的影响,一般使用Cox回归来分析。生存分析可以用的工具也有不少,比如SPSS、R和SAS。SPSS的教程推荐下面这个,目前小编看过的最好的SPSS教程和结果解读,没有之一:
https://www.mediecogroup.com/method_article_detail/31/
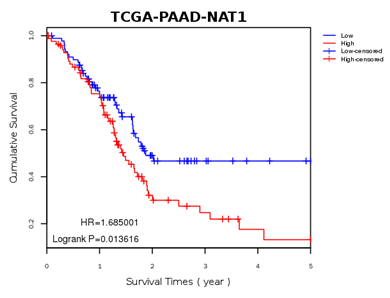
图1 Gene Matrix 绘制的生存分析函数图
图1 比较了Low和High两组之间的生存时间分布。HR是风险比,指的是实验组(High)所产生的风险率与对照组(Low)所产生的风险率的比值;Logrank指的是生存分析所使用的显著性检验方法。
生存分析最重要的还是样本,临床样本不足是一个令大多数临床医生头疼的事情。再推荐一个数据库,The Cancer Genome Atlas (TCGA),目前最为综合全面的癌症病人相关组学数据库之一,收录了33种人类癌症的临床病理数据、mRNA表达量、miRNA表达量、甲基化等各种数据。现在 TCGA 已并入 Genomic Data Commons Data Portal(GDC),数据下载烦烦烦,所幸样本并没有什么增加,下载数据还可以去下面这个链接偷个懒:
http://gdac.broadinstitute.org/runs/stddata__2016_01_28/data/
数据下载可以偷懒,后续的数据整理就是硬功夫了。高低表达、癌和癌旁、临床病理数据、生存数据等等都需要人工处理。此外,发表论文还需要花钱买个正版的SPSS软件。鉴于前番提及的种种困难,吉凯基因云平台 Gene Matrix 隆重推出新应用:TCGA生存分析。不用下数据,无需买软件,打开应用市场,选择TCGA生存分析,输入基因名和癌症类型,点击提交,漂亮的生存函数图就出来了!有了 Gene Matrix 傍身,生存分析就是这么简单,你只需跨出注册的第一步,剩下的 999 步,Gene Matrix 都替你走完!扫描下方二维码,马上注册登录,免费解锁功能。
图2 TCGA生存分析应用图标
任何生信相关的事儿,有事儿没事儿聊一发
📞 021-5132 0189,分机号:8179、8657、8039
📧 bioinfo@genechem.com.cn