R语言二分类问题案例分析:以泰坦尼克号沉船为例
作者:黄天元,复旦大学博士在读,目前研究涉及文本挖掘、社交网络分析和机器学习等。希望与大家分享学习经验,推广并加深R语言在业界的应用。
微信:hope9057
Kaggle上最经典的泰坦尼克号入门级教程,我们这里尝试玩转它(https://www.kaggle.com/c/titanic)。先讲数据背景,我们有各种各样的乘客数据,想要利用这些数据,预测在泰坦尼克号沉船的时候,这个乘客是否能够存活。具体的数据字典可以参照:
https://www.kaggle.com/c/titanic/data。
先导入数据
#数据导入 set.seed(201891) library(pacman) p_load(tidyverse) p_load(caret,caretEnsemble) setwd("E:\\_data_hope\\Titanic\\data") read_csv("train.csv") -> train_raw1 read_csv("test.csv") -> test_raw1 read_csv("gender_submission.csv") -> gs
人工变量筛选
人工筛选变量是第一步,这是机器学习无法逾越的高度,因为我们知道哪些变量是真正“有关”的,哪些即使是真的提高了预测精度也只是假象而已。我们应该知道,乘客的ID号码,乘客叫什么名字,乘客在哪里上船,还有买票的号码,是与存活率完全没有直接关系的,直接删除掉。
train_raw1 %>% select(-PassengerId,-Name,-Ticket,-Embarked) -> train_raw2 test_raw1 %>% select(-PassengerId,-Name,-Ticket,-Embarked) -> test_raw2
缺失值可视化和处理
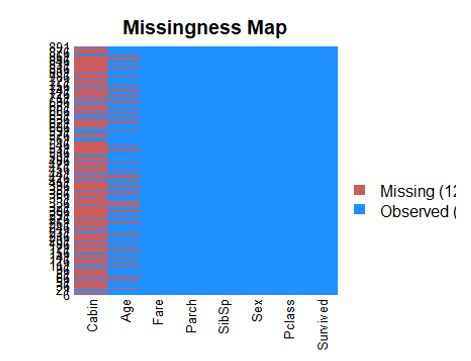
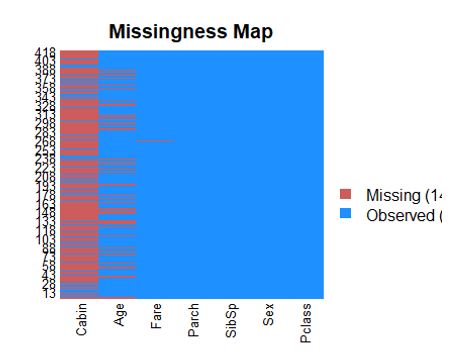
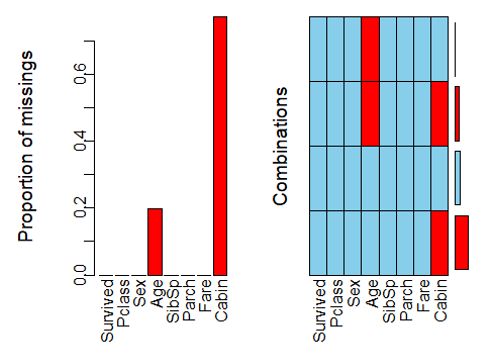
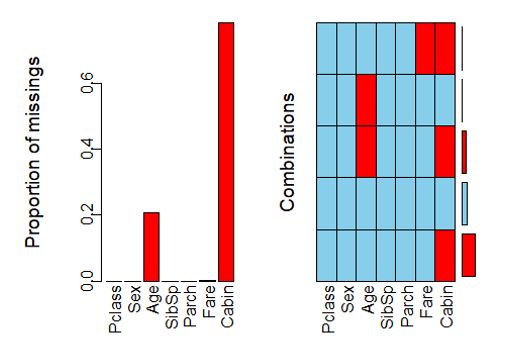
如果数据中有一些属性含有大量缺失值,那么它对预测的贡献几乎为零,甚至具有不良的干扰。当然有的时候缺和不缺本来就是一种信息,但是这里我们无法深入判断。首先我们先看看是否有缺失值,有的话缺多少?
p_load(VIM,Amelia) missmap(train_raw2)
missmap(test_raw2)
train_raw2 %>% aggr()
test_raw2 %>% aggr()
Cabin,也就是舱位号码缺了很多,因此我们应该直接删除掉整列。年龄数据存在缺失,但是缺失比例不大,而且年龄可能会提供重要信息,所以需要保留。能够直接删除缺失行吗?答案是不行,因为待预测的验证集包含有缺失值,因此必须对它们进行必要的处理才行。
这个例子中,我倾向于使用KNN插值法,原理就是,相似的乘客可能会有相同的年纪。需要注意的是,KNN插值法不允许变量中包含有非数值型变量,因此这里直接先转为因子再转为数值。性别只有两个,因此没有关系,直接化为因子就可以。如果有多于两个的因子,应该先用one-hot encoding这种方法把它化为稀疏矩阵再来做。
p_load(DMwR) #KNN插值法需要用的包 train_raw2 %>% select(-Cabin) -> train1 test_raw2 %>% select(-Cabin) -> test1 train1 %>% mutate(Sex=as.numeric(as.factor(Sex))) %>% as.data.frame() %>% knnImputation() %>% pull(Age) -> train_age test1 %>% mutate(Sex=as.numeric(as.factor(Sex))) %>% as.data.frame() %>% knnImputation() %>% pull(Age) -> test_age train1 %>% mutate(Age=train_age) -> train.wash test1 %>% mutate(Age=test_age) -> test.wash
这样一来我们就得到了清洗好的训练集train.wash和测试集test.wash。
零模型:探索模型的表现的基准
一般建模之初,应该设定两个模型:零模型与全模型。零模型即随机猜测我们能够得到的正确率。什么?你认为是50%?这不对,虽然我们最终结果只有存活和不存活,但是因为样本中存活和非存活的比例不同,因此需要特殊对待。
train.wash %>% count(Survived) %>% mutate(n/sum(n)) ## # A tibble: 2 x 3 ## Survived n `n/sum(n)` ## <int> <int> <dbl> ## 10 549 0.616 ## 21 342 0.384 gs %>% count(Survived) %>% mutate(n/sum(n)) ## # A tibble: 2 x 3 ## Survived n `n/sum(n)` ## <int> <int> <dbl> ## 10 266 0.636 ## 21 152 0.364
我们可以看到,有61.6%的乘客最后不能存活,38.4%的乘客可以存活。也就是我们对任意一个乘客都假设他不能够存活,我们就会得到61.6%的准确率。如果我们的模型在训练集中最后准确率不能够超越这个数值,那么就白忙一场了。
在验证集中也一样,如果最终我们的accuracy没有超越63.6%,那么还不如瞎猜这个乘客肯定不能够存活更好。
模型选择
首先,我们的问题数据量不大,我们看看样本量多少。
train.wash ## # A tibble: 891 x 7 ## Survived Pclass Sex Age SibSp Parch Fare ## <int> <int> <chr> <dbl> <int> <int> <dbl> ## 1 0 3 male 22 1 0 7.25 ## 2 1 1 female 38 1 0 71.3 ## 3 1 3 female 26 0 0 7.92 ## 4 1 1 female 35 1 0 53.1 ## 5 0 3 male 35 0 0 8.05 ## 6 0 3 male 27.1 0 0 8.46 ## 7 0 1 male 54 0 0 51.9 ## 8 0 3 male 2 3 1 21.1 ## 9 1 3 female 27 0 2 11.1 ## 10 1 2 female 14 1 0 30.1 ## # ... with 881 more rows
891个样本量的时候,我们决定进行三折交叉验证,不过尝试进行重复的交叉验证,这里我们先重复五次,设定如下:
ctrl= trainControl(method = "repeatedcv",number = 3,repeats=5,search="random", summaryFunction = twoClassSummary, classProbs = TRUE, savePredictions = "final")
注意我们用了search=“random”,从而采取了随机超参数搜索,对于一些模型来说设置网格比较费时,我们先看个大概,因此采用这种方法。需要注意的是,建模前最好把所有变量都转化为数值变量,计算机只认得数字,任何情况都是如此,就算有字符串也是转为因此变量再来做的,我们这里就先转化为因子变量来做。
train.wash %>% mutate(Sex=as.factor(Sex)) %>% mutate(Survived=ifelse(Survived==1,"Alive","Dead")) -> train test.wash %>% mutate(Sex=as.factor(Sex)) -> test gs %>% mutate(Survived=ifelse(Survived==1,"Alive","Dead")) -> gs
能够进行二分类的模型非常多,大类是线性和非线性。线性一般来说解释性强但是效果一般,非线性效果好一点但是解释性弱一点,而且容易出现过拟合。我们用零模型设定了基准,这里我们广泛采用不同的模型看看哪个表现更好。采用的线性模型包括:逻辑回归(glm)、具有惩罚项的逻辑回归(glmnet)、偏最小二乘判别分析(pls)、线性判别分析(lda)和PAM模型(pam)来做;非线性模型包括:非线性判别(mda)、神经网络(nnet)、灵活判别分析(fda)、支持向量机(svm)、K近邻(KNN)、朴素贝叶斯(nb)、随机森林(rf)还有大名鼎鼎的Xgboost(xgbLinear/xgbTree)。需要注意的是,这里神经网络就是三层的全连接神经网络,这个问题还没有如此有“深度”,因此还没有涉及深度学习的领域。为了能够一下子拟合所有模型,我们祭出caretEnsemble::caretList这个利器。这样我们可以对各种模型做一个初筛,虽然只能方便地比较训练集而不是把测试集一起比较了,但是尽管在训练集表现好不一定在测试集表现就好,但是在训练集表现不好的在测试集一般来说一定就不太好。
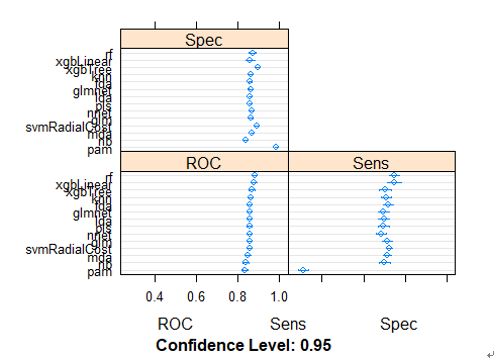
model_list=caretList( Survived~.,data=train, trControl=ctrl, metric="ROC", preProcess=c("center","scale"), methodList=c("glm","glmnet","pls","lda","pam", "mda","fda","svmRadialCost","knn","nb","rf","xgbLinear","xgbTree"), tuneList = list(nnet=caretModelSpec(method="nnet",trace=F)) ) ## 1234567891011121314151617181920212223242526272829301111111111111111 results <- resamples(model_list) summary(results) ## ## Call: ## summary.resamples(object = results) ## ## Models: nnet, glm, glmnet, pls, lda, pam, mda, fda, svmRadialCost, knn, nb, rf, xgbLinear, xgbTree ## Number of resamples: 15 ## ## ROC ## Min. 1st Qu. Median Mean 3rd Qu. Max. ## nnet 0.8129374 0.8509251 0.8606078 0.8582015 0.8679657 0.9011121 ## glm 0.8117390 0.8493433 0.8603921 0.8581664 0.8661202 0.9032212 ## glmnet 0.8134647 0.8514284 0.8602243 0.8585722 0.8672946 0.9025022 ## pls 0.8159093 0.8515842 0.8592657 0.8583757 0.8660004 0.9016873 ## lda 0.8167242 0.8518958 0.8597929 0.8584827 0.8661442 0.9012559 ## pam 0.7858067 0.8249569 0.8399722 0.8366248 0.8498346 0.8795897 ## mda 0.8059870 0.8384503 0.8553590 0.8514077 0.8654252 0.8974691 ## fda 0.8157655 0.8521474 0.8620458 0.8590595 0.8693917 0.9042997 ## svmRadialCost 0.8175630 0.8466710 0.8583549 0.8574521 0.8698471 0.9058096 ## knn 0.8234829 0.8474140 0.8626929 0.8630508 0.8763901 0.9067683 ## nb 0.7802943 0.8309007 0.8414582 0.8397006 0.8490916 0.8993864 ## rf 0.8379829 0.8630045 0.8843352 0.8818362 0.8948687 0.9349295 ## xgbLinear 0.8255201 0.8616743 0.8763302 0.8782395 0.8927835 0.9344262 ## xgbTree 0.8233870 0.8598289 0.8675822 0.8697121 0.8826934 0.9328204 ## NA's ## nnet 0 ## glm 0 ## glmnet 0 ## pls 0 ## lda 0 ## pam 0 ## mda 0 ## fda 0 ## svmRadialCost 0 ## knn 0 ## nb 0 ## rf 0 ## xgbLinear 0 ## xgbTree 0 ## ## Sens ## Min. 1st Qu. Median Mean 3rd Qu. Max. ## nnet 0.6140351 0.6622807 0.6842105 0.6847953 0.7105263 0.7543860 ## glm 0.6491228 0.6842105 0.7105263 0.7128655 0.7280702 0.8333333 ## glmnet 0.6315789 0.6710526 0.6929825 0.6964912 0.7149123 0.8070175 ## pls 0.6140351 0.6710526 0.7017544 0.6970760 0.7149123 0.8070175 ## lda 0.6228070 0.6710526 0.7017544 0.6988304 0.7192982 0.8070175 ## pam 0.2368421 0.2807018 0.3070175 0.3087719 0.3333333 0.3947368 ## mda 0.6754386 0.6842105 0.7105263 0.7140351 0.7368421 0.7982456 ## fda 0.6491228 0.6842105 0.7105263 0.7187135 0.7368421 0.8245614 ## svmRadialCost 0.6842105 0.7017544 0.7192982 0.7239766 0.7543860 0.7631579 ## knn 0.6491228 0.6842105 0.7017544 0.7093567 0.7324561 0.7894737 ## nb 0.6315789 0.6754386 0.6929825 0.7011696 0.7324561 0.7807018 ## rf 0.6666667 0.7105263 0.7543860 0.7485380 0.7850877 0.8333333 ## xgbLinear 0.6842105 0.7017544 0.7280702 0.7485380 0.7982456 0.8508772 ## xgbTree 0.6315789 0.6666667 0.6929825 0.7029240 0.7280702 0.8421053 ## NA's ## nnet 0 ## glm 0 ## glmnet 0 ## pls 0 ## lda 0 ## pam 0 ## mda 0 ## fda 0 ## svmRadialCost 0 ## knn 0 ## nb 0 ## rf 0 ## xgbLinear 0 ## xgbTree 0 ## ## Spec ## Min. 1st Qu. Median Mean 3rd Qu. Max. ## nnet 0.8469945 0.8633880 0.8688525 0.8699454 0.8797814 0.8961749 ## glm 0.8251366 0.8579235 0.8633880 0.8633880 0.8743169 0.8852459 ## glmnet 0.8360656 0.8551913 0.8633880 0.8652095 0.8797814 0.8907104 ## pls 0.8415301 0.8497268 0.8633880 0.8601093 0.8688525 0.8797814 ## lda 0.8415301 0.8497268 0.8579235 0.8586521 0.8688525 0.8743169 ## pam 0.9508197 0.9781421 0.9890710 0.9857923 0.9945355 1.0000000 ## mda 0.8524590 0.8579235 0.8743169 0.8703097 0.8743169 0.8961749 ## fda 0.8360656 0.8524590 0.8633880 0.8619308 0.8743169 0.8852459 ## svmRadialCost 0.8524590 0.8743169 0.8907104 0.8918033 0.9071038 0.9289617 ## knn 0.8415301 0.8497268 0.8633880 0.8637523 0.8743169 0.8961749 ## nb 0.7978142 0.8251366 0.8469945 0.8404372 0.8524590 0.8743169 ## rf 0.8306011 0.8469945 0.8743169 0.8721311 0.8879781 0.9289617 ## xgbLinear 0.7868852 0.8469945 0.8688525 0.8619308 0.8825137 0.9125683 ## xgbTree 0.8743169 0.8825137 0.8961749 0.8965392 0.9098361 0.9234973 ## NA's ## nnet 0 ## glm 0 ## glmnet 0 ## pls 0 ## lda 0 ## pam 0 ## mda 0 ## fda 0 ## svmRadialCost 0 ## knn 0 ## nb 0 ## rf 0 ## xgbLinear 0 ## xgbTree 0 dotplot(results)
# correlation between results modelCor(results) ## nnet glm glmnet pls lda pam ## nnet 1.0000000 0.9964623 0.9985821 0.9959158 0.9959797 0.9740745 ## glm 0.9964623 1.0000000 0.9983260 0.9948820 0.9955163 0.9594457 ## glmnet 0.9985821 0.9983260 1.0000000 0.9980440 0.9981462 0.9659061 ## pls 0.9959158 0.9948820 0.9980440 1.0000000 0.9997897 0.9642498 ## lda 0.9959797 0.9955163 0.9981462 0.9997897 1.0000000 0.9635982 ## pam 0.9740745 0.9594457 0.9659061 0.9642498 0.9635982 1.0000000 ## mda 0.9173182 0.9373646 0.9223877 0.9055397 0.9077165 0.8484849 ## fda 0.9466791 0.9473209 0.9505168 0.9481600 0.9463399 0.9136922 ## svmRadialCost 0.6297685 0.6462695 0.6458028 0.6397843 0.6367630 0.5758754 ## knn 0.8617429 0.8809390 0.8734330 0.8571032 0.8584613 0.8115088 ## nb 0.8887790 0.8916700 0.8939506 0.8849860 0.8812802 0.8525138 ## rf 0.8374398 0.8593235 0.8511846 0.8363713 0.8369300 0.7998105 ## xgbLinear 0.8295344 0.8349773 0.8296049 0.8133293 0.8109480 0.8317105 ## xgbTree 0.9298302 0.9301195 0.9347325 0.9292743 0.9271813 0.9103171 ## mda fda svmRadialCost knn nb ## nnet 0.9173182 0.9466791 0.6297685 0.8617429 0.8887790 ## glm 0.9373646 0.9473209 0.6462695 0.8809390 0.8916700 ## glmnet 0.9223877 0.9505168 0.6458028 0.8734330 0.8939506 ## pls 0.9055397 0.9481600 0.6397843 0.8571032 0.8849860 ## lda 0.9077165 0.9463399 0.6367630 0.8584613 0.8812802 ## pam 0.8484849 0.9136922 0.5758754 0.8115088 0.8525138 ## mda 1.0000000 0.9161242 0.6471664 0.8950366 0.8427981 ## fda 0.9161242 1.0000000 0.6160408 0.8759144 0.9175588 ## svmRadialCost 0.6471664 0.6160408 1.0000000 0.7903877 0.6854170 ## knn 0.8950366 0.8759144 0.7903877 1.0000000 0.8883843 ## nb 0.8427981 0.9175588 0.6854170 0.8883843 1.0000000 ## rf 0.8622639 0.8366536 0.7694248 0.9629252 0.8754177 ## xgbLinear 0.8163424 0.8056738 0.7492483 0.9250997 0.8380506 ## xgbTree 0.8711245 0.8857206 0.7031082 0.8450892 0.8545409 ## rf xgbLinear xgbTree ## nnet 0.8374398 0.8295344 0.9298302 ## glm 0.8593235 0.8349773 0.9301195 ## glmnet 0.8511846 0.8296049 0.9347325 ## pls 0.8363713 0.8133293 0.9292743 ## lda 0.8369300 0.8109480 0.9271813 ## pam 0.7998105 0.8317105 0.9103171 ## mda 0.8622639 0.8163424 0.8711245 ## fda 0.8366536 0.8056738 0.8857206 ## svmRadialCost 0.7694248 0.7492483 0.7031082 ## knn 0.9629252 0.9250997 0.8450892 ## nb 0.8754177 0.8380506 0.8545409 ## rf 1.0000000 0.9312219 0.8407419 ## xgbLinear 0.9312219 1.0000000 0.8413261 ## xgbTree 0.8407419 0.8413261 1.0000000 splom(results)
筛选发现,所有模型准确率大致都在0.83~0.89之间,不会相差太大。其中,基于决策树的模型表现比较好,以随机森林为最好,其次是xgbLinear。不过,我们发现基于决策树之间的结果相关性比较大,但是它们与KNN、朴素贝叶斯、PAM方法相关性比较弱,于是我们决定要进行集成学习(Ensemble);其中KNN和PAM相关性比较强,我们仅采用其中ROC值更高的KNN模型。主模型采用随机森林(rf),辅助模型采用KNN,NaiveBayes。目前我们单独采用随机森林能够达到的ROC值(AUC)为0.8875979。希望经过集成学习后能够突破它。
集成学习
对模型进行初筛之后,我们来确定一下模型列表:
model_list2=caretList( Survived~.,data=train, trControl=ctrl, metric="ROC", preProcess=c("center","scale"), methodList=c("rf","nb","knn") )
然后,我们进行集成学习建模。因为是二分类问题,我们用逻辑回归glm来进行集成学习。
glm_ensemble <- caretStack( model_list2, method="glm", metric="ROC", trControl=trainControl( method="boot", number=10, savePredictions="final", classProbs=TRUE, summaryFunction=twoClassSummary ) ) glm_ensemble ## A glm ensemble of 2 base models: rf, nb, knn ## ## Ensemble results: ## Generalized Linear Model ## ## 4455 samples ## 3 predictor ## 2 classes: 'Alive', 'Dead' ## ## No pre-processing ## Resampling: Bootstrapped (10 reps) ## Summary of sample sizes: 4455, 4455, 4455, 4455, 4455, 4455, ... ## Resampling results: ## ## ROC Sens Spec ## 0.8784721 0.7300053 0.8954497
这个结果中集成学习还不如单纯用随机森林得到的效果好。注意每次运行都有随机性,所以结果是不唯一的。我们这里不set.seed,但是需要知道每次的结果都不尽相同,但是一般来说集成学习都会提高总体的准确率。
验证
目前我们已经确定了模型,首先我们认为随机森林模型是比较好的;其次我们认为以随机森林为主,辅助以KNN和朴素贝叶斯方法有提高模型表现的可能,因此要用集成学习方法。在验证阶段,我们需要构建随机森林模型和它的集成模型,并比较两种方法的效果。
test %>% mutate(PassengerId=test_raw1$PassengerId) %>% na.omit -> new.test predict(glm_ensemble,newdata=new.test) -> pre.ensemble predict(model_list2[["rf"]],newdata=new.test) -> pre.rf new.test %>% mutate(rf=pre.rf,ensemble=pre.ensemble) %>% select(PassengerId,rf,ensemble) %>% left_join(gs) %>% mutate_all(funs(as.factor(as.character(.))))-> pre ## Joining, by = "PassengerId" confusionMatrix(pre$rf,pre$Survived) ## Confusion Matrix and Statistics ## ## Reference ## Prediction Alive Dead ## Alive 112 31 ## Dead 40 234 ## ## Accuracy : 0.8297 ## 95% CI : (0.7902, 0.8646) ## No Information Rate : 0.6355 ## P-Value [Acc > NIR] : <2e-16 ## ## Kappa : 0.6278 ## Mcnemar's Test P-Value : 0.3424 ## ## Sensitivity : 0.7368 ## Specificity : 0.8830 ## Pos Pred Value : 0.7832 ## Neg Pred Value : 0.8540 ## Prevalence : 0.3645 ## Detection Rate : 0.2686 ## Detection Prevalence : 0.3429 ## Balanced Accuracy : 0.8099 ## ## 'Positive' Class : Alive ## confusionMatrix(pre$ensemble,pre$Survived) ## Confusion Matrix and Statistics ## ## Reference ## Prediction Alive Dead ## Alive 36 244 ## Dead 116 21 ## ## Accuracy : 0.1367 ## 95% CI : (0.1052, 0.1734) ## No Information Rate : 0.6355 ## P-Value [Acc > NIR] : 1 ## ## Kappa : -0.5798 ## Mcnemar's Test P-Value : 2.179e-11 ## ## Sensitivity : 0.23684 ## Specificity : 0.07925 ## Pos Pred Value : 0.12857 ## Neg Pred Value : 0.15328 ## Prevalence : 0.36451 ## Detection Rate : 0.08633 ## Detection Prevalence : 0.67146 ## Balanced Accuracy : 0.15804 ## ## 'Positive' Class : Alive ##
在验证集中,我们发现集成学习出现了严重的过拟合现象,不如单纯使用随机森林的效果好。这里其实我没有对模型的超参数进行调整,因为我认为这个准确率已经能够接受,其实可以让模型自动再对超参数进行优化,可能会得到更好的效果。继续做下去的话,就是选定随机森林之后对我们的模型进行进一步超参数的调整。
发现网上有人能做到百分百,其实这是完全没有意义的。泰坦尼克号案例就是学习用的,具体应用场景我能够想到的,就是保险业,给每个人投保的时候需要考虑乘客的存活率。不过泰坦尼克的例子已经是多年以前了,现在能够拿到的乘客信息比以前要多得多,更加精细,在具体问题的时候我们还是要不断调整我们的模型。
公众号后台回复关键字即可学习
回复 爬虫 爬虫三大案例实战
回复 Python 1小时破冰入门回复 数据挖掘 R语言入门及数据挖掘
回复 人工智能 三个月入门人工智能
回复 数据分析师 数据分析师成长之路
回复 机器学习 机器学习的商业应用
回复 数据科学 数据科学实战
回复 常用算法 常用数据挖掘算法