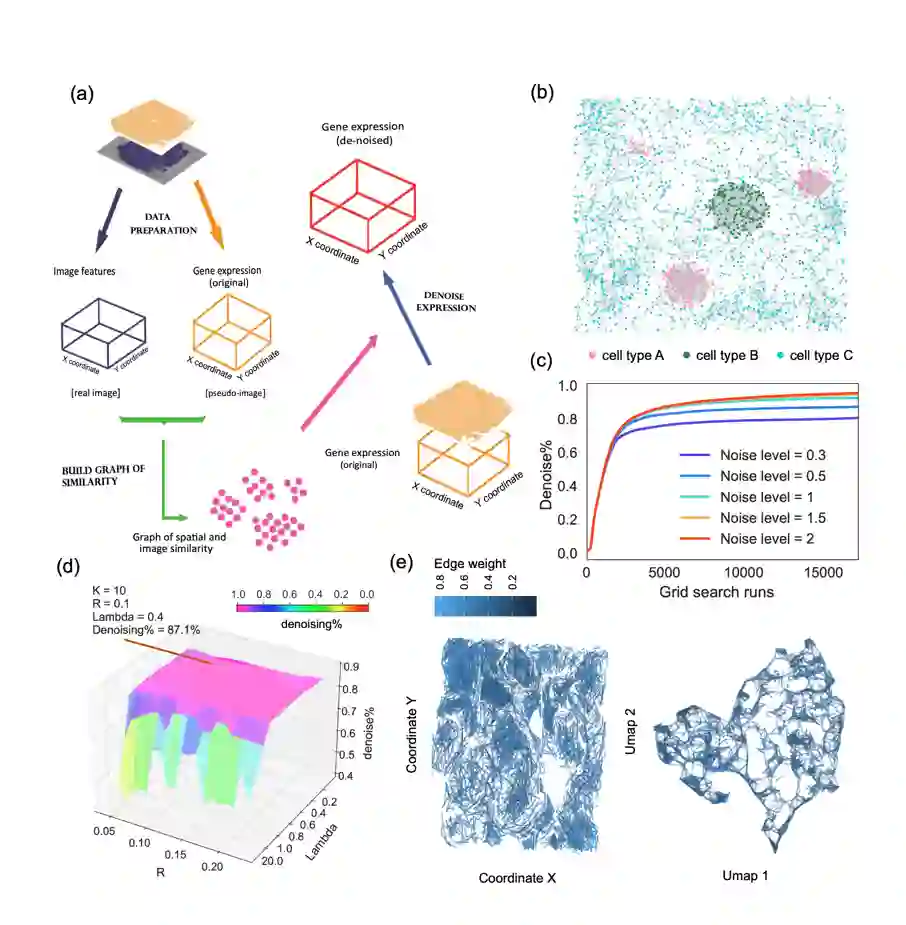
2022年8月4日,来自美国德州大学西南医学中心(UT Southwestern Medical Center)定量生物学研究中心(Quantitative Biomedical Research Center)的王涛博士团队(Tao Wang Lab)和德州大学阿灵顿分校(UT Arlington)的王莉教授在Nature Methods(《自然-方法》)杂志上发表了一篇题为 Sprod for De-noising Spatial Transcriptomics Data Based on Position and Image Information 的文章。该团队报告了他们发明的Sprod方法,其使用空间转录组数据特有的空间位置和病理学图像的信息,来修正空间转录组基因表达谱数据中的噪音。
空间转录组(spatial transcriptomics)是近年来越来越热门的高通量测序技术。空间转录组能够在提供类似于单细胞测序数据的基因表达谱数据的同时,提供测序的位置信息甚至对应的病理学图像数据。然而,空间转录组技术(特别是最新的高分辨率空间转录组技术)的基因表达谱数据中含有大量的噪音。这些噪音来自于每个测序位点上摊薄的低通量测序深度以及为了保留测序位置所执行的额外实验步骤。这些噪音包括但是不限于单细胞测序数据中的drop-out现象。这些噪音给研究者们从宝贵的空间转录组数据提取出有价值的信息制造了巨大的障碍。
在Sprod的降噪过程中,测序的每个位点会向附近的位点借用基因表达信息。在病理学图像上(有图像的空间转录组技术,如Visium)或者总体转录谱上(Overall transcriptomics profile,针对没有图像的空间转录组技术,如Slide-Seq)更相似且测序位置相邻的位点,会互相借用更多的信息。基于这个原理,Sprod构建了一个隐图模型(Latent Graph Model, Fig 1),通过分析不同测序位点的空间距离和基因表达谱特征,将测序位点放入这个隐图中。空间转录组的表达谱信息根据这个隐图流动,从而实现表达谱数据的降噪。Sprod可以应用于各种空间转录组技术,如Visium、 Slide-Seq、HDST、Seq-Scope等。分辨率越高的技术类型,噪音越大,Sprod的作用也越大。
Fig 1:Sprod软件内部数学模型的工作原理
王涛课题组接下来在不同的空间转录组技术数据集上,验证了Sprod降噪的可靠程度。比如,在Fig. 2中,王涛课题组展示了在一个卵巢癌的Visium数据集上使用Sprod降噪前后的效果。这个数据集提供了与空间转录组对应的免疫荧光染色(IF)图像,其中包括CD45。如Fig. 2左所示,CD45 IF和Visium数据中的基因PTPRC (PTPRC的翻译产物为CD45)的RNA表达数据吻合度非常差,但在使用了Sprod之后(出于独立验证的目的,IF图像的CD45 channel在Sprod的计算过程中被剔除了),PTPRC的基因表达量和CD45的染色强度有了很好的重合。此外,王涛课题组还将Sprod与scRNA-seq数据分析中常用的drop-out矫正方法(scImpute和SAVER)做了比较,并显示出Sprod在降噪效果上优于scImpute和SAVER。
Fig 2:PTPRC的基因表达量和CD45的免疫荧光染色的吻合度。左:原始Visium数据;右:Sprod降噪之后的数据
王涛课题组进而将Sprod应用到了一系列的Visium, Slide-Seq, Seq-Scope等其他空间转录组数据集上,验证了Sprod能够给各种数据有效降噪。降噪后的数据在差异表达分析(differential expression),信号通路富集(pathway enrichment),细胞间通讯(cell-to-cell communications)等各方面的下游分析中,都有了生物学上更合理(biologically relevant)的结果。单细胞测序数据的drop-out矫正方法单纯地利用表达谱本身来矫正表达谱数据中的问题。这会造成一种类似于过拟合(overfitting)或者过平滑(oversmoothing)的现象,因此在学界受到了一些批评。与之不同的是,Sprod利用的是空间转录组数据中独有的测序位置和病例图像的信息。通过这样的独立的信息,Sprod得以更精确的执行降噪操作。
总而言之,空间转录组技术给生物医学研究提供了一个强大的工具。空间转录组数据的分析随着技术的发展,变得越来越有挑战性。王涛课题组认为,严谨的数据预处理,是正确分析和理解空间转录组数据的关键,而Sprod降噪正是预处理中重要且有力的一环。
该论文的共一作为王允冠博士与宋冰博士。论文的其他主要作者包括西南医学中心的谢阳教授,肖光华教授,王诗丹助理教授。定量生物学研究中心有多个博士后招聘职位(qbrc.swmed.edu/labs/wanglab,qbrc.swmed.edu/labs/xielab,qbrc.swmed.edu/labs/xiaolab)。我们欢迎各个专业的生物信息学人才加盟我们。 参考资料 Wang, Y., Song, B., Wang, S. et al. Sprod for de-noising spatially resolved transcriptomics data based on position and image information. Nat Methods 19, 950–958 (2022). https://doi.org/10.1038/s41592-022-01560-w
数据
https://www.10xgenomics.com/resources/datasets
代码