来自微软和人大《生物分子和自然语言的多模态学习》综述
生物分子建模与自然语言(BL)的整合已经成为一个前景光明的跨学科领域,位于人工智能、化学和生物学的交汇处。这种方法利用文本数据源中包含的关于生物分子的丰富、多面的描述来增强我们的基本理解,并使下游计算任务成为可能,如生物分子属性预测。将通过自然语言表达的细腻叙述与通过各种分子建模技术描述的生物分子的结构和功能特性的融合,为全面表示和分析生物分子开辟了新途径。通过将围绕生物分子的上下文语言数据纳入其建模中,BL旨在捕捉一个全面视角,包括通过语言传达的象征性质以及量化的结构特性。在这篇综述中,我们提供了通过生物分子和自然语言的交叉建模所取得的最新进展的广泛分析。(1)我们首先概述了采用的生物分子的技术表示,包括序列、二维图和三维结构。(2)然后我们深入探讨了有效多模态整合语言和分子数据源的基本原理和主要目标。这包括探索机器学习框架,如基于GPT的预训练和多流神经网络,以及表示学习的各个方面,如网络架构、训练任务和策略。(3)随后我们调研了到目前为止在这个发展中的研究领域中启用的实际应用,重点是属性预测、分子描述的生成和从文本中检索生物分子数据的用例。(4)我们还汇编和总结了可用的资源和数据集,以促进未来的工作。(5)展望未来,我们确定了几个有前景的研究方向,值得进一步探索和投资,以继续推进该领域的发展。最终,通过这一全面分析,我们旨在为生物学、化学和AI领域的跨学科研究者提供当前状态和BL未来潜力的彻底基础。相关资源和内容在更新中 https://github.com/QizhiPei/Awesome-Biomolecule-Language-Cross-Modeling。
生物研究领域长期以来一直认识到彻底理解生物分子(如分子和蛋白质)在推进药物发现、人类理解以及其他与生物分子相关的应用中的至关重要性。作为化学物质的基本单元,分子在生化反应和细胞功能中发挥重要作用,而蛋白质则因其在结构支持、酶催化、信号传导等多种功能中的关键作用而至关重要。因此,这些实体是现代生物研究的基石。
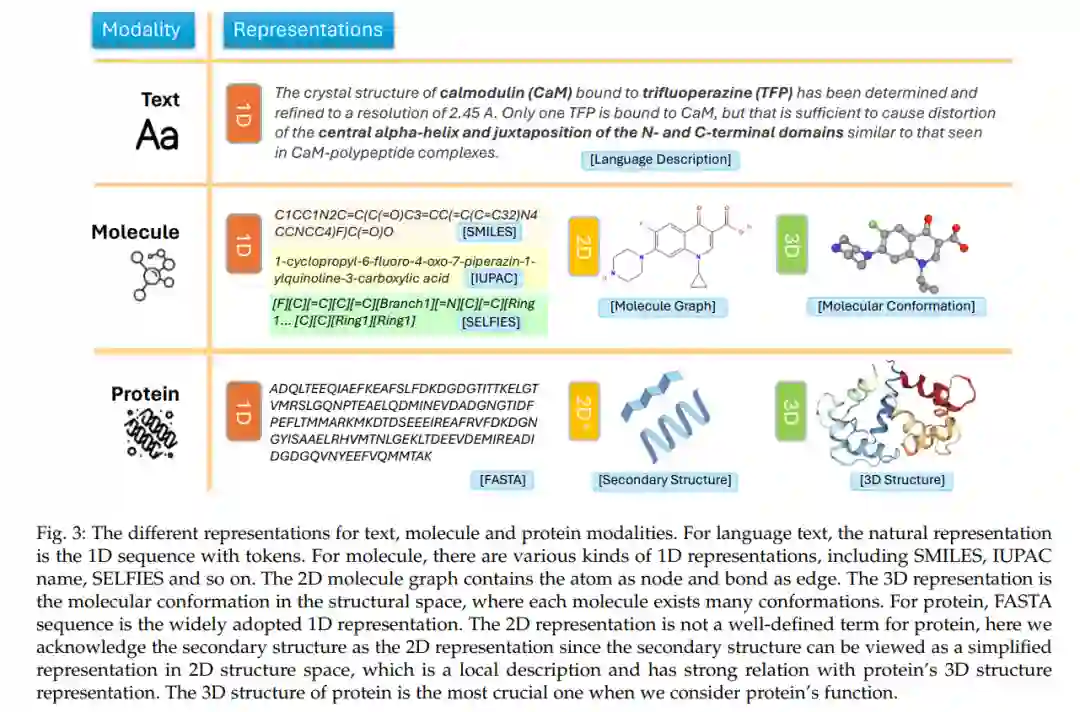
生物分子可以以多种形式表示,以计算方式建模其结构和属性。一种常见的表示是生物序列,将生物分子编码为如核苷酸或氨基酸等单体的线性链。例如,分子可以通过简化的分子输入行条目系统(SMILES)表示,蛋白质通常由FASTA序列表示。如ChemBERTa、ProtTrans和ESM等基于序列的方法在建模这些序列属性方面取得了成功。此外,生物分子可以通过将原子表示为节点,化学键表示为边的2D图来建模。基于图的方法如MolCLR和Graphormer利用图神经网络(GNNs)来学习从图结构到功能映射。除了序列和基于图的编码外,通过实验或预测确定的分子和蛋白质的3D结构/构象也可以作为深度学习技术的输入。例如,AlphaFold、ProteinMPNN和Uni-Mol利用3D坐标数据来建模结构属性。
尽管这些基于深度学习的表示方法对于从不同抽象层次捕获固有的生物分子特征非常有力,但它们通常忽视了如生物医学文献和数据库等丰富的外部知识来源。例如,PubMed包含大量关于生物分子的注释和详细实验发现的出版物。像PubChem和UniProtKB这样的资源同样汇编了已知生物分子的无数属性、功能和相互作用。直觉上,这些外部知识来源提供了生物分子的广泛多面向文本描述,提供了从分子表示中缺失的语言上下文。然而,当前的生物分子建模范式在系统利用这些丰富的语言数据构建更全面模型的能力上受到限制。 在计算机视觉(CV)和自然语言处理(NLP)技术融合的背景下,多模态建模取得了显著进展。如PaLM、BLIP2和LLaVA等模型有效地整合了图像和文本等不同数据类型,以开发对复杂现实世界领域的更丰富理解。在此势头上建立,尤其是像GPT-4、LLaMA和Alpaca这样的大型预训练语言模型(LLMs)的发展,激发了对生物分子和自然语言联合建模的新兴兴趣。
最近在这一领域开发的模型,如MolT5、BioT5和KEDD,将生物分子的文本描述直接纳入其预训练目标。这使得模型能够学习捕捉结构和语言视角下的生物分子的多面向表示。这些先进技术促进的集成建模提供了对生物功能、属性和活动的更深入见解。例如,属性预测、生物医学自然语言处理和分子检索等领域的下游任务已经从这些联合表示中受益。特别是,基于强大的BERT架构的KV-PLM模型擅长学习分子-文本对齐,并通过其集成的生物分子-语言表示在相关任务上展现出了比传统方法更优的性能。
虽然通过像BioT5和KV-PLM这样的方法在联合建模生物分子和自然语言方面取得了显著进展,但目前这个领域缺乏一个全面调查进展和正在开发的各种方法的统一资源。为了填补这一空缺,我们提出了一个关于生物分子和语言的跨模态整合的广泛综述,我们将其称为跨生物分子-语言(BL)建模。
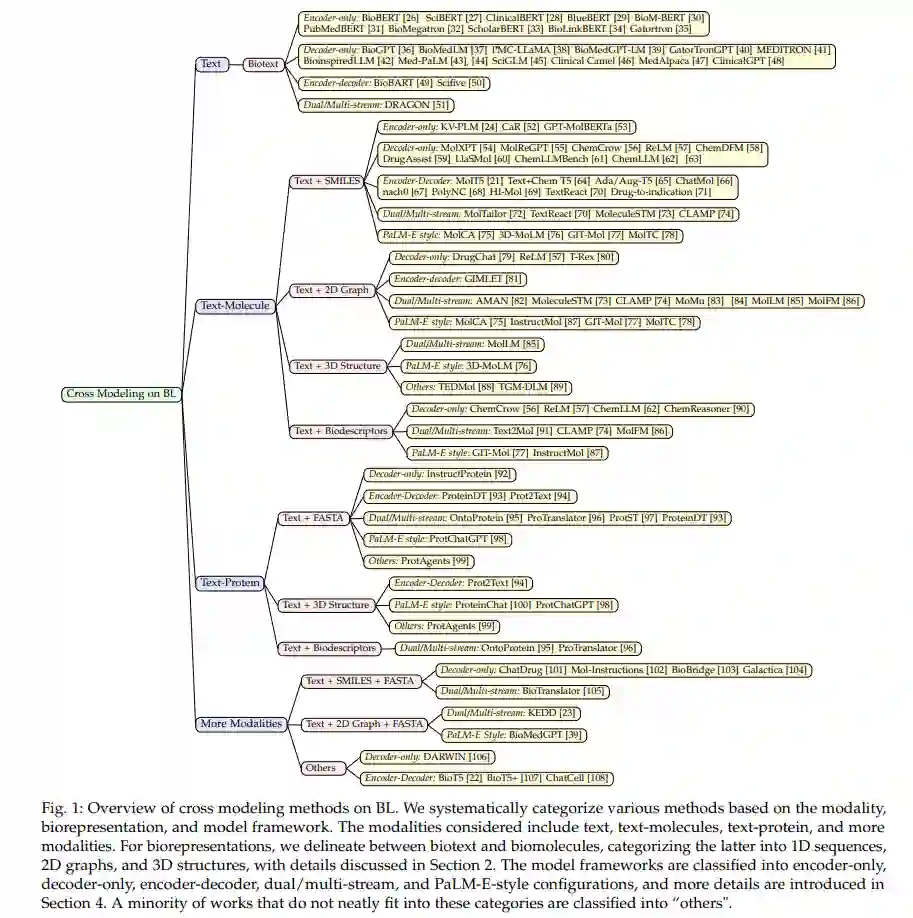
通过这篇综述,我们的目标是为生物学、化学和人工智能交叉领域的跨学科和AI4Science研究人员提供当前技术、挑战和未来方向的深入理解,这是这个迅速发展的研究领域内的内容。我们提供了生物分子表示方法、多模态整合算法、表示学习框架以及从BL中受益的多样化应用领域的详尽分析。我们还讨论了可用的生物分子和语言数据集以促进进一步进展。图1中的分层树状图促进了对BL领域中使用的多样化方法的结构化理解。
此外,我们确定了有前景的研究途径和开放的挑战,这些挑战值得进一步探索。通过整合BL现有作品的见解,我们旨在为科学社区提供一个基础参考。我们希望这篇全面的综述能够帮助指导并催化推动该领域向前发展的新调查,最终通过结构和语言知识的多模态整合支持通过增强的生物分子特征化、发现和理解。 这篇全面的综述如下组织:在第2节中,我们首先深入检查了该领域中使用的常见生物分子表示技术,包括1D序列、2D图和3D结构。第3节则从知识表示和机器学习的角度实证分析了不同数据模态之间的关系,并探索了它们整合的动因。第4节深入研究了所使用的突出机器学习框架,如基于GPT的预训练和多流神经网络架构。第5节进一步详细讨论了表示学习方法论,分析了网络架构设计、训练目标和学习策略等方面。接下来在第6节中,我们调查了到目前为止启用的实际应用,重点是预测建模、生成建模和信息检索等领域。第7节提供了公开可用的数据集、模型和基准结果的汇编。在第8节中,我们确定了开放挑战和未来方向,例如提高模型的可解释性和泛化能力。最后,在第9节中我们总结了我们的发现,并通过概述几个有前景的研究途径来结束。通过对过去的工作、当前技术和未来展望的系统阐述,我们旨在为AI和科学社区提供一个全面的资源。
生物分子表示
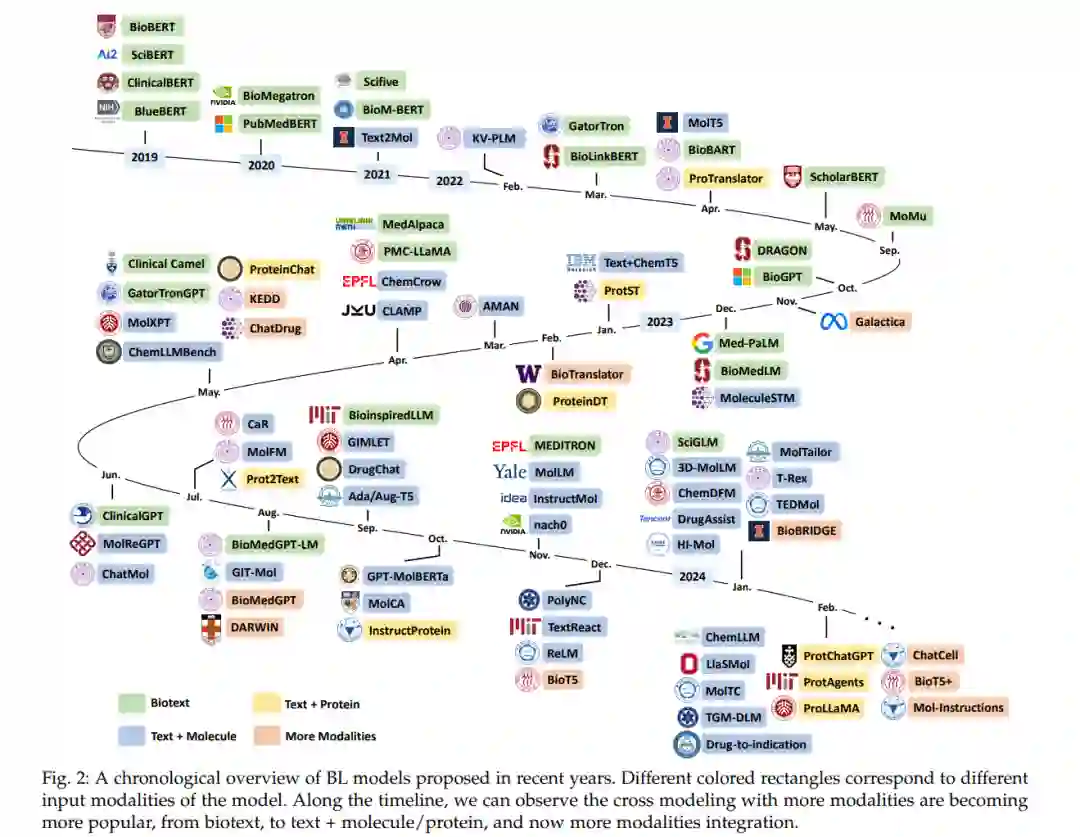
在本节中,我们简洁回顾了生物分子的各种表示及相关建模方法。一般来说,语言文本、分子和蛋白质的模态可以用不同的表示来观察,如1D序列、2D图和3D结构。不同模态及其表示方法的总结在图3中呈现。此外,本节介绍的不同模态的现有BL模型开发的时间顺序概述在图2中展示。这些模型的介绍和更多细节贯穿整个综述。
跨模型
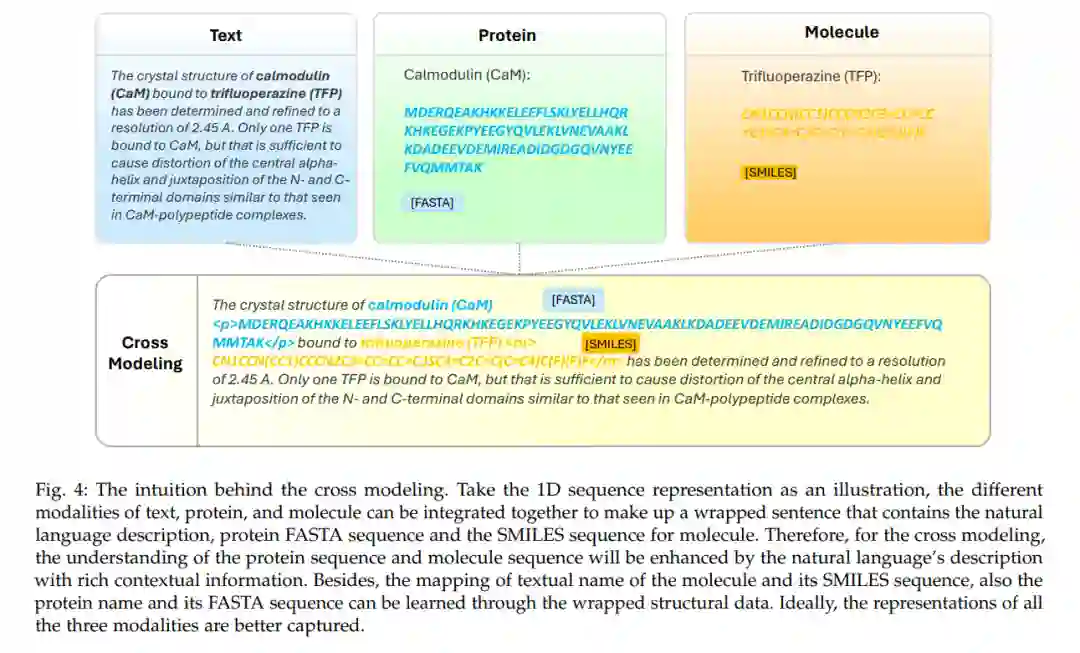
跨模型的直觉 跨模型整合生物分子与自然语言旨在克服传统生物分子表示固有的局限性。尽管现有的表示方法可以有效捕捉生物分子的固有属性,但它们常常忽略了可以进一步丰富理解的外部知识。这种外部知识,可通过生物学文献、数据库和知识图谱等各种格式访问,提供了对生物分子更广泛的生物学背景和功能方面的全面描述和见解。例如,考虑一个分子M,其毒性特征在科学文本中有详细描述。基于这类文本预训练的模型装备有推断类似新分子M'的潜在毒性的能力。此外,语言为生物分子设计提供了一种更灵活的媒介。通过生物分子和语言的集成建模,模型可以利用广泛的生物学知识来执行与生物分子相关的任务,实现对分子生成和编辑的更细致控制。这种整合促进了对广阔分子空间的探索,允许创造具有所需属性的新生物分子。为了更好地说明,我们在图4中展示了这些模态的序列表示如何在跨模型中整合。与单一模态建模不同,分子(SMILES)和蛋白质(FASTA)的序列表示被整合成一个包含自然语言描述的包裹句子,这提供了更丰富的上下文信息,以便更好地理解分子和蛋白质。
跨模型的目标
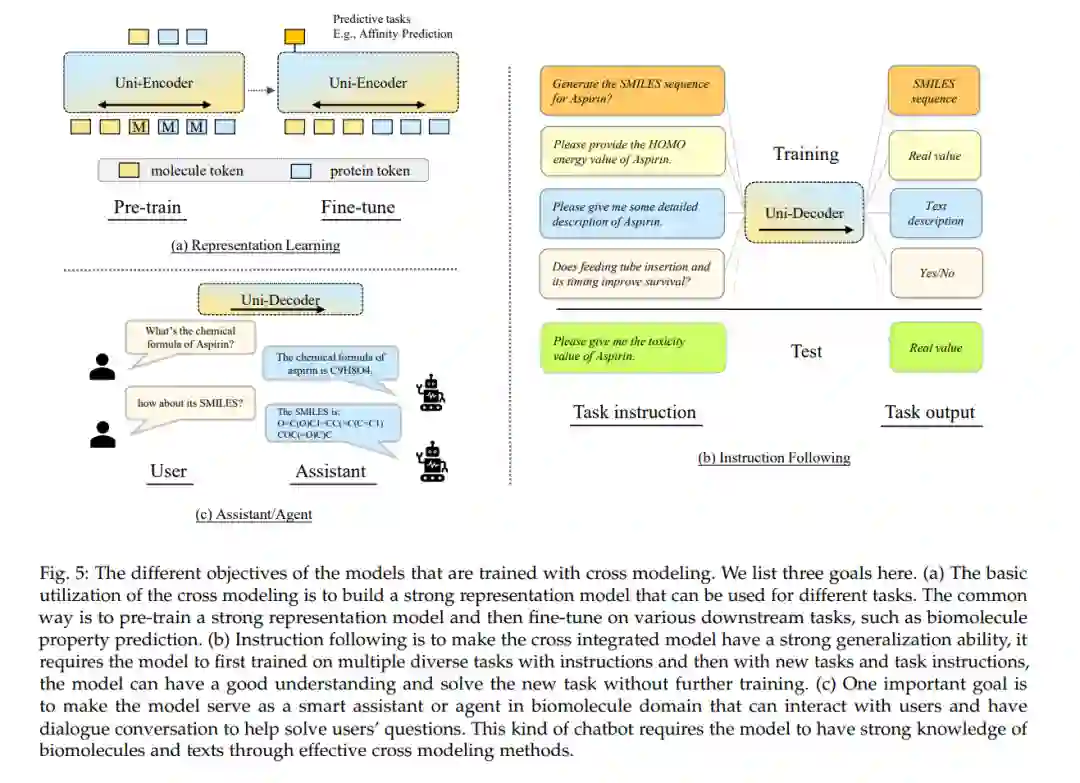
整合生物分子与外部知识源的主要目标是开发能够理解和预测生物分子在生物学上下文中的复杂行为和相互作用的模型。通过结合内在的分子特征和外部文本信息,这些模型可以获得生物分子的更全面表示。我们探索了体现整合目标的三种不同方法:表示学习、指令跟随和代理/助理模型,每种方法都独特地贡献于该领域(见图5以获得视觉理解)。
学习框架
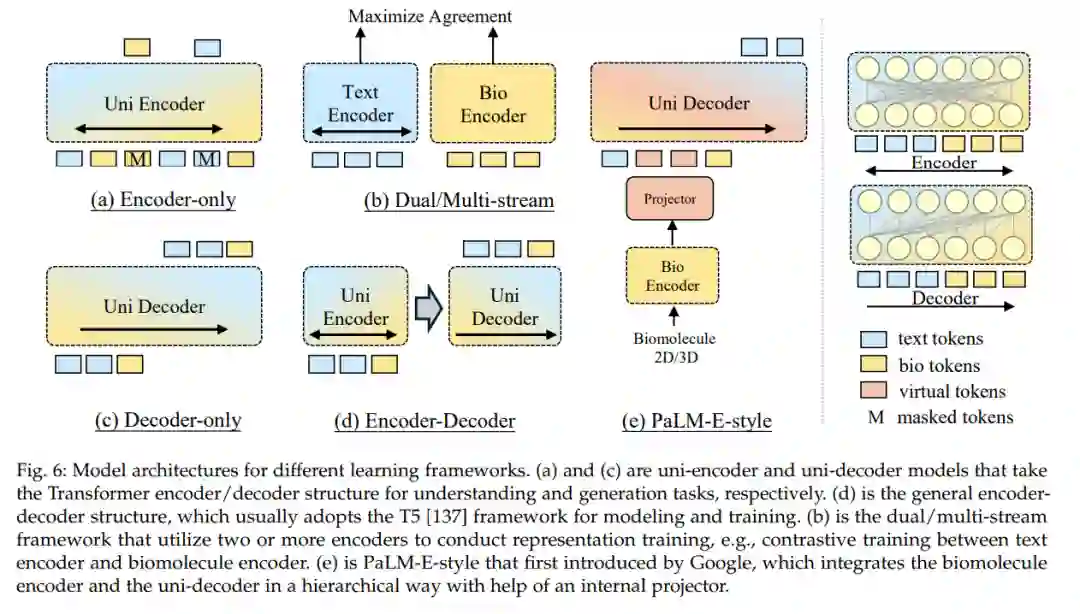
Transformer架构已成为BL领域大多数现代模型框架的基石。在本节中,我们首先介绍用于BL的传统Transformer模型,包括仅编码器/解码器和编码器-解码器架构。此外,我们探讨了用于BL的创新Transformer变体,包括利用基础大型语言模型(LLMs)与外部编码器和模态投影器的PaLM-E风格模型,以及采用多个编码器处理不同模态的双/多流模型。这些架构的概览展示在图6中。后续部分将详细阐述这些模型。
Transformer架构通过其自注意力(Self-Attention)机制有效捕获序列内元素之间的长距离依赖关系,这使得其在处理复杂序列数据,如文本、蛋白质序列或分子结构时表现出色。在BL领域,这一特性尤为重要,因为它允许模型同时考虑生物分子的结构特性和相关的语言描述,提供一种整合不同数据模态的有效方法。 仅编码器或仅解码器的Transformer架构专注于单一任务,如序列分类或生成任务。而编码器-解码器架构则更适用于翻译任务,能够将一个模态的信息转换为另一个模态的表达,例如从自然语言描述生成分子的SMILES表示。
PaLM-E风格模型和双/多流模型进一步扩展了Transformer的应用,通过引入额外的编码器或投影器来增强模型处理多模态数据的能力。这些变体模型通过整合更广泛的生物学知识和数据,旨在提高模型在生物分子预测、生成和解释任务中的性能。
这些不同的Transformer架构为BL领域带来了灵活性和创新,允许研究人员探索从基本的表示学习到复杂的多模态整合和交互任务的广泛应用。随着这些模型在详细解析生物分子的复杂性和多样性方面的不断发展,它们在促进生物学发现和应用中的潜力也在增长。
表示学习
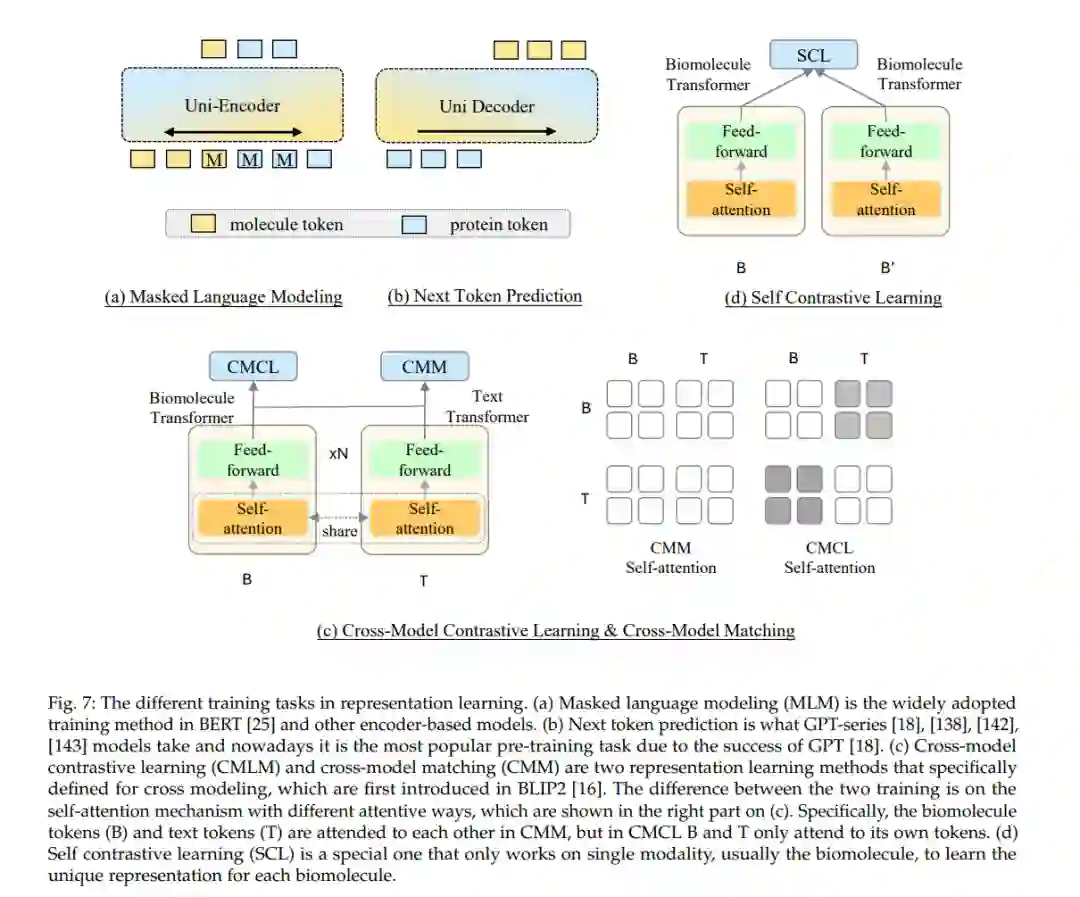
在本节中,我们讨论了使用生物分子和文本数据进行BL预训练中流行的任务和策略。这包括单模态预训练和跨模态预训练。
单模态预训练专注于增强对单一模态的理解,包括分子、蛋白质和文本数据,以提高模型在特定领域内的性能。相比之下,跨模态预训练旨在在这些不同模态之间建立相互联系,促进跨领域的更加整合的理解。在这里,我们详细阐述了在研究社区内获得广泛接受的训练任务和策略。另外,我们在7.1节中列举了各种预训练数据资源。
单模态预训练任务通常涉及如语言模型训练、序列分类或序列到序列的转换,这些任务专注于提升模型在处理特定类型数据时的能力,如理解文本语义、预测蛋白质结构或识别分子属性。
跨模态预训练任务则设计更为复杂的策略,如联合嵌入学习、对齐任务或模态转换任务,这些任务旨在提高模型对不同数据类型之间相互关系的理解,从而增强模型的泛化能力和跨模态推理能力。例如,一个跨模态预训练任务可能要求模型根据文本描述生成相应的分子结构,或者从分子结构中提取出与文本描述匹配的特征。 在表示学习中采用的策略包括自监督学习、对比学习和多任务学习等。自监督学习通过生成任务或填空任务来促使模型捕获数据内在的模式和结构。对比学习则通过比较正样本对和负样本对来学习数据中的区分性特征。多任务学习通过同时训练模型在多个相关任务上表现良好,以提升模型的通用性和鲁棒性。
通过这些表示学习的任务和策略,BL模型能够更有效地捕获生物分子的复杂性和文本数据的丰富性,从而在各种生物学和医学应用中实现更准确的预测和分析。随着预训练数据资源的不断扩展和表示学习方法的持续创新,我们预期BL领域将继续取得显著进展,为生物学研究和药物发现带来新的洞察和机会。
结论 我们提供了对生物分子与自然语言整合这一新兴领域的深入综述。我们从详细介绍各种生物分子表示开始,如图和序列,强调它们在生物学研究中的重要性。然后,我们讨论了整合背后的直觉和目标,强调将生物分子数据与自然语言相结合以增强理解和获得新见解的好处。接着,我们探索了不同的学习框架,如基于GPT的训练和多流建模,并深入到表示学习的复杂性中,涵盖了训练任务和策略等方面。我们还展示了这种集成方法已被证明有益的各种应用。此外,我们编译了关于数据集/基准、模型和代表性基准总结的资源,以促进该领域的研究和开发。尽管取得了显著进展,该领域仍面临着几个挑战,呼吁未来进一步的努力。