Blend the Separated: Mixture of Synergistic Experts for Data-Scarcity Drug-Target Interaction Prediction
**作者:**翟新龙,王春辰,王睿嘉,康佳政,李书杰,陈博宇,马腾飞,周子凯,杨成,石川 **单位:**北京邮电大学,中国电信云计算研究院,湖南大学 **摘要:**药物-靶点交互(Drug-Target Interaction, DTI)预测在药物发现和临床应用等各种场景中至关重要。DTI 预测中广泛使用的输入数据有两种视角:内在数据代表药物或靶标的结构,外在数据代表药物或靶标与其他生物实体的关系。**然而,对于药物或靶点,输入数据的两个角度中的任何一个都可能是稀疏的,特别是对于那些不流行或新发现的药物或靶点。此外,特定交互类型的真实标签也可能很少。因此,我们提出了一个方法来解决输入数据和/或标签稀缺情况下的 DTI 预测。**为了使我们的模型在只有一种输入数据可用时发挥作用,我们设计了两个独立的专家来分别处理内在和外在数据,并根据不同的样本自适应地融合它们。此外,为了使两种角度相互补充并弥补标签稀缺性,两个专家以相互监督的方式相互协同,以利用大量的未标签数据,并从数学上证明了我们的协同机制的有效性。在不同程度的输入数据稀缺和/或标签稀缺情况下对 3 个真实世界数据集进行的广泛实验表明,我们的模型显著且稳定地优于现有技术,最大改进为 53.53%。我们还在没有任何数据稀缺的情况下测试了我们的模型,它也优于当前的方法。
1. 背景与动机
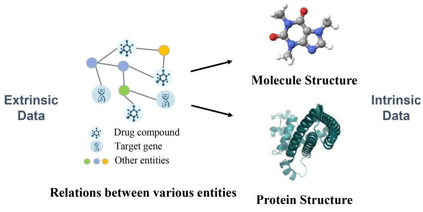
药物-靶点交互(DTI)预测在各个生物领域至关重要,尤其是在药理学领域中。在这个任务中,我们输入一个药物(分子)和一个靶点(基因或基因编码的蛋白质),输出它们交互的概率。神经网络方法在 DTI 预测方面的发展迅速,显著降低了领域知识的需要,并展示了优异的结果。通常,这些方法可以利用两种数据视角,如图 1 所示。数据的第一种视角是分子或蛋白质是如何组成的,比如分子的结构和氨基酸残基的序列。我们将这种数据视角称为内在数据。数据的第二个视角是除了药物和靶点之外,各种生物实体之间的关系,例如疾病、药物、基因、症状、副作用之间的相互关系。我们将这种数据视角称为外在数据。实体和它们之间的关系可以构成图,因此可以应用各种图嵌入方法。考虑利用两种数据视角来实现更好的预测性能也是很自然的,目前也有一些此类方法。 然而,有两种形式的数据稀缺限制了所有此类方法的使用:(1)内在或外在输入数据稀缺。例如,就内在数据而言,获得最准确和精密的蛋白质结构仍然依赖于使用冷冻电子显微镜等昂贵设备的湿实验,导致精确蛋白质结构的稀缺。对于外在数据,尽管生物实体之间积累了大量的关系数据,但新发现或不流行的药物或靶点仍然可能与其他实体具有非常少的联系。(2) 交互标签稀缺性。药物和靶点之间的交互有多种特定类型。虽然有丰富的表示药物和靶点之间是否有交互的数据,但是特定交互类型的标签仍然可能不甚丰富。
图1:内在和外在数据图示。 这项工作的主要研究目标是提出一种方法,该方法可以有效地利用内在和外在数据,同时在输入数据和/或交互标签稀缺性下仍然有效。这就要求我们解决以下两个挑战:(1) 如何灵活、实质性地融合内在和外在数据。直接融合内部数据和外部数据的模型(例如直接拼接两个视角数据的嵌入)无法在缺失一种视角的数据时进行预测。此外,在没有单一数据视角的情况下进行预测时,我们是否仍然能够利用训练期间从该视角的数据中学到的知识?(2) 如何使用有限的交互标签进行有效的模型训练。内在数据包含药物和靶点本身的成分信息,而外在数据包含药物和靶点之间的更高层次的语义信息。因此,如何通过利用两个数据视角之间的互补性来更有效地优化模型还有待探索。 在本文中,我们提出了一种新的方法 MoseDTI,即用于数据稀缺的药物-靶点交互预测的协同混合专家,该方法在这两种类型的数据稀缺中的任何一种或两种情况下均表现良好。我们提出了一种新颖的模型架构,称为协同混合专家,以统一和有机地应对这两个挑战。我们设计了两个异构专家,分别根据内在和外在数据预测 DTI 交互的概率。然后,使用门控模型,根据样本的内在数据还是外在数据更可靠,自适应地使用二者的输出。并且,这两位专家之间存在协同,即一位专家在训练期间监督另一位专家,将一个角度的知识注入另一个专家。如果在预测时没有内在或外在数据,其中一位专家仍然可以正常预测。具体来说,这两位专家设计为彼此生成伪标签作为监督方法。生成的伪标签有效地扩大了两位专家和门控模型的训练样本,并充分利用了两个数据视角之间的互补性。
2. 符号与预备知识
2.1 外在数据
我们将外在数据视为一个知识图谱(KG),表示为 ,其中包含不同类型生物实体之间的丰富关系信息。 是实体集合, 是关系集合, 是观测到的三元组集合 。在一个三元组中, 分别表示头实体、关系和尾实体。实体集合 包含各种生物实体,例如疾病、副作用和症状;药物和靶点集合是实体集合的子集:。为了防止标签数据泄露,我们从 中移除所有药物和靶点之间的直接连接,即移除所有满足 或 的 。
2.2 内在数据
对于一个药物 ,我们使用其 SMILES 序列 作为内在特征。SMILES,即简化分子输入线性表示系统,是一种以线性符号表示化学物质结构的规范,使用短的 ASCII 字符串描述化学物质的结构。对于一个靶基因 ,我们通过 UniProt 数据库获取其编码蛋白质的氨基酸序列,作为其内在特征,记为 。
2.3 DTI预测任务
在药物-靶点交互预测任务中,我们的目标是估计药物-靶点对 在特定交互类型下的交互概率 ,其中 。这样的 DTI 数据集可以描述为 ,其中 或 分别表示 ,用于指示这些药物-靶点对是否具有该类型的相互作用。 和 分别表示所有药物和靶点的内在数据。
3. 方法
3.1 模型设计
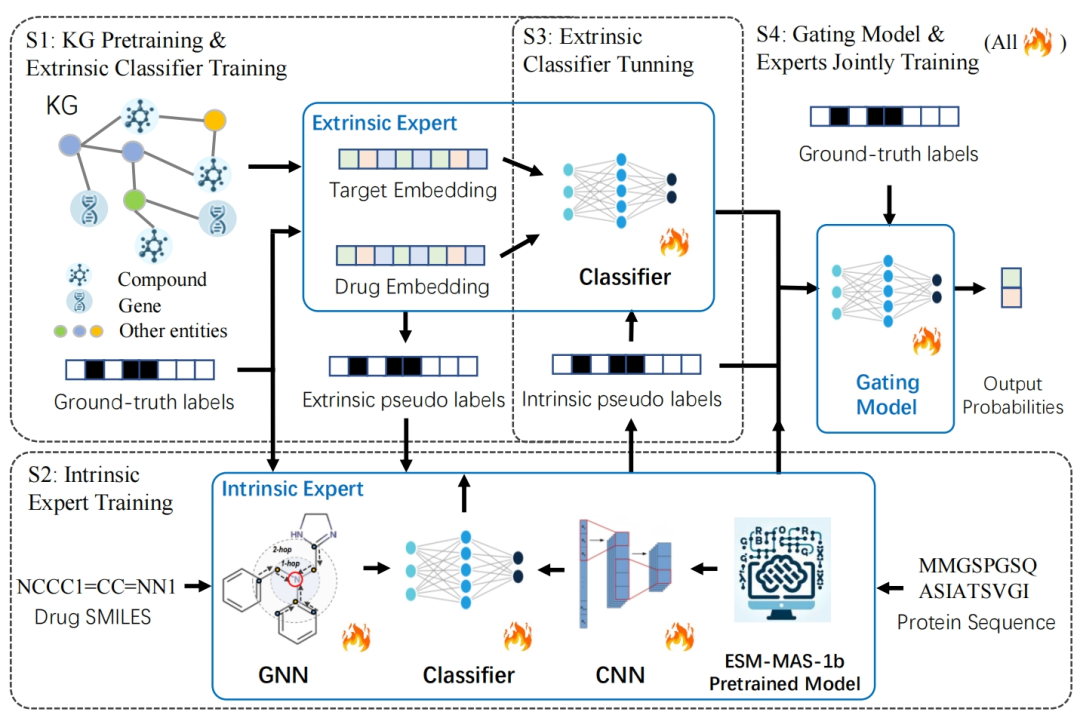
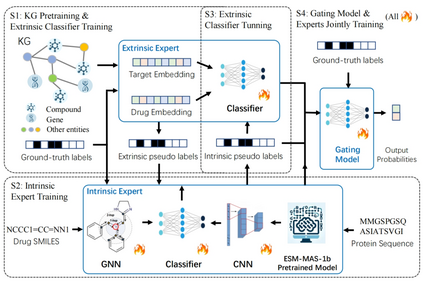
我们提出了一种新模型框架MoseDTI,用于在数据稀缺条件下的药物-靶点交互预测。MoseDTI 的模型主体是一个融合内在数据(如分子结构、蛋白质序列)与外在数据(如生物实体间关系)的混合专家架构,能够根据样本数据的特性自适应地调整专家输出。这里只简要介绍大致框架,见图2,更多模型细节详见论文。我们的工作主要在于提出一种模型的框架,而框架中各具体的encoder都可以使用更先进的encoder作为替代。
图2:我们的 MoseDTI 框架。 模型的三个部分用蓝色矩形包围,前三个训练步骤 S1 到 S3 用黑色虚线矩形包围。对于最后一步训练S4,所有带小火苗的组件都进行联合训练。 模型由以下三部分组成:
- 外在专家:利用知识图谱中的关系数据预测相互作用概率。通过预训练生成药物和靶点的外在嵌入,再通过一个简单的分类器输出预测值:
- 内在专家:基于药物的 SMILES 序列和靶点的氨基酸序列进行预测。药物编码器通过图神经网络(GNN)处理分子图,靶点编码器通过预训练的蛋白质语言模型提取序列特征,分类器输出预测值:
- 门控模型:对两个专家的输出进行加权融合,根据样本的特性决定哪个专家更可靠:最终输出为:
3.2 优化过程
为了充分利用未标注数据,模型引入了一个专家协同的机制,其中两个专家相互生成伪标签以扩大训练样本:
- 伪标签生成:一个专家在候选样本集上预测高置信度的正负样本,并将其用作另一个专家的监督信号。例如:对应的损失函数为:
- 训练步骤:
- S1:预训练知识图谱嵌入,训练外在专家分类器。
- S2:利用外在专家生成伪标签,训练内在专家。
- S3:利用内在专家生成伪标签,微调外在专家。
- S4:联合训练门控模型与两个专家,进一步融合伪标签和真实标签。
4. 实验
4.1 实验设置
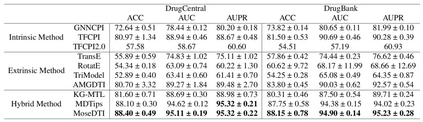
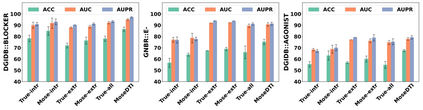
评估方法: 使用 ACC(准确率)、AUC(ROC曲线下面积)和 AUPR(精确召回曲线下面积)作为性能评价指标。在数据稀缺实验中(RQ1),对每个数据集构建了 5 个不同的交叉验证变种,报告平均值和标准差。对于其他实验,基于不同的随机种子重复 5 次实验,报告平均值和标准差。 数据集: 输入数据分为两部分:一部分是内在数据(分子SMILES序列和氨基酸序列)和外在数据(知识图谱DRKG)。另一部分是标签数据。此部分的数据集分为两类。第一类是特定交互的数据集,有DGIDB::BLOCKER(拮抗剂),包含 253 个正样本;GNBR::E-(降低表达),包含 1401 个正样本;DGIDB::AGONIST(激动剂),包含 1338 个正样本。第二类是宽泛的交互数据集,即其中的交互不区分特定类别,有DrugBank 和 DrugCentral,分别包含 18480 和 18066 个样本。
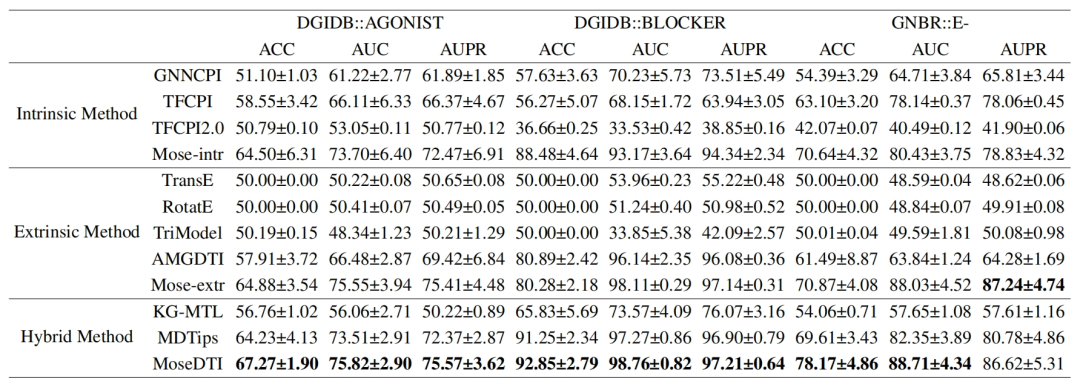
4.2 主实验:验证模型在数据稀缺条件下的表现 (RQ1)
目的:评估 MoseDTI 在内在数据或外在数据稀缺,或交互标签稀缺情况下的预测性能。 方法:设计了 9 种稀缺场景组合,包括:
- 仅有内在数据、仅有外在数据以及两者都存在的情况。
- 每种情况下训练样本的稀缺程度分别为 10-shot、20-shot 和 40-shot。
结果:MoseDTI在所有稀缺设置下均显著优于现有方法,表明其能够有效应对数据稀缺问题。
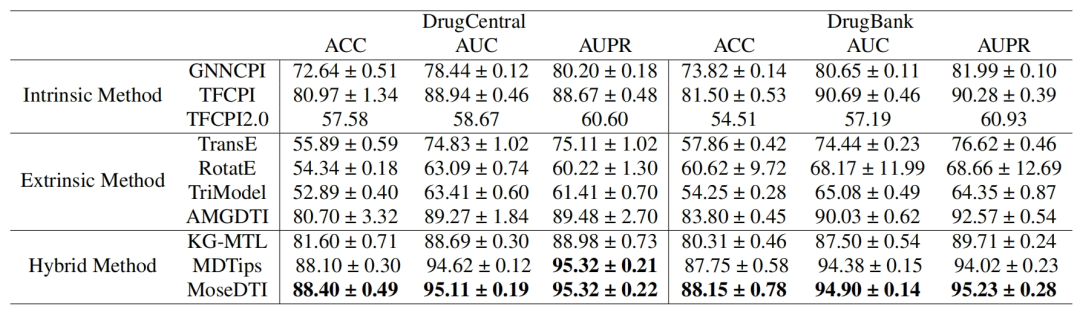
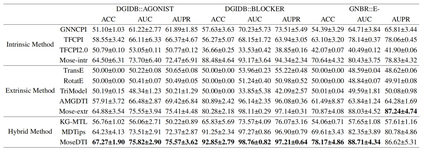
4.3 验证模型在无数据稀缺条件下的表现 (RQ2)
目的:测试 MoseDTI 在没有数据稀缺限制的情况下,是否仍然具有竞争力。 方法:在两个宽泛的DTI交互数据集(DrugBank 和 DrugCentral)上,与多种内在方法、外在方法和混合方法进行对比。 结果:MoseDTI 在两个数据集上的性能均超过其他方法,证明其对数据的普适性。
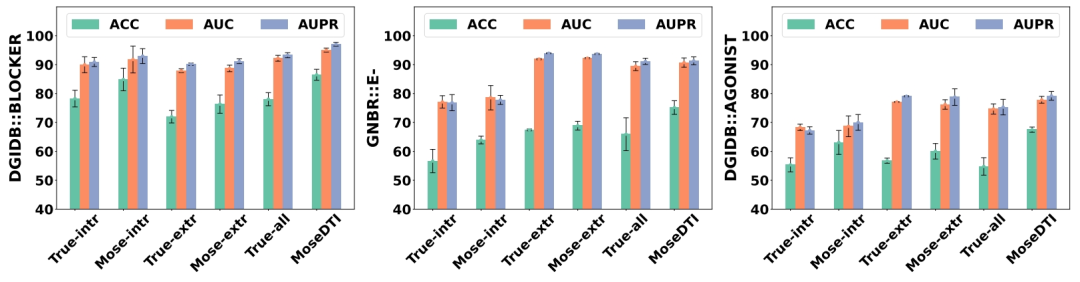
4.4 模型组件的消融实验 (RQ3)
目的:分析模型中专家协同机制和专家混合架构对整体性能的影响。 方法:对比以下模型变种:仅用真实标签训练的内在专家(True-intr)、外在专家(True-extr),以及整个模型(True-all)。比较通过协同机制(Mose-intr 和 Mose-extr)或专家混合架构(MoseDTI)的模型是否提升了性能。 结果:专家协同机制显著提升了单一专家的预测性能。专家混合架构的设计进一步增强了整体模型的表现,验证了 MoseDTI 的有效性。
论文链接: http://shichuan.org/doc/187.pdf 代码链接: https://github.com/BUPT-GAMMA/MoseDTI