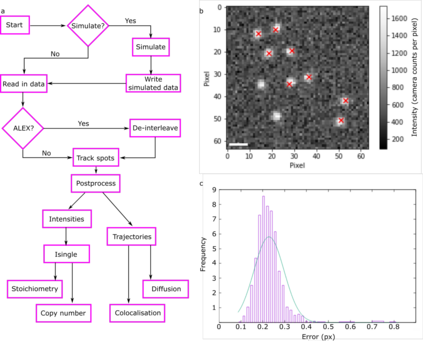
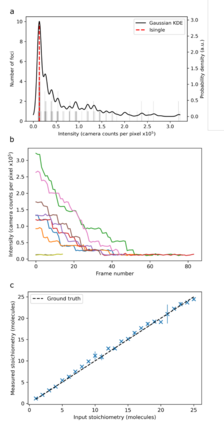
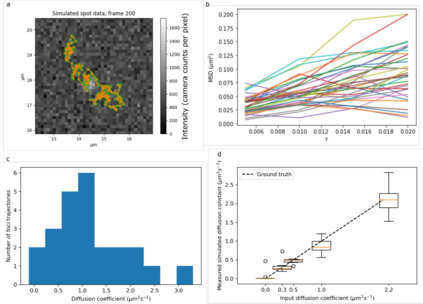
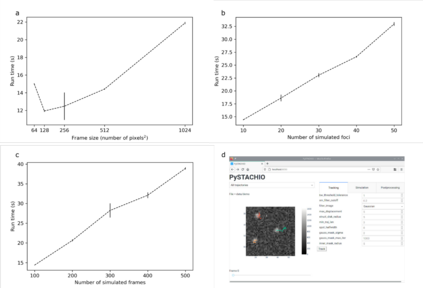
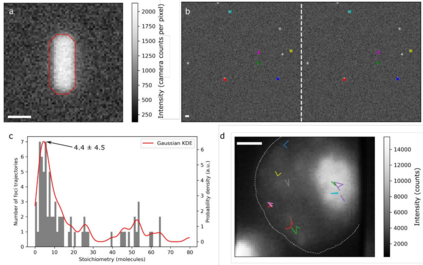
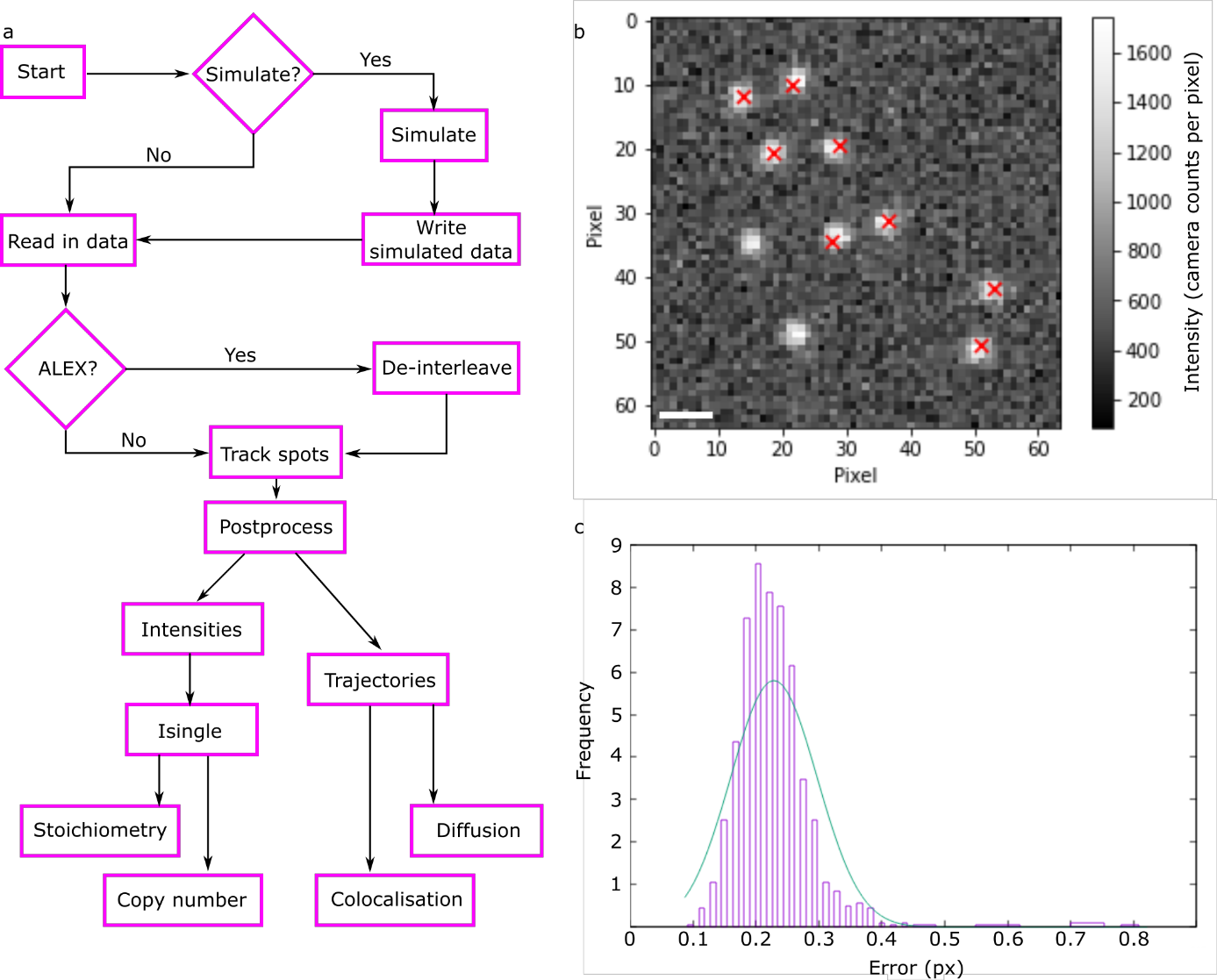
As camera pixel sensors grow larger and faster and optical microscopy techniques become ever more refined, there has been explosions in the quantity of data acquired during routine light microscopy. At the single-molecule level, this analysis involves multiple steps and can quickly become computationally expensive and intractable on ordinary office workstations. Moreover, complex bespoke software can present high activation barriers for new users. Here, we present our recent efforts to redevelop our quantitative single-molecule analysis routines into an optimized and extensible Python program, with both GUI and command-line implementations to facilitate its use on both local machines and remote clusters, and by beginners and advanced users alike. We demonstrate the performance of this code matches our previous MATLAB implementation but at a fraction of the computational cost. We show the code can extract fluorescence intensity values corresponding to single reporter dye molecules and, using these, to estimate molecular stoichiometries and single cell copy numbers of fluorescently labeled biomolecules. It can also evaluate diffusion coefficients for the relatively short single-particle tracking data that is characteristic of time-resolved image stacks. To facilitate benchmarking against other codes, we also include data simulation routines which may trivially be used to compare different analysis programs. Finally, we show that PySTACHIO works also with two-color data and can perform colocalization analysis based on overlap integrals, to infer interactions between differently labelled biomolecules. We hope that by making this freely available for use and modification we can make complex single-molecule analysis of light microscopy data more accessible.
翻译:随着照相像素传感器的扩大和速度的加快以及光学显微镜技术的日益完善,在常规光显微镜检查期间获得的数据数量发生了爆炸。 在单分子一级,这种分析涉及多个步骤,可以很快在普通办公工作站上迅速成为计算费用昂贵和棘手的。 此外,复杂的显微软件可以给新用户带来较高的激活障碍。在这里,我们展示了我们最近为将定量单分子分析程序重新发展成一个最优化和可扩展的Python程序所作的努力,同时使用图形界面和命令线执行,以便利在本地机器和远程集以及生物初生者和高级用户中使用这些数据。我们展示了这一代码的性能与我们以前的 MATLAB 实施过程相匹配,但只是计算成本的一部分。我们展示了该代码可以提取与单报告器染色分子的分子密质分子的强度值值值,并利用这些来估算分子缩略微镜像复制的复制数量。我们也可以通过直白生物细胞来评估在相对短暂的单分子和远程集群组中使用的显微数字分析中进行扩散系数的参数。我们也可以评估用于相对简短的简单的单层分析。我们用来分析,我们用来分析的常规分析。