【ICML2022】蛋白质适应度预测与自回归transformers和推理时间检索
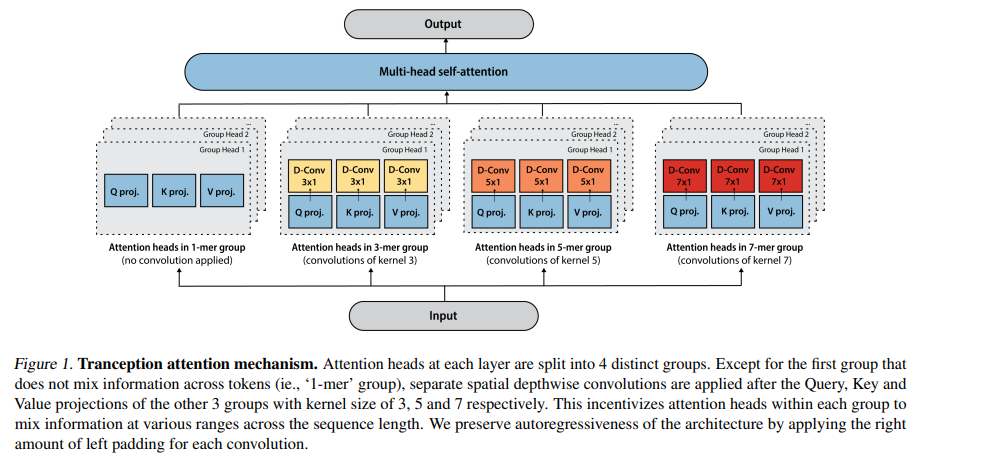
从量化人类变异对疾病可能性的影响,到预测病毒的免疫逃逸突变,以及设计新的生物治疗蛋白,精确模拟蛋白质序列适应度的能力对广泛的应用至关重要。迄今为止,基于多种序列比对训练的蛋白质序列深度生成模型是解决这些任务的最成功的方法。然而,这些方法的性能取决于是否有足够深入和多样化的可靠训练。因此,由于许多蛋白质家族很难(如果不是不可能的话)对齐,它们的潜在范围受到了限制。基于来自不同家族的大量非对齐蛋白序列的大型语言模型解决了这些问题,并显示出最终弥合性能差距的潜力。我们引入了Tranception,一种新颖的transformer 架构,利用自回归预测和同源序列的检索来实现最先进的适应度预测性能。鉴于其在多个突变体上的显著更高的性能,对浅对齐的鲁棒性和评分索引的能力,我们的方法提供了比现有方法的显著增益范围。为了能够在更广泛的蛋白质家族中进行更严格的模型测试,我们开发了ProteinGym——一套广泛的变异效应复合测定法,与现有基准相比,大大增加了测定法的数量和多样性。
专知便捷查看
便捷下载,请关注专知公众号(点击上方蓝色专知关注)
后台回复“TRIT” 就可以获取《【ICML2022】蛋白质适应度预测与自回归transformers和推理时间检索》专知下载链接


登录查看更多
相关内容
Arxiv
14+阅读 · 2021年2月14日