编辑:好困 拉燕
【新智元导读】一个模型即可破译非编码DNA的进化历史和未来?
这次,来自麻省理工学院和英属哥伦比亚大学等机构的研究人员构建了一个深度学习神经网络模型——「神谕」。
利用数亿次实验观测结果进行训练之后,「神谕」可以预测酵母中的非编码DNA序列的突变会如何影响基因表达。
论文链接:
https://www.nature.com/articles/s41586-022-04506-6#Abs1
此外,研究人员还提出了一种独特的方法,可以在两个维度表示适应度地形,从而让理解酵母之外的生物体更加轻松。甚至还能设计出一种通用的基因表达模式,用于推进基因治疗和工业化应用。
虽然我们每个人体细胞都包含大量基因,但是所谓的「编码DNA」仅仅占我们所有基因的1%。而剩下的99%,都不是具备编码能力的DNA,不能通过这些DNA生成蛋白质。
这种非编码DNA(戏称垃圾DNA),有一个重要功能。即,控制基因的「开或关」,以及生成的蛋白质的数量。
随着时间的推移,细胞会复制DNA以生长和分裂。在这些非编码区,突变时常会发生,包括功能上的微调,或是改变控制基因表达的方式。
很多突变都是不值一提的,甚至还有一些突变是有好处的。然而,这些突变偶尔也会增加一些常见疾病(比如2型糖尿病)的患病几率,或者更严重的一些疾病(比如癌症)的患病几率。
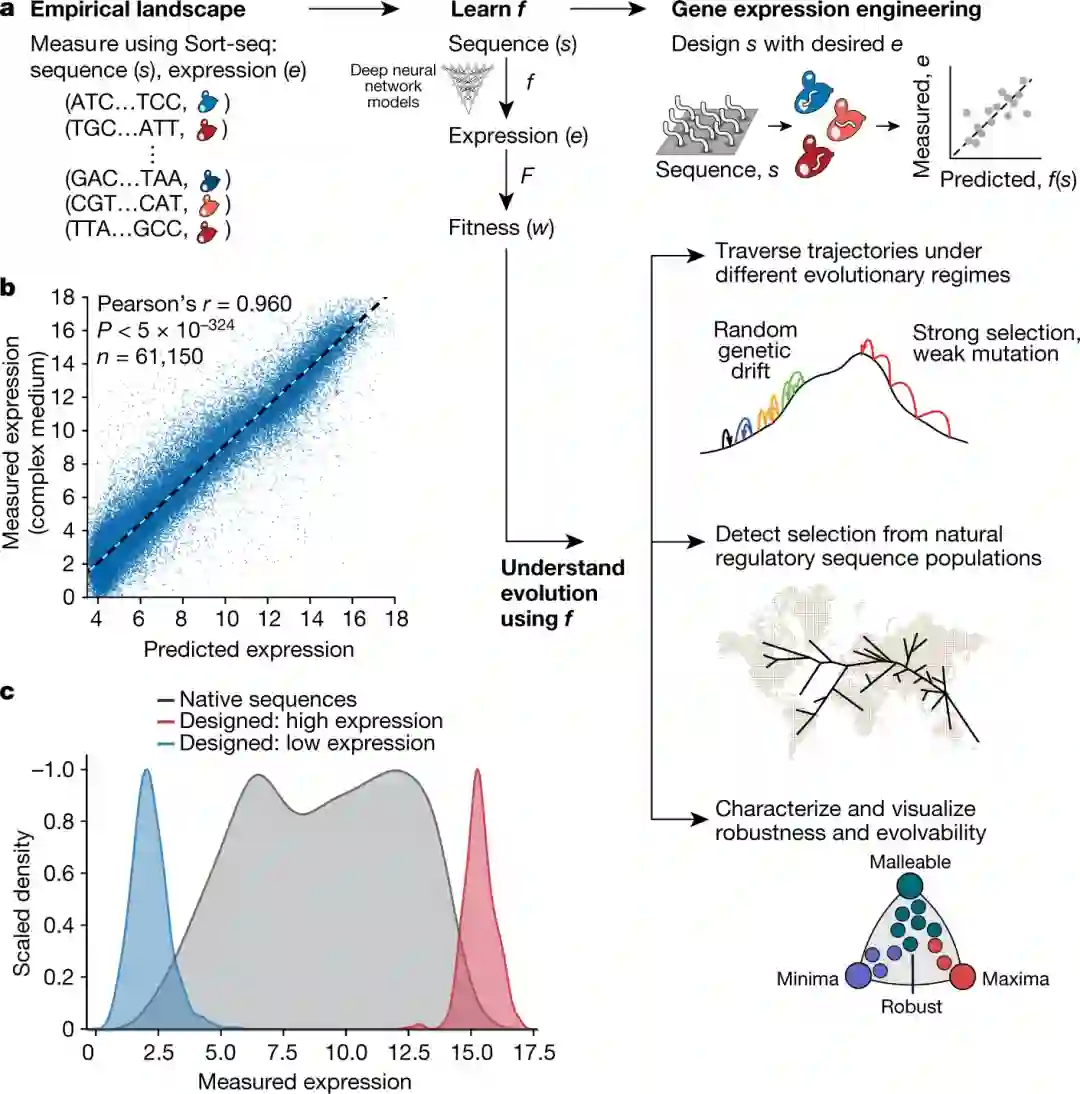
为更好的了解这种突变带来的影响,研究人员一直在进行对数学图谱的研究,从而观察生物体的基因组,预测哪些基因会被表达,并确定该种表达会如何影响生物体可观察的特征。
这些图谱被称作「适应度地形」,大约一个世纪以前,「适应度地形」被提出,目的是理解基因组成如何影响生物体的适应型,尤其是繁殖成功率。早期的图谱比较简单,只关注少量的突变。
如今,研究人员拥有更为丰富的数据库,但他们仍然需要额外的工具来描述这些复杂的数据,并实现数据的可视化。
这种能力一方面可以让研究人员更好地理解一个单独的基因是如何随着时间的推移进化,另一方面还可以帮助预测未来可能出现的基因序列和基因表达的变化。
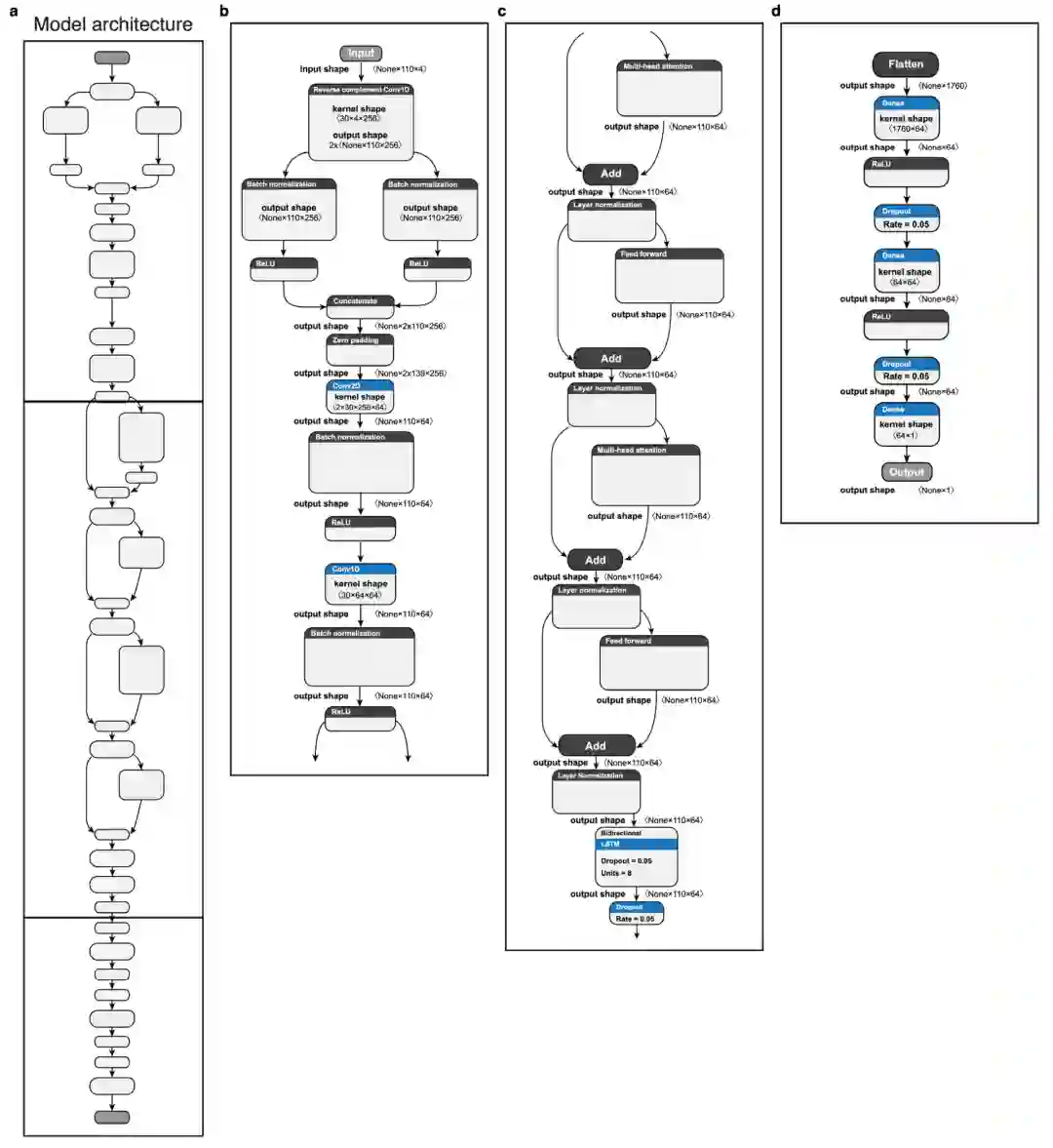
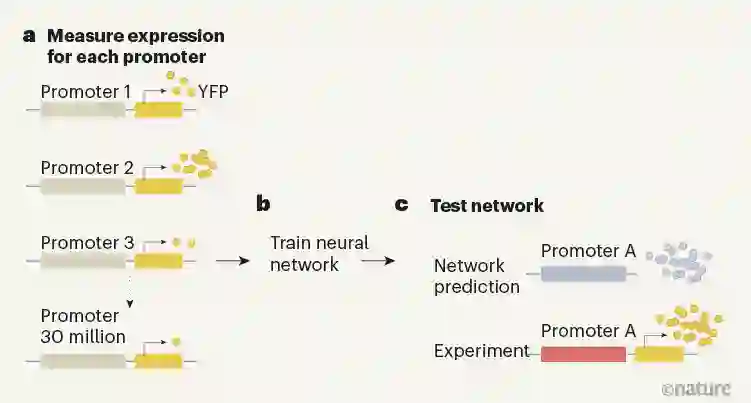
麻省理工学院的研究生Eeshit Dhaval Vaishnav、共同一作Carl de Boer,还有他们的同事们,为了实现这一目标,构建了一个神经网络模型来预测基因表达。
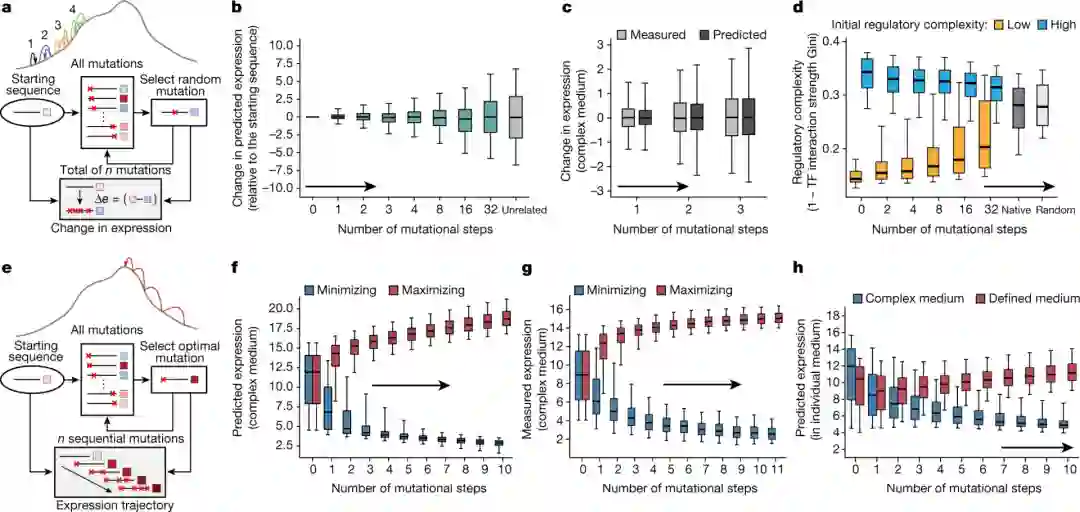
他们通过在酵母中输入上百万个完全随机的非编码DNA序列组成的数据集训练模型,来观察每一个随机序列是如何影响基因表达的。
首先,研究人员在一大群酵母细胞中测量了编码黄色荧光蛋白(YFP)基因的表达情况。
其中,不同的细胞会携带不同的启动子。这些启动子位于一小块环状DNA上靠近YFP基因的地方,作为蛋白质的结合位点,启动子可以控制附近基因的表达。
具体来说,研究人员使用了3000多万个不同的启动子,每个启动子的长度是80个碱基对,并对每个含有这些启动子之一的细胞产生的YFP进行量化。
随后,研究人员将得到的表达数据输入到卷积神经网络之中,并训练该网络从数据中预测基因表达。
为了验证其有效性,研究人员合成了数千个未用于训练的启动子序列,并测量了它们驱动基因表达的能力。
结果表明,神经网络非常准确地预测了每个启动子序列驱动基因表达的程度。
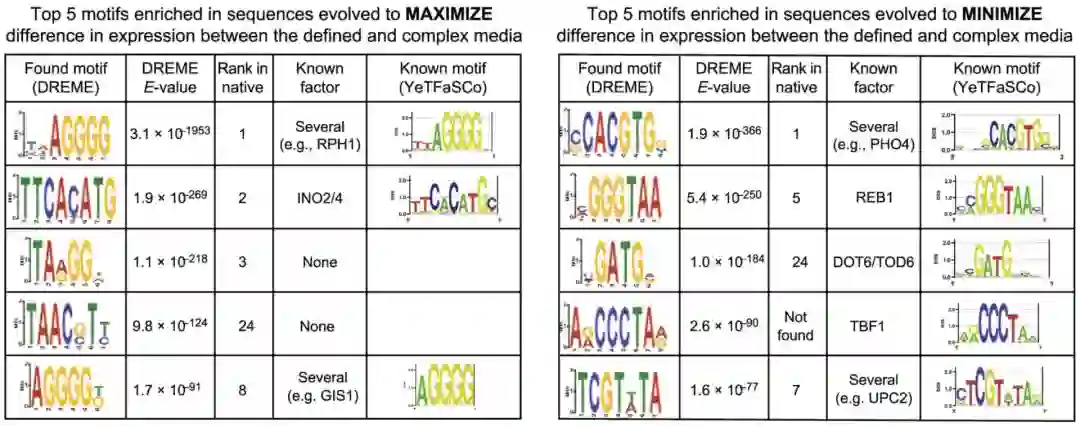
此外,研究人员还向该网络提供了随机的起始序列,结果同样证明了,AI从序列中预测基因表达的能力可以用于将这些起始序列转化为极端YFP表达的启动子序列。
最后,研究人员又合成了500个这些序列,并测量了它们驱动YFP表达的能力。结果表明计算机模拟的序列确实可以驱动非常高和非常低的表达。
为了搞清楚最基础的进化问题,Vaishnav和他的同事们查阅了各类论文,甚至还把一个现有的研究中所有的数据集全放到了模型里进行尝试。
而想构建一个强大到可以探测任何基因的工具,还需要找到一种办法来预测非编码序列的进化模式,哪怕没有完整的数据集。
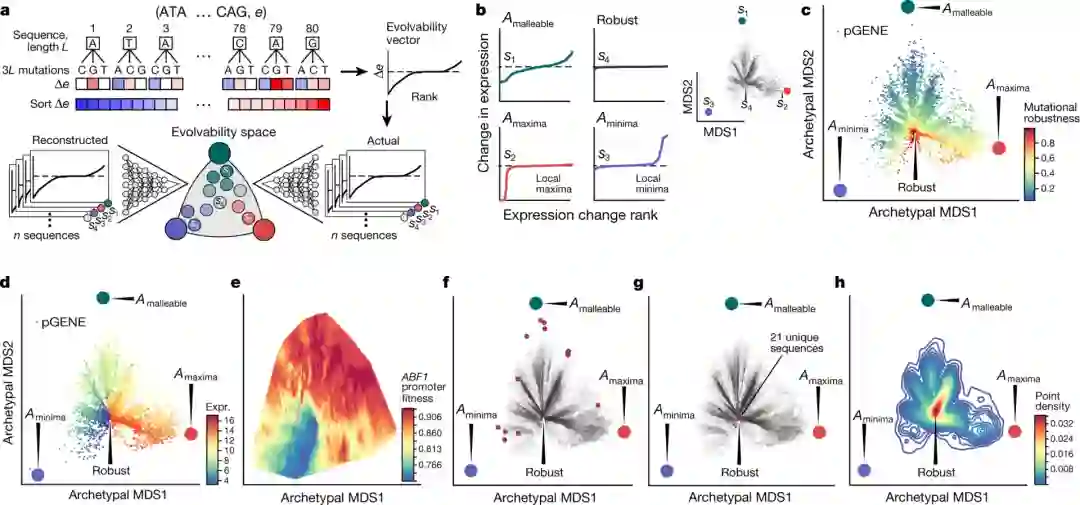
为了实现这个目标,他们设计出了一种计算技巧,可以把预测从框架里插到二维图像上。
如此一来便可以使用简单的方式,了解任何一个非编码的DNA蓄力了是如何影响基因表达和基因的适应性的,且无需在实验室耗时耗力的做任何实验。
50多年来,生物学家们都在试图通过非编码DNA序列来准确预测基因表达的强度。然而基因表达的生化机制是非常复杂的,即便是学界尽了最大的努力也没有实现这一目标。
在这项研究发表以前,研究人员大多只能使用已知的突变来训练模型(充其量有些微小的变化)。
然而,Regev的小组迈出了更大的一步。他们构建的无偏模型,能够预测生物体的适应性和基因表达,这基于任何可能的DNA序列,哪怕有些基因序列从来没有见到过。
实验证明,对于大多数起始序列,3、4个突变足以使序列演化出非常高或非常低的表达。而大约70%的酵母基因在其表达上为稳定选择(有利于不会导致表达发生巨大变化的突变的选择)。
此外,受稳定选择影响的基因对非编码DNA突变的抵抗力更强。也就是说,其启动子的突变在较小程度上改变了基因的表达。
「神谕」的出现和其他诸如预测蛋白质折叠的深度学习应用一样,为科学家们探索和解释更加广泛的领域来带了一种新的方法。
此外,「神谕」也能让研究人员出于制药目的控制细胞,这包括最新的治疗癌症和自身免疫失调的疾病。
麻省理工学院的生物学博士,同时也是哈佛大学和麻省理工学院博德研究所的核心成员的Aviv Regev说:「现在,我们有一个『神谕』,我们可以向它请教很多问题,比如,如果我们把序列里所有的突变全部尝试一遍会怎样、或是我们应该设计出什么样的新序列才能得到我们想要的基因表达。」
她表示,科学家们现在可以使用模型来解决各自的生物进化课题,和为了预期的基因表达设计基因序列的相关问题等等。
爱丁堡大学医学研究委员会人类遗传学部门的教授Martin Taylor表示,该研究充分说明了,人工智能不仅可以预测非编码DNA的变化,还能揭示数百万年生物进化的底层逻辑。
尽管如此,在苏黎世大学从事进化生物学和环境研究的Andreas Wagner表示,「神谕」也有其明显的局限性。
其一,研究人员只改变了启动子--只是可能影响基因表达的几种类型的序列中的一种。它没有考虑到周围DNA变化的影响,包括可能影响基因表达的蛋白质编码区的变化。
其二,它是为酵母而开发的,在酵母中,基因调控的复杂性远低于人类。例如,酵母的调控DNA通常位于被调控基因的几百个碱基对内,而动物的调控DNA可能位于数百万个碱基对之外。因此,目前还不清楚这个方法是否能扩展到更复杂的基因调控。
最后,就像神话中的神谕一样,这个模型可以进行预测但无法解释。
它没有告诉我们为什么一个启动子有高表达或低表达,哪些转录因子在启动子上结合,或者它们如何相互作用。
换句话说,它在阐明基因表达的调控逻辑方面作用不是很大。
尽管用于训练的3000万个序列只是DNA的4个核苷酸可能形成的所有4^80种序列的一小部分(约2×10^-41),但该方法还是非常成功的。
由此也可以推断出,即便是在序列空间进行稀疏采样,也大概率不会成为模型的障碍。
麻省理工学院的博士生Eeshit Dhaval Vaishnav是这项研究的第一作者。
他共发表过8篇顶刊论文。分别是「Nature」3篇,子刊「Nature Medicine」、「Nature Biotechnology」、「Nature Communications」各1篇,以及「Cell」1篇。
此前在印度理工学院获得计算机科学与工程和生物科学与生物工程双学位。
英属哥伦比亚大学生物医学工程学院助理教授Carl de Boer博士是共同一作。
他于2008获得滑铁卢大学计算机科学和生物信息学学士学位,并于2014年获得多伦多大学分子遗传学博士学位,此后便一直从事博士后研究。2020年进入英属哥伦比亚大学成为助理教授。
麻省理工学院的生物学教授Aviv Regev博士是这项研究的资深研究员。
她分别于1997年和2003年在特拉维夫大学获得硕士和博士学位,是麻省理工学院和哈佛大学Broad研究所的核心成员以及麻省理工学院生物系的教授,也是Genentech Research和Early Development的负责人。曾与Sarah Teichmann一起创立并领导了人类细胞图谱项目。
她的研究方向是生物网络、基因调控和进化。工作重点是剖析复杂的分子网络,以确定它们在面对遗传和环境变化时,以及在分化、进化和疾病期间是如何运作和演变的。
参考资料:
https://www.nature.com/articles/s41586-022-04506-6
https://news.mit.edu/2022/oracle-predicting-evolution-gene-regulation-0311
https://www.nature.com/articles/d41586-022-00384-0
![]()
![]()