清华曾坚阳研究组三维基因组研究成果入选2018年度“中国生物信息学十大进展”
清华大学交叉信息研究院曾坚阳研究组2018年发表于英国著名期刊《核酸研究》(Nucleic Acids Research)的三维基因组研究成果《基于流形学习的三维基因组结构重构》(Reconstructing spatial organizations of chromosomes through manifold learning),近日经《基因组蛋白质组与生物信息学报》,简称GPB(Genomics, Proteomics and Bioinformatics)评审,成功入选2018年度“中国生物信息学十大进展”。
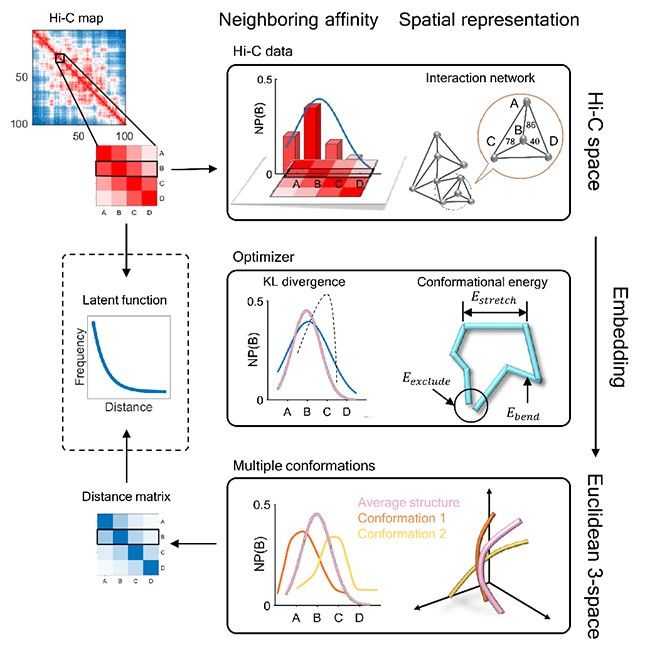
基因组在细胞核中以复杂的三维空间构象的形式存在,这种三维构象随着细胞状态和时间不断地发生动态变化,通常反映了基因组中不同区域发生相互作用的情况。三维基因组结构蕴含着非常丰富的信息,与细胞内绝大多数生物过程相关联,例如基因的转录、染色体的可接近性、DNA的甲基化以及表观遗传具有密切的关系,甚至有研究发现不同的基因组三维构象在疾病中作用也大不相同。因此,解析基因组的三维结构对研究基因调控有着重要意义。近年来,基于高通量测序技术(例如Hi-C)的三维基因组建模方法得到了广泛研究,然而大部分方法都依赖于对基因相互作用频率与染色体空间距离之间的关系进行假设。
三维基因组研究示意图
曾坚阳研究组首次基于流形学习直接将基因相互作用数据映射到相应的欧几里得空间,并结合生物分子能量规律,对三维基因组结构进行建模。该方法在模拟实验和真实数据上的基因组建模精度均显著超过了已有的方法,并且能够准确地还原出基因相互作用频率与染色体空间距离之间的关系函数。此外,该方法首次利用解出的三维结构进一步重构出了测序实验中丢失的基因相互作用数据,利用该方法重构出的基因相互作用数据大部分都能从已知的数据中获得证实。
《核酸研究》是国际核酸研究领域的权威专业期刊,2018年度影响因子为11.561;GPB是由中国科学院主管、中科院北京基因组所与中国遗传学会共同主办的英文版核心期刊。此项工作同以色列希伯来大学的Tommy Kaplan教授和美国伊利诺伊大学厄巴纳-香槟分校的Jian Peng教授合作完成。论文的第一作者为清华大学交叉信息研究院直博生朱广翔,论文通讯作者为交叉信息院曾坚阳副教授。研究工作得到国家自然科学基金和清华大学结构生物学高精尖创新中心的经费支持。
论文链接:
https://academic.oup.com/nar/article/46/8/e50/4835049

来源:清华新闻网
供稿:交叉信息研究院