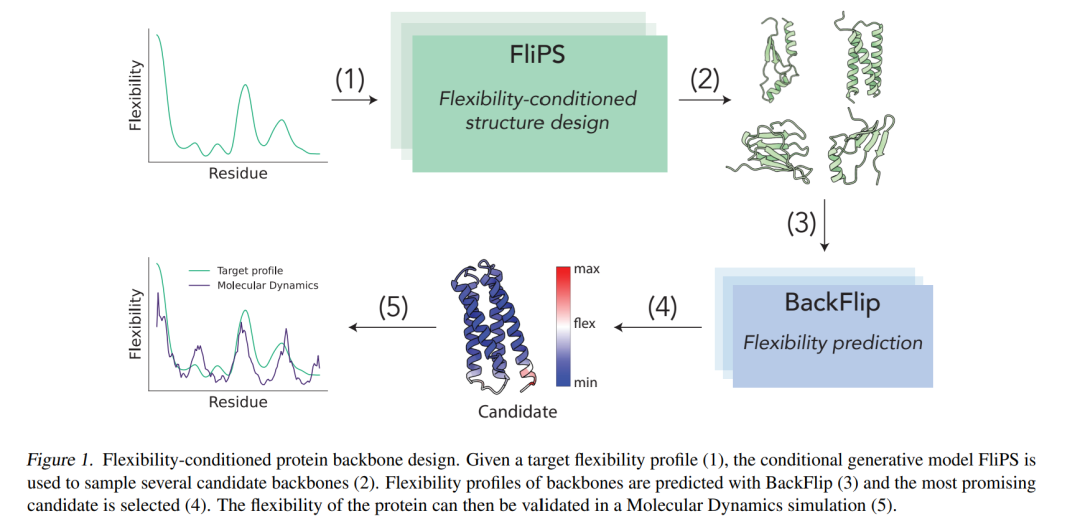
几何深度学习与生成建模的最新进展,使得具有多种目标属性的新型蛋白质设计成为可能。然而,当前的最新方法通常仅限于生成具有静态目标属性(如结构基序和对称性)的蛋白质。在本研究中,我们提出了一种新的框架,将**柔性(flexibility)**作为条件引入结构生成,从而迈出克服这一局限性的第一步。柔性在催化作用或分子识别等关键功能中至关重要。
我们首先提出 BackFlip,一种等变(equivariant)神经网络,用于从输入的主链结构预测逐残基(per-residue)的柔性。基于 BackFlip,我们进一步提出 FliPS,一种 SE(3)-等变的条件流匹配模型(conditional flow matching model),用于解决逆问题,即生成具有目标柔性分布的蛋白质主链。
在实验中,我们展示了 FliPS 能够生成符合目标柔性分布的全新且多样化的蛋白质主链,并通过分子动力学(Molecular Dynamics, MD)模拟验证了其有效性。FliPS 与 BackFlip 的代码可在以下地址获取: https://github.com/graeter-group/flips
成为VIP会员查看完整内容
相关内容
Arxiv
42+阅读 · 2023年4月19日
Arxiv
216+阅读 · 2023年4月7日
Arxiv
81+阅读 · 2023年4月4日
Arxiv
147+阅读 · 2023年3月29日