一天发表两篇论文,海普洛斯陈实富带您走近ctDNA液体活检的精准分析和临床应用
点击上方“公众号”可以订阅哦!
深圳市未来产业促进会会员单位:深圳市海普洛斯生物科技有限公司
2/2
一天发表两篇论文
海普洛斯陈实富带您走近ctDNA液体活检的精准分析和临床应用
引言
随着液体活检领域的发展,大多数行业人士已经对该技术不再陌生。作为液体活检的核心技术之一,ctDNA在癌症的个性化诊断、术后监测、癌症分型甚至早期筛查领域都有非常大的应用潜力。但是ctDNA的突变频率非常低,在数据分析中如何预防假阴性的发生呢?在临床应用中,ctDNA又如何应用于术后监测呢?恰好,今天海普洛斯有两篇相关的论文正式发表,海普洛斯CTO陈实富博士是两篇文章的共同通讯作者,让我们一起来听一下陈博士给您带来的解读。
2
Shifu Chen et al, MutScan: fast detection and visualization of target mutations by scanning FASTQ data. BMC Bioinformatics.
在很多基因检测应用,比如ctDNA基因检测应用中,我们都需要对一些已知位点进行高敏度检测,比如 EGFR p.L858R, BRAF p.V600E等位点。
然而,传统的NGS数据分析流程通常会有很多环节,而每一个环节都可能带来信息的损耗,这导致在最后一步进行Variant Calling的时候,对于一些超低频的突变可能会漏检。
为了解决ctDNA测序数据分析中的假阴性问题,陈博士开发了MutScan,该工具可以不经过常规的过滤、比对、校正、变异检出等环节,而直接从原始的FASTQ文件中检测目标变异。
基于优化的DNA检索算法,该软件可以比常规的NGS数据分析流程(例如 BWA + GATK)快20倍以上,而且敏感度非常高,哪怕一个变异的支持序列(Supporting Reads)只有一两条,也可以进行检测,在数据分析上排除了液体活检的假阴性问题。
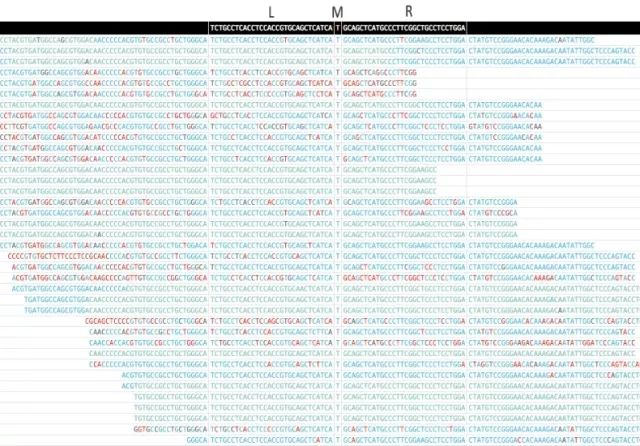
该软件可以对目标变异的原始序列进行可视化,并形成可交互性的HTML报告。在HTML结果中,可以查看每一个变异的所有支持序列,包括其中每一个碱基的质量值。下图展示了一个EGFR p.T790M的HTML可视化报告结果。
MutScan是一款开源软件,属于陈博士发起的开源项目组OpenGene中的一个重要项目,感兴趣的用户可以到其开源项目地址上进行下载使用:
https://github.com/OpenGene/MutScan
除了对变异点进行扫描检测并进行可视化之外,MutScan还可以应用于其他需要对目标序列进行扫描的领域。比如有的研究员就在使用MutScan来辅助CRISPR进行基因编辑的设计。
论文原文:
https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-018-2024-6
另外,看到这里的你,一定很关心第一篇是什么,点击以下链接,让大脑来一场文献风暴吧!
注:投稿请电邮至124239956@qq.com ,合作 或 加入未来产业促进会请加:www13923462501 微信号或者扫描下面二维码:
文章版权归原作者所有。如涉及作品版权问题,请与我们联系,我们将删除内容或协商版权问题!联系QQ:124239956