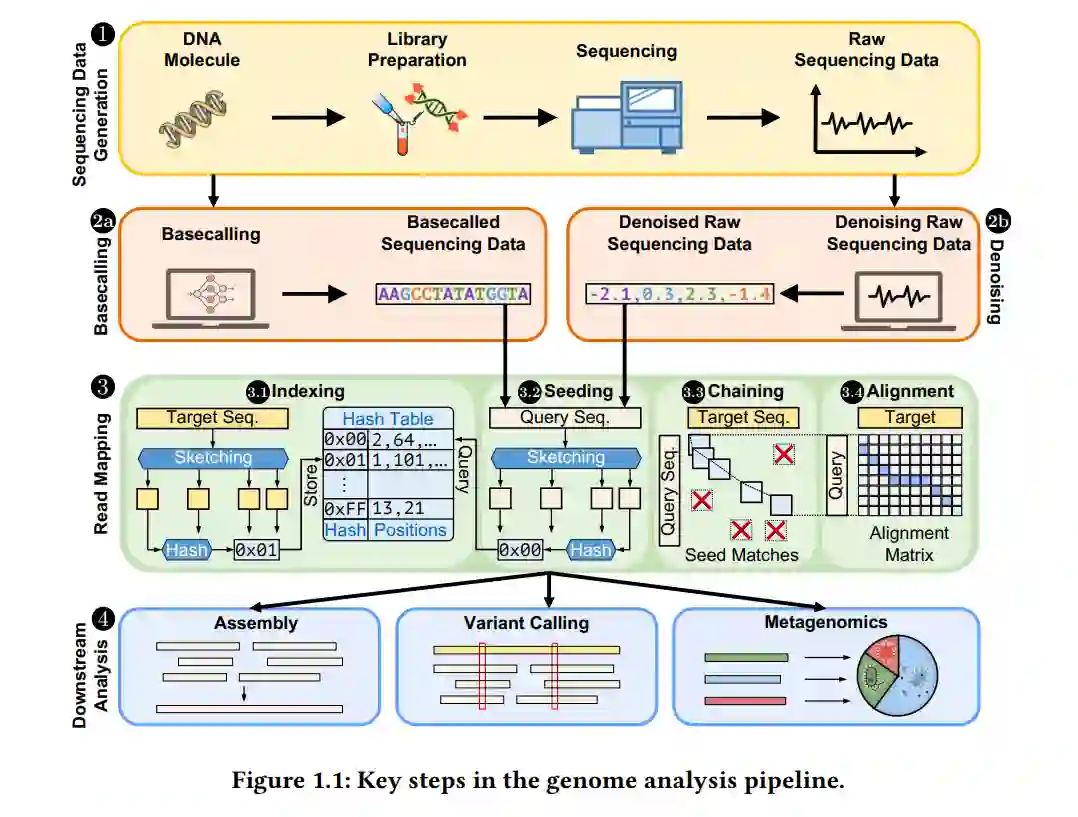
高通量测序(HTS)技术的出现彻底改变了基因组分析,使得大规模基因组的快速且经济高效的测序成为可能。尽管取得了这些进展,基因组数据的复杂性和数据量的不断增加仍然带来了与准确性、可扩展性和计算效率相关的重大挑战。这些挑战主要源于测序数据中各种形式的不必要和未处理的变异,统称为噪声。解决这些挑战需要深入理解基因组数据中不同类型的噪声,并开发技术以减轻噪声对基因组分析的影响。 在本论文中,我们旨在理解影响基因组分析流程的噪声类型,并通过开发新的计算技术来容忍或减少噪声,从而实现对不同类型测序数据(例如纳米孔测序的原始电信号)更快、更准确和可扩展的分析。 首先,我们介绍了BLEND,一种噪声容忍的哈希机制,能够通过单次哈希值查找快速识别完全匹配和高度相似但存在任意差异的序列。其次,为了实现可扩展且准确的噪声原始纳米孔信号分析,我们提出了RawHash,这是一种新颖的机制,通过提出首个基于哈希的原始纳米孔信号相似性搜索技术,有效减少原始纳米孔信号中的噪声,并实现准确的实时分析。第三,我们通过RawHash2扩展了RawHash的功能,这一改进机制:1)更好地理解原始纳米孔信号中的噪声以更有效地减少噪声;2)提高映射决策的鲁棒性。第四,我们通过引入Rawsamble探索了原始纳米孔信号分析的更广泛影响和新应用。Rawsamble是首个使用基于哈希搜索的原始信号全对全重叠机制,能够直接从原始信号构建从头组装,而无需进行碱基识别,这为原始纳米孔信号分析开辟了新的方向和应用。
本论文全面理解了不同类型基因组数据中的噪声如何影响基因组分析流程,并提供了减轻噪声影响的新颖解决方案。我们的研究结果表明,通过使用新的计算技术有效容忍和减少噪声,我们可以:1)显著提高基因组分析的性能、准确性和可扩展性;2)通过实现新的应用和方向扩展原始信号分析的范围。我们希望并相信,本论文中提出的方法和见解将有助于发明和开发更强大、更高效、功能更全面的基因组分析工具,特别是在原始信号分析领域。


